
枯草芽孢杆菌简介及应用 枯草芽孢杆菌简介 枯草芽孢杆菌(Bacillaceae)编号为Strain Number ACCC 11060,是芽孢杆菌属的一种。单个细胞0.7~0.8×2~3微米,着色均匀。无荚膜,周生鞭毛,能运动。革兰氏阳性菌,芽孢0.6~0.9×1.0~1.5微米,椭圆到柱状,位于菌体中央或稍偏,芽孢形成后菌体不膨大。菌落表面粗糙不透明,污白色或微黄色,在液体培养基中生长时,常形成皱醭,需氧菌。可利用蛋白质、多种糖及淀粉,分解色氨酸形成吲哚[1]。早在1835 年,Ehrenberg 所描述的“Vibrio subtilis”即是现在大家熟悉的“枯草芽孢杆菌”,它是由Cohn 于1872 年正式命名的,现作为芽孢杆菌科(Bacillaceae) 的模式菌株,芽孢杆菌是人类发现最早的细菌之一。 枯草芽孢杆菌应用 枯草杆菌(B acillus Subtilis) 是一种重要的α-淀粉酶生产菌。同时枯草芽孢杆菌作为一种安全、高效、多功能和极具开发潜力的微生物菌种已广泛应用于工业、农业、医药、卫生、食品、畜牧业、水产及科研诸领域。随着经济的发展、科研水平的提高,枯草芽孢杆菌与人们的日常生活将更为密切,它也必将作为一种十分重要的工业微生物菌种越来越引起人们的普遍关注和青睐。 ①枯草芽孢杆菌在工业酶生产中的应用 工业酶的生产是工业微生物发酵的重要组成部分。枯草芽孢杆菌是当今工业酶生应用最广泛的菌种之一,据不完全统计,枯草芽孢杆菌所产的酶占整个酶市场的50 %。由于其产酶量高、种类多、安全性好和环保等优点,在现代工业生产中被广泛用作生产菌种,其发酵生产的酶已在食品、饲料、洗涤、纺织、皮革、造纸和医药等领域均发挥着十分重要的作用。 ②枯草芽孢杆菌在生物防治领域中的应用 枯草芽孢杆菌作为植物病害生防细菌之一,具有较强的防病作用美国迄今已有4 株枯草芽孢杆菌生防菌株获得环保局商品化或有限商品化生产应用许可,如美国AgraQuest公司用枯草芽孢杆菌QST713 菌株开发出活菌制剂杀菌剂SerenadeTM,并于2000 年通过美国环保局(EPA) 的登记,用于防治多种作物的白粉病、霜露病、疫病、灰霉病等病害。我国利用枯草芽孢杆菌防治植物病害的应用研究也达到了世界先进水
全基因组重测序数据分析 1. 简介(Introduction) 通过高通量测序识别发现de novo的somatic和germ line 突变,结构变异-SNV,包括重排 突变(deletioin, duplication 以及copy number variation)以及SNP的座位;针对重排突变和SNP的功能性进行综合分析;我们将分析基因功能(包括miRNA),重组率(Recombination)情况,杂合性缺失(LOH)以及进化选择与mutation之间的关系;以及这些关系将怎样使 得在disease(cancer)genome中的mutation产生对应的易感机制和功能。我们将在基因组 学以及比较基因组学,群体遗传学综合层面上深入探索疾病基因组和癌症基因组。 实验设计与样本 (1)Case-Control 对照组设计; (2)家庭成员组设计:父母-子女组(4人、3人组或多人); 初级数据分析 1.数据量产出:总碱基数量、Total Mapping Reads、Uniquely Mapping Reads统计,测序深度分析。 2.一致性序列组装:与参考基因组序列(Reference genome sequence)的比对分析,利用贝叶斯统计模型检测出每个碱基位点的最大可能性基因型,并组装出该个体基因组的一致序列。3.SNP检测及在基因组中的分布:提取全基因组中所有多态性位点,结合质量值、测序深度、重复性等因素作进一步的过滤筛选,最终得到可信度高的SNP数据集。并根据参考基 因组信息对检测到的变异进行注释。 4.InDel检测及在基因组的分布: 在进行mapping的过程中,进行容gap的比对并检测可信的short InDel。在检测过程中,gap的长度为1~5个碱基。对于每个InDel的检测,至少需 要3个Paired-End序列的支持。 5.Structure Variation检测及在基因组中的分布: 能够检测到的结构变异类型主要有:插入、缺失、复制、倒位、易位等。根据测序个体序列与参考基因组序列比对分析结果,检测全基因组水平的结构变异并对检测到的变异进行注释。
全基因组关联分析(Genome-wide association study;GWAS)是应用基因组中 数以百万计的单核苷酸多态性(single nucleotide ploymorphism ,SNP)为分子 遗传标记,进行全基因组水平上的对照分析或相关性分析,通过比较发现影响复杂性状的基因变异的一种新策略。 随着基因组学研究以及基因芯片技术的发展,人们已通过GWAS方法发现并鉴定了大量与复杂性状相关联的遗传变异。近年来,这种方法在农业动物重要经济性状主效基因的筛查和鉴定中得到了应用。 全基因组关联方法首先在人类医学领域的研究中得到了极大的重视和应用,尤其是其在复杂疾病研究领域中的应用,使许多重要的复杂疾病的研究取得了突破性进展,因而,全基因组关联分析研究方法的设计原理得到重视。 人类的疾病分为单基因疾病和复杂性疾病。单基因疾病是指由于单个基因的突变导致的疾病,通过家系连锁分析的定位克隆方法,人们已发现了囊性纤维化、亨廷顿病等大量单基因疾病的致病基因,这些单基因的突变改变了相应的编码蛋白氨基酸序列或者产量,从而产生了符合孟德尔遗传方式的疾病表型。复杂性疾病是指由于遗传和环境因素的共同作用引起的疾病。目前已经鉴定出的与人类复杂性疾病相关联的SNP位点有439 个。全基因组关联分析技术的重大革新及其应用,极大地推动了基因组医学的发展。(2005年, Science 杂志首次报道了年龄相关性视网膜黄斑变性GWAS结果,在医学界和遗传学界引起了极大的轰动, 此后一系列GWAS陆续展开。2006 年, 波士顿大学医学院联合哈佛大学等多个研究机构报道了基于佛明翰心脏研究样本关于肥胖的GWAS结果(Herbert 等. 2006);2007 年, Saxena 等多个研究组联合报道了与2 型糖尿病( T2D ) 关联的多个位点, Samani 等则发表了冠心病GWAS结果( Samani 等. 2007); 2008 年, Barrett 等通过GWAS发现了30 个与克罗恩病( Crohns ' disrease) 相关的易感位点; 2009 年, W e is s 等通过GWAS发现了与具有高度遗传性的神经发育疾病——自闭症关联的染色体区域。我国学者则通过对12 000 多名汉族系统性红斑狼疮患者以及健康对照者的GWAS发现了5 个红斑狼疮易感基因, 并确定了4 个新的易感位点( Han 等. 2009) 。截至2009 年10 月, 已经陆续报道了关于人类身高、体重、 血压等主要性状, 以及视网膜黄斑、乳腺癌、前列腺癌、白血病、冠心病、肥胖症、糖尿病、精神分 裂症、风湿性关节炎等几十种威胁人类健康的常见疾病的GWAS结果, 累计发表了近万篇 论文, 确定了一系列疾病发病的致病基因、相关基因、易感区域和SNP变异。) 标记基因的选择: 1)Hap Map是展示人类常见遗传变异的一个图谱, 第1 阶段完成后提供了 4 个人类种族[ Yoruban ,Northern and Western European , and Asian ( Chinese and Japanese) ] 共269 个个体基因组, 超过100 万个SNP( 约1
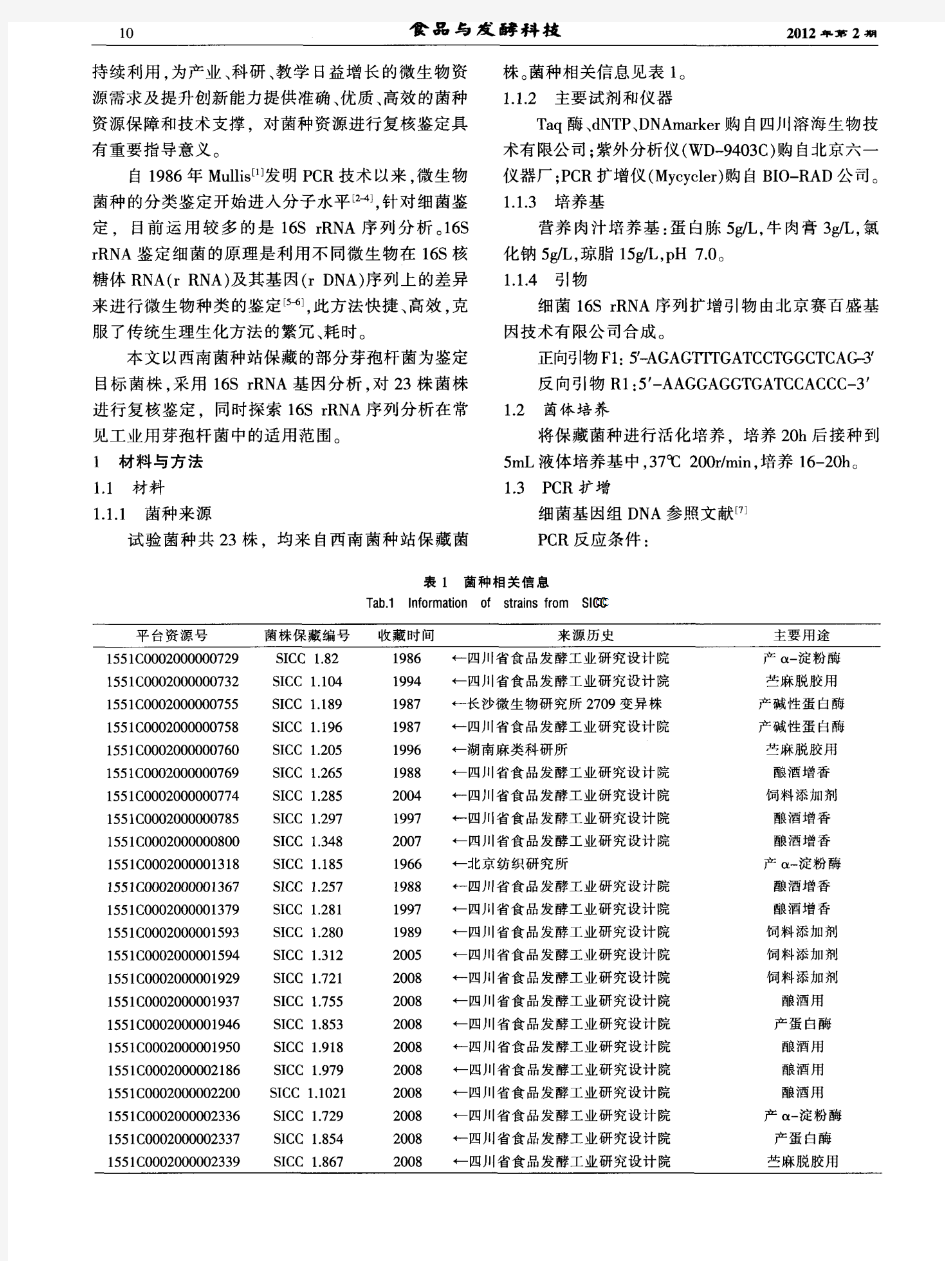
目录 第一章芽孢杆菌的简要介绍 (1) 第一节芽孢杆菌种类 (1) 第二节芽孢杆菌表达系统发展简史 (2) 第二章枯草芽孢杆菌的转化系统 (3) 第一种方法:电转化 (3) 第二种方法:Spizizen转化 (3) 第三种方法:原生质体法(Takashi) (4) 第四种方法:原生质体转化之二 (4) 第五种转化方法:质粒混合法(BGSC推荐) (5) 第三章芽孢杆菌表达系统发展简史 (6) 第一节芽孢杆菌表达系统的优点(相对于大肠杆菌) (7) 第二节芽孢杆菌的缺点 (7) 第三节助表达系统 (7) 第四节芽孢杆菌基因表达的主要特点 (7) 第四章枯草芽孢杆菌转录翻译系统 (8) 第一节:转录系统 (9) 第二节:翻译系统 (9) 第五章芽孢杆菌常用的宿主和载体 (10) 第六章芽孢杆菌表达系统应用实例 (11) 1 中国 (11) 2 日本 (12) 3 加拿大 (12) 第七章芽孢杆菌其他产品 (13) 第一节核苷类产品 (13) 第二节核黄素 (13) 第三节微生物制剂/益生菌 (13) 第八章结语 (14) 附录一. 芽孢杆菌的相关经典文章 (14) 附录二. 枯草芽孢杆菌相关数据库 (15) 致谢及参考文献 (15)
第一章芽孢杆菌的简要介绍 芽孢杆菌作为一个属,于1872年被首次提出,至今已有一百多年。目前人们对芽孢杆菌的研究几乎涉及到了革兰氏阳性可生孢细菌的各个领域。尤其是在感受态、芽孢形成及其调控、遗传操作、菌种改良、生物技术等领域进行了大量的工作。芽孢杆菌是一个泛泛的概念,而科学研究中应用最多的当属枯草芽孢杆菌,例如168菌株及其大量的衍生菌株。枯草杆菌的研究之所以领先于其他芽孢杆菌的种,主要是由于他的转化、转导方法较完善,以及大量的衍生菌株。 目前应用最多的芽孢杆菌属菌种有枯草芽孢杆菌、嗜碱芽孢杆菌、解淀粉芽孢杆菌、短芽孢杆菌、地衣芽孢杆菌、巨大芽孢杆菌、短小芽孢杆菌、球形芽孢杆菌、嗜热脂肪芽孢杆菌、苏云金芽孢杆菌和耐碱的芽孢杆菌以及病原菌炭疽芽孢杆菌等12种。 第一节芽孢杆菌种类 目前,芽孢杆菌属很多菌株的全基因组序列已经报道,截至2011年10月,在KEGG 上公布全基因组序列的芽孢杆菌属菌种有: 简称菌种名称测序时间测序链接 bsu Bacillus subtilis1997RefSeq bss Bacillus subtilis subsp. spizizenii W232010RefSeq bst Bacillus subtilis subsp. spizizenii TU-B-102011 RefSeq bsn Bacillus subtilis BSn52011RefSeq bha Bacillus halodurans2000RefSeq ban Bacillus anthracis Ames2003RefSeq bar Bacillus anthracis Ames 05812004RefSeq bat Bacillus anthracis Sterne2004 RefSeq bah Bacillus anthracis CDC 6842009 RefSeq bai Bacillus anthracis A02482009 RefSeq bal Bacillus cereus biovar anthracis CI2010RefSeq bce Bacillus cereus ATCC 145792003RefSeq bca Bacillus cereus ATCC 109872004RefSeq bcz Bacillus cereus ZK2004RefSeq bcr Bacillus cereus AH1872008 RefSeq bcb Bacillus cereus B42642008 RefSeq bcu Bacillus cereus AH8202009 RefSeq bcg Bacillus cereus G9******* RefSeq bcq Bacillus cereus Q12009RefSeq
功能基因的序列比对 <1>.切除载体和(或)引物 a.打开所有的原始引物序列于一个EditSeq的窗口中 b. export all as one c.保存 d.打开这个保存的文件,开始切除载体和引物 e.选择载体插入点两侧的序列(10-15个的样子)搜索注意:不存在正反向的问题,都是一个
方向,因为测序的时候是选择两个载体上的引物其中的一条来往后测序的! 切完之后另存为 f. 重新打开这个文件,开始切除引物 方法同切载体,但是要注意正反向的问题。比如mcrA基因,其引物为Forward: 5'-GGTGGTGTMGGATTCACACARTAYGCWACAGC-3' Reverse: 5'-TTCATTGCRTAGTTWGGRTAGTT-3'
先找Forward 5’端,此时只找到的部分序列。切去5’端。 然后再切这些切掉5’端序列的3’端的序列,此时其3’端序列应该是Reverse 的反向互补序列。 切去这个反向互补序列,这样一来这个些序列就已经被切去两端的引物了。 但此时还剩下另一部分未切除任何引物的序列,此时记下这些序列的编号,先切去Reverse 5’
端。 再用Forward 的反向互补序列切去3’端,这样剩下的序列也都被切除两端的引物了。 <2>将所有序列调整为同向序列: a. 选择前面记录编号的序列,将这些序列一个个都转换为其反向互补序列。这样一来所有的序列都成为同向序列了,即在DNA两条反向互补链的其中一条上的比较了。
b. 保存该文件 <3> 生成OTUs Google 搜索”Fastgroup II” 或https://www.doczj.com/doc/e118245168.html,/fg_tools.htm
如何查找基因序列?(转载) (2010-08-01 11:47:41) 如何查找基因序列? ——在Genbank中寻找目的基因的实例 ——献给受类似问题困扰的广大酷友,以及给我动力和信心发表原创帖的基因酷的朋友们。 酷友感言:网络的世界很精彩,网络的查询很无奈。为了我们的科学研究事业,为了我们能够顺利毕业,我们的广大酷友们在网络的海洋里遨游…遨游…咋就找不到彼岸呢?今天要设计这个基因的PCR引物,明天又要查那个基因的信息,那么大一张网,唉想起来就郁闷……鉴此,我们推出了利用Genbank查找基因序列的帖子,希望对大家有所帮助,并请大家多多指教!当然,如果您已经是此中高手,那就权当我是班门弄斧了,呵呵。 1. 根据文献 搞reasearch肯定要读文献的,如果你曾经在文献中看到过你感兴趣的基因,而且文中还提到了该基因在Genbank中的ID号,那就好办了,直接打开https://www.doczj.com/doc/e118245168.html,,在Search后的下拉框中选择Nucleotide,把Genbank ID号输入GO前面的文本框中,点“GO”,就可以找到他了。 举例说明,例如:在2003年JBC的文章(Conditional Knock-out of
Integrin-linked Kinase Demonstrates an Essential Role in Protein Kinase B/Akt Activation)中出现了“calreticulin (GenBank accession number gi 16151096)”,那么把“16151096”输入GO前面的文本框中,点“GO”,就可以找到该基因了(当然包括基因序列等相关信息)。 在出现了检索结果界面(下图)后,直接点击红箭头所指的 AY047586就可以看到基因的相关信息了...(呵呵,是不是有点太......easy 了) 这里需要指出一下,在显示基因的页面右侧有一个Link,点击后出现一个小菜单,里面是与该基因相关的链接,很有用的,值得一个一个地去看看,这里我就不多说了。点击 AY047586后出现的界面如下:如果你只想获得序列(例如去设计PCR引物的时候),那就可以选择FASTA,这样就得到了FASTA格式的序列文件,没有其他数字和格式的干扰。 (缩略图,点击图片链接看原图)这就是FASTA格式的序列: (缩略图,点击图片链接看原图)2. 根据已经获得的基因的相关信息进行查找(待续......) 鼓励一下吧,累坏了正如路漫漫所说,如果只是知道基因的名字,怎么查序列呢?还是举例说明,比如我想做的基因名称是人的VEGF基因,那么怎么在Genbank中找到它呢?还是一步一步来...打开https://www.doczj.com/doc/e118245168.html,/ 在search后面的下拉框中选择Gene,然后在中间的文本框中输入基
1 技术优势 全基因组测序(Whole Genome Sequencing,WGS)是利用高通量测序平台对人类不同个体或群体进行全基因组测序,并在个体或群体水平上进行生物信息分析。可全面挖掘DNA 水平的遗传变异,为筛选疾病的致病及易感基因,研究发病及遗传机制提供重要信息。 全基因组测序 平台优势 HiSeq X 测序平台 读长:PE150 通量:1.8T/run 测序周期:3 天 专为人全基因组测序准备、测序周期短、通量高
生物信息分析 技术路线 技术参数 样品要求 样本类型:DNA 样品 样本总量:≥1.0 μg DNA (提取自新鲜及冻存样本) ≥1.5 μg DNA (提取自FFPE 样本)样品浓度:≥ 20 ng/μl 测序平台及策略HiSeq X PE150 测序深度 肿瘤:癌组织(50X),癌旁组织/血液样本(30X)遗传病:30~50 X 项目周期37天
3 案例解析 该研究选取3个家系中6个患者和1个正常个体,首先使用基因芯片寻找纯合突变位点,然后对其中无亲缘关系的2例患者采用全基因组测序研究,在2例患者非编码区域均发现相同的变异,10号染色体PTF1A 末端发生一个点突变(chr10:23508437 A>G),且变异在患病人群和细胞试验中均得到了验证。研究解释了生长发育启动子隐性变异是罕见孟德尔遗传病的常见致病原因,同时说明许多疾病的致病突变也可能位于非编码区。 图1 检出的变异信息 智力障碍是影响新生儿心智发育的一类疾病。这项研究选取50个经过基因芯片和全外显子测序未确诊致病因子的trio 家系,全基因组测序检出84个de novo SNVs 和8个de novo CNVs,及一些结构变异(如VPS13B、STAG1、IQSEC2-TENM3),检出率为42%。揭示编码区的de novo SNVs 和de novo CNVs 是导致智力障碍的主要因素,全基因组测序可以作为可靠的遗传性检测应用工具。 案例一 单基因病研究——全基因组测序鉴定PTF1A末端增强子常染色体隐性突变导致胰腺 发育不全[1] 案例二 复杂疾病研究——全基因组测序解析智力障碍的主要致病因素[2] 图2 PTF1A 的家系图谱
Bioprocess 生物过程, 2018, 8(1), 1-10 Published Online March 2018 in Hans. https://www.doczj.com/doc/e118245168.html,/journal/bp https://https://www.doczj.com/doc/e118245168.html,/10.12677/bp.2018.81001 Phylogenetic Analysis of Rab3A Gene Zhanlong Qiao, Jingfen Yang, Zhiwei Wei, Tao Liu* College of Science, North China University of Science and Technology, Tangshan Hebei Received: Feb. 24th, 2018; accepted: Mar. 5th, 2018; published: Mar. 13th, 2018 Abstract Rab3A is the most abundant Rab protein in the animal’s brain. Rab proteins play an important role in vesicular trafficking by exocytosis and endocytosis [1]. Thus, the Rab3A gene plays an impor-tant role in the regulation of animal memory. The original nucleotide sequence was downloaded from the NCBI database. Collinearity analysis was carried out by using Blast + homology alignment and ColinearScan and MCscanX. Using MEGA7.0 software Maximum Likelihood method, Neigh-bor-joining method to construct phylogenetic tree, due to the homology and collinearity analysis and comparison from the original data, a consistent phylogenetic tree was obtained. The phyloge-netic tree of Rab3A gene is consistent with the species developmental tree. This shows that the evolution of the gene occurs along with the divergence of species, which is a relatively ancient gene. Keywords Rab3A, System Evolution and Development, Genes, Animals 记忆基因(Rab3A)的系统发育分析 乔战龙,杨静芬,魏志伟,刘涛* 华北理工大学理学院,河北唐山 收稿日期:2018年2月24日;录用日期:2018年3月5日;发布日期:2018年3月13日 摘要 Rab3A是动物大脑中丰度最高的Rab蛋白,而Rab蛋白通过胞吞和胞吐的方式在囊泡运输中起重要作用 [1],由此可见,Rab3A基因对于调控动物的记忆有很重要的作用。从NCBI数据库下载原始核苷酸序列, 运用Blast+同源性比对、共线性分析软件ColinearScan、MCscanX进行共线性分析,用MEGA7.0软件中*通讯作者。
全球首次完成杨树全基因组测序 由美国能源部启动并实施的杨树全基因组测序计划已圆满完成,并于2004年9月21日对公众开放了全序列数据库。南京林业大学科研人员尹佟明副教授参与了此项研究。杨树基因组的新闻发布及庆祝会定于12月6日在美国加州举行。该项研究可望使杨树这一重要树种的品种改良时间大大缩短,用区区几十年跨越千年关。 研究的完成,使杨树成为继拟南芥和水稻之后,第三个测定全序列的植物,并且是第一个测定全基因组序列的多年生木本植物。杨树因此被广泛接受为研究多年生植物基因组的模式物种,这使该项工作具有重大的科学意义。杨树同时又是一种重要的工业用材树种,杨树全基因组计划实施,将为生物能源的开发提供知识贮备,具有重要的实际应用价值。目前,杨树的改良还处在一种半野生的初级改良阶段,在基因组研究的基础上,通过群体和数量遗传学的手段在杨树属不同树种间开发有用等位基因,并通过遗传工程的手段进行基因重组,可望在几十年的时间里完成一般作物几千年的改良历程。 杨树全基因组全序列用“鸟枪法测定”,序列库中共含有7,649,993个序列片段,去除叶绿体基因组的污染,测得的序列大约为8×基因组长度。目前对序列拼接的组装已完成了483Mb,占杨树基因组物理全长的90%以上,基本上覆盖了杨树基因组常染色体的大部分。基于基因芯片和单核苷酸多态性检测技术,对小的序列拼接及序列间隙的填充工作正在进行中,预期这部分工作将于明年完成。南京林业大学尹佟明副教授自2001年以来一直参与此项研究,对杨树基因组的注释工作将于今年12月初完成。 国际杨树基因组计划协作组的总负责人杰瑞先生认为,从世界范围来看,杨树在中国的林业生产中占有的比重是最大的,因此在杨树基因组信息的应用方面,中国在未来的研究中可能会居于世界前列。杨树全基因组计划的完成对我国从事林业及生物技术的科学家而言,提供了前所未有的机遇和挑战。 Science 15 September 2006: Vol. 313. no. 5793, pp. 1596 - 1604 DOI: 10.1126/science.1128691 RESEARCH ARTICLES The Genome of Black Cottonwood, Populus trichocarpa (Torr. & Gray) G. A. Tuskan,1,3* S. DiFazio,1,4S. Jansson,5J. Bohlmann,6I. Grigoriev,9U. Hellsten,9N. Putnam,9S. Ralph,6S. Rombauts,10 A. Salamov,9J. Schein,11L. Sterck,10 A. Aerts,9 R. R. Bhalerao,5 R. P. Bhalerao,12 D. Blaudez,13 W. Boerjan,10 A. Brun,13 A. Brunner,14 V. Busov,15 M. Campbell,16 J. Carlson,17 M. Chalot,13 J. Chapman,9 G.-L. Chen,2 D. Cooper,6 P. M. Coutinho,19 J. Couturier,13 S. Covert,20 Q. Cronk,7 R. Cunningham,1 J. Davis,22 S. Degroeve,10 A. Déjardin,23 C. dePamphilis,18 J. Detter,9 B. Dirks,24 I. Dubchak,9,25 S. Duplessis,13 J. Ehlting,7 B. Ellis,6 K. Gendler,26 D. Goodstein,9 M. Gribskov,27 J. Grimwood,28 A. Groover,29 L. Gunter,1 B. Hamberger,7 B. Heinze,30 Y. Helariutta,12,31,33 B. Henrissat,19 D. Holligan,21 R. Holt,11 W. Huang,9 N. Islam-Faridi,34 S. Jones,11 M. Jones-Rhoades,35 R. Jorgensen,26 C. Joshi,15 J. Kangasj?rvi,32 J. Karlsson,5 C. Kelleher,6 R. Kirkpatrick,11 M. Kirst,22 A.
枯草芽孢杆菌的介绍 完成者:河岸hkfced(https://www.doczj.com/doc/e118245168.html,/hkfced)完成时间:2012-3-23 版本:第二版本
目录 第一章芽孢杆菌的简要介绍 (1) 第一节芽孢杆菌种类 (1) 第二节芽孢杆菌的表达系统发展简史 (2) 第二章枯草芽孢杆菌的转化系统 (3) 第一节:常见转化方法 (3) 1 化学转化法 (3) 2 电转化 (3) 3 原生质体转化 (3) 4 碱金属离子转化 (4) 5 质粒的其它转移方式 (4) 第二节:标准操作 (4) 第一种方法:电转化 (4) 第二种方法:Spizizen转化 (5) 第三种方法:原生质体法(Takashi) (5) 第四种方法:原生质体转化之二 (6) 第五种转化方法:质粒混合法(BGSC推荐) (7) 第三章芽孢杆菌的表达系统 (8) 第一节芽孢杆菌表达系统的优点(相对于大肠杆菌) (8) 第二节芽孢杆菌的缺点 (9) 第三节助表达系统 (9) 第四节芽孢杆菌基因表达的主要特点 (9) 第四章枯草芽孢杆菌的转录翻译系统 (9) 第一节:转录系统 (10) 第二节:翻译系统 (11) 第五章芽孢杆菌常用的宿主和载体 (12) 第六章芽孢杆菌应用实例 (13) 1 中国 (13) 2 日本 (13) 3 加拿大 (14) 第七章芽孢杆菌的产品 (14) 第一节核苷类产品 (14) 第二节核黄素 (14) 第三节微生物制剂/益生菌 (15) 第四节工业酶制剂 (15) 第八章结语 (15) 附录一. 芽孢杆菌的相关经典文章 (16) 附录二. 枯草芽孢杆菌相关数据库 (16) 致谢及参考文献 (17)
已完成植物基因组测序情况(更新至2014年11月) 中文名拉丁名发表时间刊物科、属基因组大小拟南芥Arabidopsis thaliana 2000.12 Nature 十字花科、鼠耳芥属125M 水稻Oryza sativa. ssp. indica 2002.04 Science 禾本科、稻属466M 水稻Oryza sativa. ssp. japonica 2002.04 Science 禾本科、稻属466M 杨树Populus trichocarpa 2006.09 Science 杨柳科、杨属480M 葡萄Vitis vinifera 2007.09 Nature 葡萄科、葡萄属490M 衣藻Chlamydomonas reinhardtii 2007.01 Science 衣藻科、衣藻属130 M 小立碗藓Physcomitrella pattens 2008.01 Science 葫芦藓科、小立碗藓属480M 番木瓜Carica papaya 2008.04 Nature 番木瓜科、番木瓜属370M 百脉根Lotus japonicus 2008.05 DNA Res. 豆科472 Mb 三角褐指藻Phaeodactylum tricornutum 2008.11 Nature 褐指藻属27.4M 高粱Sorghum bicolor 2009.01 Nature 禾本科、高粱属730M 玉米Zea mays ssp. mays 2009.11 Science 禾本科、玉米属2300M 黄瓜Cucumis sativus 2009.11 Nature Genetics 葫芦科、黄瓜属350M 大豆Glycine max 2010.01 Nature 豆科、大豆属1100M 二穗短柄草Brachypodium distachyon 2010.02 Nature 禾本科、短柄草属260M 褐藻Ectocarpus 2010.06 Nature 水云属196M 团藻Volvox carteri 2010.07 Science 团藻属138M 蓖麻Ricinus communis 2010.08 Nature Biotechnology 大戟科、蓖麻属350M 小球藻Chlorella variabilis 2010.09 Plant Cell 小球藻科46M 苹果Malus × domestica 2010.09 Nature Genetics 蔷薇科、苹果属742M 森林草莓Fragaria vesca 2010.12 Nature Genetics 蔷薇科、草莓属240M 可可树Theobroma cacao 2010.12 Nature Genetics 梧桐科、可可属430-Mb 野生大豆Glycine soja 2010.12 PNAS 豆科、大豆属915.4 Mb 褐潮藻类Aureococcus anophagefferens 2011.02 PNAS 57M 麻风树Jatropha curcas 2010.12 DNA Res. 大戟科、麻风树属410M 卷柏Selaginella moellendorffii 2011.05 Science 卷柏属212M 枣椰树Phoenix dactylifera 2011.05 Nature biotechnology 棕榈科685M 琴叶拟南 芥 Arabidopsis lyrata 2011.05 Nature Genetics 十字花科、鼠耳芥属206.7 Mb 马铃薯Solanum tuberosum 2011.07 Nature 茄目、茄科、茄属844M 条叶蓝芥Thellugiella parvula 2011.08 Nature Genetics 盐芥属140M
全基因组从头测序(de novo测序) https://www.doczj.com/doc/e118245168.html,/view/351686f19e3143323968936a.html 从头测序即de novo 测序,不需要任何参考序列资料即可对某个物种进行测序,用生物信息学分析方法进行拼接、组装,从而获得该物种的基因组序列图谱。利用全基因组从头测序技术,可以获得动物、植物、细菌、真菌的全基因组序列,从而推进该物种的研究。一个物种基因组序列图谱的完成,意味着这个物种学科和产业的新开端!这也将带动这个物种下游一系列研究的开展。全基因组序列图谱完成后,可以构建该物种的基因组数据库,为该物种的后基因组学研究搭建一个高效的平台;为后续的基因挖掘、功能验证提供DNA序列信息。华大科技利用新一代高通量测序技术,可以高效、低成本地完成所有物种的基因组序列图谱。包括研究内容、案例、技术流程、技术参数等,摘自深圳华大科技网站 https://www.doczj.com/doc/e118245168.html,/service-solutions/ngs/genomics/de-novo-sequencing/ 技术优势: 高通量测序:效率高,成本低;高深度测序:准确率高;全球领先的基因组组装软件:采用华大基因研究院自主研发的SOAPdenovo软件;经验丰富:华大科技已经成功完成上百个物种的全基因组从头测序。 研究内容: 基因组组装■K-mer分析以及基因组大小估计;■基因组杂合模拟(出现杂合时使用); ■初步组装;■GC-Depth分布分析;■测序深 度分析。基因组注释■Repeat注释; ■基因预测;■基因功能注释;■ ncRNA 注释。动植物进化分析■基因家族鉴定(动物TreeFam;植物OrthoMCL);■物种系统发育树构建; ■物种分歧时间估算(需要标定时间信息);■基因组共线性分析; ■全基因组复制分析(动物WGAC;植物WGD)。微生物高级分析 ■基因组圈图;■共线性分析;■基因家族分析; ■CRISPR预测;■基因岛预测(毒力岛); ■前噬菌体预测;■分泌蛋白预测。 熊猫基因组图谱Nature. 2010.463:311-317. 案例描述 大熊猫有21对染色体,基因组大小2.4 Gb,重复序列含量36%,基因2万多个。熊猫基因组图谱是世界上第一个完全采用新一代测序技术完成的基因组图谱,样品取自北京奥运会吉祥物大熊猫“晶晶”。部分研究成果测序分析结果表明,大熊猫不喜欢吃肉主要是因为T1R1基因失活,无法感觉到肉的鲜味。大熊猫基因组仍然具备很高的杂合率,从而推断具有较高的遗传多态性,不会濒于灭绝。研究人员全面掌握了大熊猫的基因资源,对其在分子水平上的保护具有重要意义。 黄瓜基因组图谱黄三文, 李瑞强, 王俊等. Nature Genetics. 2009. 案例描述国际黄瓜基因组计划是由中国农业科学院蔬菜花卉研究所于2007年初发起并组织,并由深圳华大基因研究院承担基因组测序和组装等技术工作。部分研究成果黄瓜基因组是世界上第一个蔬菜作物的基因组图谱。该项目首次将传
目 录 声明: (1) 大肠杆菌感受态细胞的制备 (2) 一、CaCl2感受态细胞的制备 (3) 二、电转感受态细胞的制备 (4) 枯草芽孢杆菌感受态细胞的制备及转化 (14) 农杆菌感受态细胞的制备及转化 (17) 酵母感受态细胞的制备及转化 (18) 关于载体连接的讨论 (23)
声明: 本文所涉及的技术要点主要来源于相关文献、网络和书籍,此外,一些操作中的技巧及经验等来自于本人及同学、朋友的总结,仅供大家参考,同时也请下载者不要传播尤其是以商业为目的的复制、传播等等;如有转载、传播请注明来源于“螺旋网”。若本文侵犯了您的版权或有任何不妥,请Email:lofeel@https://www.doczj.com/doc/e118245168.html, or helixnet@https://www.doczj.com/doc/e118245168.html,告知。 此外,由于学识、经验有限,本文难免会存在一些错误或缺陷,敬请不吝赐教,联系方式:lofeel@https://www.doczj.com/doc/e118245168.html, or helixnet@https://www.doczj.com/doc/e118245168.html,;同时因存在环境、试剂、仪器以及人为等原因,不能保证实验100%的成功,仅供参考。
大肠杆菌感受态细胞的制备 体外连接的DNA重组分子导入合适的受体细胞才能大量增殖。野生型E.coli 并不容易转化,这是由于细菌产生一种酶能迅速降解进入的外源DNA。为了提高受体菌摄取外源DNA的能力,提高转化效率以获得更多的转化子,人们摸索出不同的方法处理细菌,经过多年的努力,科学家们发现了可以增加细胞吸收外源DNA 效率的方法,那就是用化学方法处理细胞,使其改变膜对DNA的通透性。这种经过理化方法诱导细胞,使其处于最适摄取和容纳外来DNA的生理状态,该细胞就称为感受态细胞,即细胞处于能摄入核酸分子时的生理状态。这种方法已经成为基因工程的常规技术,它对于我们利用体外DNA重组技术来了解真核和原核生物的基因功能特别重要。细菌的转化有两种类型:一种是自然转化(natural transformation),在自然转化中细菌可以自由地吸收DNA,通过它来进行遗传转化;另一种是工程转化(engineered transformation),在这种转化中,细菌发生改变使得它们能摄入并转化外源DNA。枯草杆菌中的转化属于自然转化,E.coli转化就属于工程转化。 对于E.coli,目前主要采用电转化法和CaCl2法将外源DNA导入受体细胞中,并需要相应地制备电转化感受态细胞和CaCl2感受态细胞。其中,电转化法是利用瞬间高压在细胞上打孔,因而需用冰冷的超纯水多次洗涤处于对数生长前期的细胞,以使细胞悬浮液中应含有尽量少的导电离子。其转化效率为109~1010转化子/μg DNA。而对于热激法,是利用冰冷的CaCl2处理对数生长期的细胞,可以诱导其产生短暂的“感受态”,易于摄取外源DNA。转化效率为106~107转化子/μg DNA。
学院:______ 班级:_______ 学号:_________ 姓名:__________ 成绩:______ 实验五基因结构预测分析 目的: 1、熟悉并掌握从基因组核酸序列中发现基因的方法。 内容: 1、用NCBI的ORF Finder分析原核生物核酸序列或真核生物的cDNA序列中的开放阅读框; 2、使用GENSCAN在线软件预测真核生物基因; 3、使用POL YAH在线预测转录终止信号; 4、使用PromoterScan在线预测启动子区域。 操作及问题: 随着测序技术的不断发展,越来越多的模式生物启动了全基因组测序计划,完成全基因组测序的物种也越来越多,使得基因结构和功能的预测成为可能。同时,通过基因组文库筛选也可得到目的基因所在克隆。获得克隆序列后,同样也需要对目的基因做结构预测以便指导后续功能研究。本实验介绍几种常用的基因预测分析工具,预测核酸序列的开放阅读框、转录终止信号、启动子、CpG岛等信息。 一、开放阅读框(open reading frame,ORF)的识别 ORF是指从核酸序列上5’端翻译起始密码子到终止密码子的蛋白质编码序列。原核生物与真核生物的基因结构存在很大不同,真核生物的ORF除外显子(平均150bp)外,还含有内含子,因此真核生物基因的预测远比原核生物复杂。 (一)利用NCBI ORF Finder预测原核生物核酸序列或真核生物的cDNA序列中的开放阅读框。https://www.doczj.com/doc/e118245168.html,/gorf/gorf.html 1、在NCBI上查找AC 号为AE008569 的核酸记录。(见实验五中的AE008569.mht) 问题1:这个序列的名称? 问题2:这个序列来源物种所属的生物学大分类?
世界第一个黄种人全基因组序列图即将完 成 世界上第一个黄种人全基因组序列图即将由我国科学家绘制完成,这项工作正在位于深圳市盐田区的深圳华大基因研究院内紧锣密鼓地进行着。 该研究计划被命名为“炎黄一号”。计划发言人叶佳说,以黄种人的基因组图谱为研究目标,将为黄种人的基因研究和疾病治疗提供更准确和更有针对性的“基因标准图库”,“好比为你的基因做了一张参考CT”。 据悉,该研究由深圳华大基因研究院、生物信息系统国家工程中心及中国科学院北京基因组研究所的科学家共同承担,这一合作团队是曾经参与了“人类基因组计划”1%任务的主要成员。计划一旦成功,意味着中国将实现人类基因组序列图绘制工作从1%到100%的成功跨越。 目前全世界共发现2000个与疾病有关的人类基因,其中有1500个已在美国用于临床诊断。叶佳说,我国经政府批准用于临床诊断的基因种类仅几十个,不到美国的3%.由于黄种人突变位点与白种人不尽相同,不能完全照搬国外的诊断标准。因此,“开展黄种人基因与疾病关系的基础和应用研究显得意义非凡,但前提是,黄种人的基因组要有一个标准序列图,深圳开展的工作不一定就能定为标准图,但一定
是黄种人的第一个”。 相关技术的进步,尤其是新测序仪器的问世,大大加快了这一计划的速度。由六国合作的“国际人类基因组计划”,耗资数亿美元,花了几年时间才完成了任务;而如今利用新技术可在几个月时间内,花几千万人民币就能完成。“随着技术上的不断突破,有望在年内降至万元水平。”叶佳说。 “将来每个人都可能拥有自己的基因组图谱,就像拥有自己的CT图一样,”叶佳说,“这意味着实现个体化诊断、个体化治疗的梦想越来越近。” “炎黄一号”计划同时也是我国科学家与英国桑格基因组研究院合作进行的千人个体基因组多态性研究的一部分。这一计划的主要内容是,以新一代测序和高性能计算机技术为支撑,通过对白、黑、黄三人种进行大样本的全基因组测序和序列比较,探索人类基因组在不同人群中的多态性分布和变化规律。 科学家认为,个人测序与之前的人类基因组计划有所不同。之前的测序没有在每个染色体的两个副本之间、甚至是不同捐赠者的DNA之间作出区分,从而混淆了等位基因。 今年以来已经宣布完成个人基因组图谱的有两人。一是今年六月美国贝勒医学院的基因组中心,宣布完成了诺贝尔奖获得者、“DNA之父”詹姆斯·沃森的个人基因组序列图;另一个是今年9月刚刚在美国学术杂志上发表的,以传统测
实验三蛋白序列比对到基因组(GeneWise and exonerate)实验目的 1)了解基因结构,acceptor, sponsor 等概念 2)理解将蛋白序列比对到基因组的应用 3)掌握利用GeneWise 将蛋白序列定位到基因组上并得到基因结构 实验数据及软件 ftp://172.28.137.55/pub/lab_materia/biosoft/lab03/ 1、Genewise 简介 Genewise 是EBI 的Ewan Birney