基因表达谱分析技术
- 格式:pdf
- 大小:128.28 KB
- 文档页数:3

水稻生长中不同基因表达谱的分析研究
随着基因测序技术的不断发展,人们越来越能深入了解生命的本质,并运用这
些知识来改良农作物和提高粮食产量。
水稻是全世界重要的粮食作物之一,为了了解其生长发育过程中不同基因的表达情况,进行基因表达谱分析是非常有必要的。
一、水稻基因组和RNA测序技术的发展
水稻的基因组结构非常复杂,包含大量的基因序列和非编码RNA序列。
在过
去几年,随着高通量RNA测序技术的发展,人们可以更快速地测定水稻RNA的
序列,从而了解水稻基因表达谱,为后续的研究奠定基础。
二、水稻生长发育中的基因表达谱分析
水稻生长发育的不同阶段基因表达谱各异。
例如,水稻种子萌发时期的基因表
达谱主要集中在细胞分裂和细胞壁溶解相关的基因上。
而在幼苗期,基因表达谱则主要包括蛋白质合成和光合作用过程中的基因。
当水稻成熟时,基因表达谱则聚焦在颖果发育和种子贮藏蛋白基因的表达上。
三、水稻基因表达谱分析的意义
对水稻基因表达谱的了解,可以帮助人们更好地分析水稻的生长发育过程,进
而找到优化生产的途径。
例如,针对不同发育阶段的基因表达谱中重点关注的基因,人们可以更加精细地调整施肥、排水和抗病等方面的工作,以提高水稻的产量和质量。
解析水稻基因表达谱还可以帮助人们更深入地了解水稻在不同情况下的表现,并为后续的基因改良和新品种研发提供依据。
总之,水稻基因表达谱分析是一项非常重要的研究工作。
通过对水稻基因组的
深入了解,人们能够更好地探究水稻生长发育的本质,更精准地将水稻种植工作做到极致,进而提高粮食产量、改善农民生活水平。


基因共表达分析基因共表达分析是分子生物学中一项重要的技术,它能够从一系列基因的表达水平中,提取出其表达谱、异选、依赖性和联系的相关信息,从而发现生物体的基因网络结构、复杂疾病的分子机制等。
基因共表达分析是一项重要的研究发现和研究分析方法,它可以帮助研究者更好地理解生物体内复杂的生物功能,更好地推动科学研究和应用。
基因共表达分析,是一种多维数据分析技术,主要用于获取关于基因表达的具体信息。
该方法基于基因芯片技术,可以研究大量基因的表达水平,形成全基因组表达谱。
这种技术不仅能够检测基因的表达和活性,而且还可以检测基因的转录和调控,从而研究细胞内复杂的生物机制。
基因共表达分析的步骤一般可分为环境采样、数据准备、统计分析和基因组学等几个步骤。
环境采样是分析的基础,基因组学则是研究的核心,其中,对基因表达谱的统计分析是基因共表达分析的核心步骤。
其主要有两种:一种是运用基于配对聚集的假设检验方法,估计所研究的基因的两两之间的相关性;另一种是使用分类器方法,建立基因表达谱的分类模型,从而探索不同类别基因表达的差异和联系。
基因共表达分析作为一项重要的研究技术,已经在生物学、医学等领域得到了广泛应用。
它可以帮助我们更深刻地理解生物体内复杂的基因网络结构,为认识疾病发生和发展提供有益的线索,帮助诊断和治疗生物学上复杂的疾病,促进药物发现,并改善生物资源的利用,同时还可以有效地提高生物全基因组表达谱的准确性和可重复性研究。
近年来,基因共表达分析技术发展迅速,广泛应用于多学科领域。
一方面,随着软件、算法技术的不断发展,提高了共表达分析软件的数量和质量,提高了其准确性;另一方面,深入的生物学研究也为基因共表达分析技术的发展提供了前沿的突破口。
今后,基因共表达分析技术将继续发展,在各个领域的研究和应用中发挥更大的作用。
为了更好地发挥基因共表达分析技术的功能,需要不断完善表达数据的采集、处理、分析流程,提高表达数据的准确性和可重复性,不断开发新技术,探索新理论,进一步完善分析流程,加快基因共表达分析分析的应用速度。

检测基因表达变化的方法基因表达变化是指基因在特定条件下转录和翻译水平的变化。
检测基因表达变化的方法有很多种,以下是几种常用的方法:1. 转录组测序(RNA-seq)转录组测序是一种基于高通量测序技术的方法,可以检测基因在不同条件下的转录水平。
该方法首先从细胞中提取总RNA,然后通过建库、测序和分析得到每个基因的转录本序列。
通过比较不同条件下的转录本序列,可以发现基因表达的变化。
RNA-seq具有高灵敏度、高分辨率和高通量等优点,适用于研究基因表达的复杂性和动态性。
2. 定量反转录聚合酶链反应(qRT-PCR)qRT-PCR是一种基于PCR技术的方法,可以检测特定基因的表达水平。
该方法首先从细胞中提取总RNA,然后通过反转录得到cDNA,再通过PCR扩增得到目的片段。
通过比较不同条件下的目的片段拷贝数,可以发现基因表达的变化。
qRT-PCR具有高灵敏度、高特异性和可重复性好等优点,适用于验证RNA-seq等高通量测序方法的结果。
3. 微阵列分析微阵列分析是一种基于芯片技术的方法,可以同时检测多个基因的表达水平。
该方法将已知序列的探针集成在芯片上,然后将待测的cDNA或RNA与探针进行杂交。
通过检测杂交信号的强度,可以发现基因表达的变化。
微阵列分析具有高通量、高效率和高灵敏度等优点,适用于大规模的基因表达谱研究。
4. 原位杂交原位杂交是一种将探针与组织切片上的目标基因进行杂交的方法,可以检测目标基因在组织中的表达位置和表达水平。
该方法将探针与组织切片上的目标基因进行杂交,然后通过荧光或免疫组化等方法显色标记杂交信号。
通过观察杂交信号的数量和分布,可以发现基因表达的变化。
原位杂交具有高特异性、高灵敏度和定位准确等优点,适用于研究基因表达的组织特异性。
5. 免疫组织化学免疫组织化学是一种利用抗体与目标蛋白进行特异性结合的方法,可以检测目标蛋白在组织中的表达位置和表达水平。
该方法将抗体与目标蛋白进行特异性结合,然后通过显色标记抗体结合的位置。

基因表达水平检测方法基因表达水平检测方法是解决生物学中一系列实验问题的重要手段之一。
从基因转录到翻译,功能蛋白的表达需要多个步骤的参与,因此需要详细检测各个节点的表达水平才能全面理解生物系统的工作原理。
本文将介绍10种不同的基因表达水平检测方法,并详细讨论其优缺点及应用范围。
1. 实时荧光定量PCR(qPCR)实时荧光定量PCR(qPCR)是测量DNA片段数量的常用方法之一,可用于定量分析RNA 和DNA的含量及检测异质核糖体。
该方法利用荧光标记的探针结合特定反应体系,通过放大和检测PCR产物的荧光信号来定量目标序列的数量。
相较于传统定量PCR方法,qPCR具有高灵敏度、高特异性和高重现性等优点,可以为基因表达量的精确定量提供可靠的实验数据。
2. RNA测序(RNA-seq)RNA测序(RNA-seq)是一种全转录组测序技术,可以检测不同组织、细胞或条件下mRNA 的表达水平。
该技术通过将RNA逐个转录成cDNA,然后对cDNA进行二代测序,并通过比对与基因组或转录组的比对,确定基因在不同组织或条件下的表达情况,并可以鉴定新的基因或异构体。
RNA-seq可以检测出非编码RNA、剪接异构体等多种信息,成为研究基因抑制、基因启动等事件的有力工具。
3. 微阵列技术微阵列技术是一种古老的基因表达测量方法,可用于同步检测数千个基因。
该技术利用特殊制备的阵列,识别和定量检测小分子或生物大分子(如基因或蛋白质)相互作用的过程。
与RNA-seq相比,微阵列技术成本相对较低,但检测范围较小,并且需要预先设计探针和矩阵。
微阵列技术也可以检测mRNA的异构体、SNP等信息,对于高通量、大规模分析有一定的优势。
4. 蛋白质质谱分析蛋白质质谱分析技术(protein mass spectrometry)可用于评估蛋白质在组织、细胞或条件下的表达量和修饰情况。
该方法将蛋白质分离和检测结合到一起,先通过酶解纯化和分离蛋白质产物,然后利用质谱技术进行检测。

表达谱基因比对表达谱和基因比对是两个在生物学和基因研究中非常重要的概念。
表达谱指的是特定生物个体在特定环境下所有基因的转录水平,即基因的表达情况。
而基因比对则是基于不同个体的基因序列相互比较,寻找它们之间的相似性和差异性。
下面将对表达谱和基因比对进行更详细的介绍:表达谱(Transcriptome)表达谱是指一个生物体在特定时间和空间范围内的所有基因的表达情况。
即在特定条件下,所有基因的表达情况构成的基因表达谱。
通过对不同条件下的一个组织或细胞的基因表达谱的分析,我们可以了解其生理和病理过程中的基因表达变化情况。
表达谱研究的主要手段是基因芯片和高通量测序技术。
在基因芯片中,已经包含了许多基因的探针,把探针固定到芯片上,使用芯片上的探针来探测目标基因的表达情况。
而在高通量测序技术中,可以对所有基因进行高通量测序,得到基因的拷贝数或RNA浓度,进而了解基因的表达情况。
表达谱在医学研究中有着广泛的应用,例如可以用来分析癌症中的基因表达谱,发现与肿瘤相关的基因,寻找潜在的治疗靶点。
基因比对(Alignment)基因比对是指对比不同生物个体之间基因序列的相似性和差异性。
在基因比对中,首先将两个或多个基因组进行比对,找到它们之间的相似位置。
然后,再在相似位置上比较两个基因组的差异,并进行分类、注释和解释。
基因比对的主要手段是计算机算法程序。
其中最常用的是BLAST程序(Basic Local Alignment Search Tool),它是一种快速的基因比对算法,可以快速找到两个序列中的相似性区域。
基因比对在基因研究和生物学研究中有着重要的应用。
例如,可以用来发现特定物种中的家族基因、转座子和RNA序列等。
总结:表达谱和基因比对是生物学和基因研究中的两个重要概念。
表达谱可以帮助研究人员了解基因表达在特定生理和病理过程中的变化情况,而基因比对则可以揭示不同生物个体之间基因序列的相似性和差异性。
这些信息可以帮助我们更好地理解生物系统的运作机理,为生物研究提供更多的基础知识。
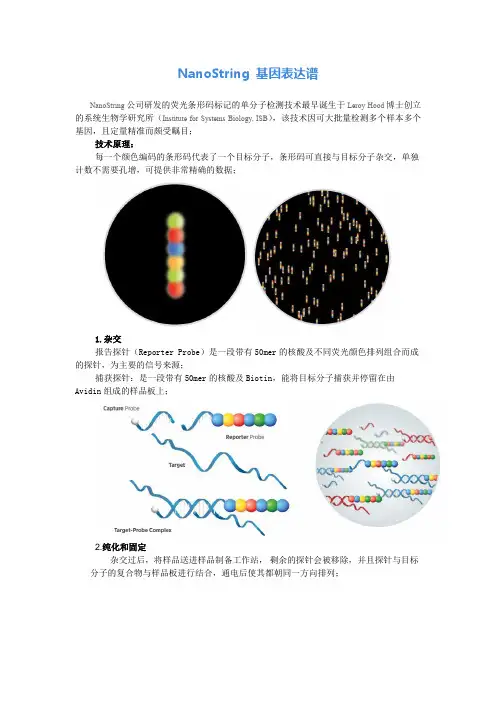
NanoString 基因表达谱NanoString公司研发的荧光条形码标记的单分子检测技术最早诞生于Leroy Hood博士创立的系统生物学研究所(Institute for Systems Biology, ISB),该技术因可大批量检测多个样本多个基因,且定量精准而颇受瞩目;技术原理:每一个颜色编码的条形码代表了一个目标分子,条形码可直接与目标分子杂交,单独计数不需要孔增,可提供非常精确的数据;1.杂交报告探针(Reporter Probe)是一段带有50mer的核酸及不同荧光颜色排列组合而成的探针,为主要的信号来源;捕获探针:是一段带有50mer的核酸及Biotin,能将目标分子捕获并停留在由Avidin 组成的样品板上;2.纯化和固定杂交过后,将样品送进样品制备工作站,剩余的探针会被移除,并且探针与目标分子的复合物与样品板进行结合,通电后使其都朝同一方向排列;3.计数将处理好的样品板放入数据分析仪进行信号数据采集,仪器会自动统计在样品板表面的荧光编码探针,并将其列表表示出来;NanoString技术应用:Nanostring技术的特点及优势使其被越来越广泛地应用到生物医学前沿领域,包括高通量基因表达结果验证、基因表达谱研究、基因调控机理、基因调控网络研究、临床疾病分子分型及诊断预后等领域;其研究涉及动植物发育、炎症、免疫、干细胞、肿瘤、药物耐药、信号转导等各个领域。
基因表达分析NanoString技术的基因表达条形码提供了一个高质量低成本的检测方式,可同时分析超过800个基因的表达水平,并且精准性与qPCR相当这项技术的实验方法简单,每个反应只需15分钟的人工操作,可直接测定组织和血液裂解产物和FFPE提取物。
检测特点:∙可同时分析超过800个基因;∙不需要荧光定量反应,不需要扩增和逆转录;∙可直接检测包括总RNA,细胞和全血裂解产物以及石蜡包埋组织(FFPE)等样品;∙采用单分子计数使数据误差最小化;灵敏度高,重现性好,动力学范围广;自定义条形码:提供了最大限度的灵活性,可自主设计特定的基因、组织和很难实现的目标序列;仪器分析性能:具体应用:1.Northcott PA, et al., “Rapid, reliable, and reproducible molecular sub-grouping of clinical medulloblastoma samples”Acta Neuropathologica123(4): 615-26. November 06, 20112. Sun Z, et al.,“Integrated Analysis of Gene Expression, CpG Island Methylation, and Gene Copy Number in Breast Cancer Cells by Deep Sequencing”PLoS ONE Vol 6-2, e17490. February 25, 20113. Reis PP, et al.,“mRNA transcript quantification in archival samples using multiplexed, color-coded probes.”BMC Biotechnol vol. 108 no. 19 7956-7961. May 09, 2011。

基因表达谱芯片数据分析及其Bioconductor实现1.表达谱芯片及其应用表达谱DNA芯片(DNA microarrays for gene expression profiles)是指将大量DNA片段或寡核昔酸固定在玻璃、硅、塑料等硬质载体上制备成基因芯片,待测样品中的mRNA被提取后,通过逆转录获得cDNA,并在此过程中标记荧光,然后与包含上千个基因的DNA芯片进行杂交反应30min~20h后,将芯片上未发生结合反应的片段洗去,再对玻片进行激光共聚焦扫描,测定芯片上个点的荧光强度,从而推算出待测样品中各种基因的表达水平。
用于硏究基因表达的芯片可以有两种:①cDNA芯片;② 寡核昔酸芯片。
cDNA芯片技术及载有较长片段的寡核昔酸芯片采用双色荧光系统:U前常用Cy3—dUTP (绿色)标记对照组mRNA, Cy5—dUTP (红色)标记样品组mRNAUl。
用不同波长的荧光扫描芯片,将扫描所得每一点荧光信号值自动输入计•算机并进行信息处理,给出每个点在不同波长下的荧光强度值及其比值(ratio值),同时计算机还给出直观的显色图。
在样品中呈高表达的基因其杂交点呈红色,相反,在对照组中高表达的基因其杂交点呈绿色,在两组中表达水平相当的显黄色,这些信号就代表了样品中基因的转录表达情况⑵。
基因芯片因具有高效率,高通量、高精度以及能平行对照研究等特点,被迅速应用于动、植物和人类基因的研究领域,如病原微生物毒力相关基因的。
基因表达谱可直接检测mRNA的种类及丰度,可以同时分析上万个基因的表达变化,来揭示基因之间表达变化的相互关系。
表达谱芯片可用于研究:①同一个体在同一时间里,不同基因的表达差异。
芯片上固定的已知序列的cDNA或寡聚核昔酸最多可以达到30 000多个序列,与人类全基因组基因数相当,所以基因芯片一次反应儿乎就能够分析整个人的基因⑶。
②同一个体在不同时间里,相同基因的表达差异。
③不同个体的相同基因表达上的差异。

基于基因表达谱的肿瘤筛选技术 肿瘤是一种恶性病变,是生物学研究的热点之一,不同的肿瘤种类有着不同的致病机制和治疗方案。因此,确定肿瘤的类型,发展程度和治疗方案是非常重要的。众所周知,基因突变是癌症的一个重要因素之一,因此,基于基因表达谱的肿瘤筛选技术已经成为了肿瘤学研究的一个重点。本文将介绍基于基因表达谱的肿瘤筛选技术的原理和应用。
一、基因表达谱的形成和意义 基因的表达是由基因本身转录成RNA,然后将RNA翻译成蛋白质,从而形成蛋白质之间的相互作用。因此,基因表达的水平决定了生物体内蛋白质的数量和种类。肿瘤发生时,由于各种因素的作用,基因的表达谱也会发生变化,这是肿瘤起始和发展的重要表现。
基于此,科学家选择测量肿瘤细胞和正常细胞中基因表达差异的方法,形成基因表达谱。基因表达谱是将细胞的RNA转录成DNA,并用DNA微阵列或RNA测序等方法分析每个基因在正常和肿瘤细胞之间的表达差异的一种方法。基因表达谱的大小和复杂性取决于RNA样品的来源和研究的目的,但它可以提供有效的筛选和发现不同肿瘤的诊断标志物和生物学通路。
二、基于基因表达谱的肿瘤筛选技术 基于基因表达谱的肿瘤筛选技术可以将肿瘤分为不同类型,并对每种类型的肿瘤进行不同的治疗方案。在肿瘤筛选中,科学家首先要对肿瘤样本中的RNA进行提取和纯化,并通过DNA串级反应(PCR)等方法将其扩增和记帐,然后运用不同的基因表达分析平台对基因表达谱进行分析。这些分析平台包括DNA微阵列,RNA序列和单细胞RNA测序等。
此外,科学家还可以使用许多技术,如聚类分析,差异表达和信号通路分析等,对这些表达谱进行进一步的分析。通过这些技术,科学家可以对不同类型的肿瘤进行鉴定和研究,并选择最合适的治疗方案。虽然这种方法很有效,但其局限性在于分析结果的真实性受到多种因素的影响(例如RNA提取和纯化效率等)。
三、基于基因表达谱的肿瘤诊断和治疗 基于基因表达谱的肿瘤筛选技术已经被广泛地应用于肿瘤的筛选和早期诊断。例如,在乳腺癌中,科学家发现了大量的基因表达谱的改变,使得诊断和治疗更加准确和有效。除此之外,这种技术还可以帮助科学家更好地理解肿瘤的发生和发展机制,从而推进肿瘤学的研究和治疗。

对于基因表达谱数据的分析是生物信息学的研究热点和难点。
转化为数学问题,分析任务是从数据矩阵 M 中找出显著性结构,结构类型包括全局模型 (model) 和局部模式 (pattern) 。
对基因表达谱数据的分析是数据挖掘问题,所采用的方法包括通过可视化进行探索性数据分析( Exploratory Data Analysis )、描述建模 (descriptive modeling) 、分类、聚类、回归和机器学习等。
基因表达谱分析所采用的常用方法是聚类,其目的就是将基因分组。
从数学的角度,聚类得到的基因分组,一般是组内各成员在数学特征上彼此相似,但与其它组中的成员不同。
从生物学的角度,聚类分析方法所隐含的生物学意义或基本假设是,组内基因的表达谱相似,它们可能有相似的功能。
然而,产物有相同功能的编码基因(例如对其它蛋白质有磷酸化作用),不一定共享相似的转录模式。
相反,有不同功能的基因可能因为巧合或随机扰动而有相似的表达谱。
尽管有许多意外的情况存在,大量功能相关的基因的确在相关的一组条件下有非常相似的表达谱,特别是被共同的转录因子共调控的基因,或者产物构成同一个蛋白复合体,或者参与相同的调控路径。
因此,在具体的应用中,可以根据对相似表达谱的基因进行聚类,从而指派未知基因的功能。
聚类分析是模式识别和数据挖掘中普遍使用的一种方法,是基于数据的知识发现的有效方法,特别适用于模式分类数不知道的情况。
聚类分析是一种无监督学习方法,不需要任何先验领域知识,它根据数学特征提取分类标准,对数据进行分类,这种数学特征的例子有统计平均值、相关系数、协方差矩阵的本征值及本征向量等。
聚类分析在基因表达数据分析中应用得很多,主要有层次聚类、 K 均值、自组织特征映射网络等。
本节将介绍基因表达数据分析中常用的聚类方法及与此相关的内容。
8.4.1 相似性度量函数对基因表达谱进行聚类分析之前,必须首先确定反映不同基因表达谱相似程度的度量函数,根据该函数可以将相似程度高的基因分为一类。

基因表达数据分析方法及其应用研究共3篇基因表达数据分析方法及其应用研究1随着技术的不断发展,基因表达数据分析在生命科学研究中扮演着越来越重要的角色。
基因表达数据分析是研究基因功能的关键一步,它使得科学家可以了解基因在特定情况下的表达水平。
在本文中,我们将讨论基因表达数据分析的方法及其应用。
1.基因表达数据的来源和类型基因表达数据是通过分析转录组和基因芯片等数据获得的。
转录组技术通过测量RNA浓度,包括RNA-seq和microarray。
而基因芯片就是一种将成千上万的基因测量并呈现的芯片。
基因表达数据存在多种类型,包括原始数据、表达矩阵、差异表达矩阵、注释文件和元数据等等。
2. 基因表达数据分析的方法(1)数据清理数据清理是数据分析过程中的第一步。
它包括数据预处理、去除冗余数据、去除噪声和填补数据空缺等操作。
(2)正则化正则化的目的是调整不同基因表达数据之间的差异,消除数据中的计量误差和探测效率的误差。
几种正则化方法包括平滑、归一化和标准化。
(3)差异分析差异分析是研究基因表达数据中各基因在不同样品之间差异的方法。
常用的差异分析方法包括t-test、ANOVA、FDR和q值等。
(4)聚类分析聚类分析是将数据根据观察指标相似度进行分类的方法。
在基因表达数据上,它通常用于发现不同条件下的基因表达模式。
(5)变异分析变异分析是一种寻找表达值变异的基因的方法。
通常,基因的变异程度与其在癌症和其他疾病中的作用有关。
(6)功能注释功能注释是将基因表达数据与已知基因功能相结合的方法,从而获得数据更深层次的信息。
它通常用于解释基因表达数据的生物学意义,如基因表达数据和肿瘤发展的相关性等。
3.应用研究基因表达数据分析可应用于许多研究领域,包括基因表达和调控、单细胞分析和肿瘤生物学等。
(1)基因表达和调控基因表达数据分析可用于挖掘基因之间的相互关系以及调控通路。
这些信息可以在理解细胞生物学、发育及疾病发生机制的过程中发挥重要作用。
空间转录组学技术
空间转录组学技术是一种利用分子技术来分析组织或细胞中基
因表达谱的技术。
与传统的转录组学技术不同的是,空间转录组学技术可以在细胞和组织水平上分析基因表达谱,从而为研究细胞和组织的功能和发展提供更详细的信息。
空间转录组学技术的主要方法是利用高通量的原位杂交技术来
分析基因表达谱。
这种技术可以将荧光探针附着到组织切片或细胞中,然后使用荧光显微镜观察表达基因的位置和数量。
通过对不同组织和细胞中的基因表达谱进行比较,可以得出基因在不同组织和细胞中的差异表达情况。
空间转录组学技术已经被广泛应用于生物学、医学和农业领域。
例如,在生物学中,它可以用来研究发育过程中基因表达的空间和时间特异性;在医学中,它可以用来研究疾病发生的分子机制;在农业领域,它可以用来研究植物的生长和发育过程中基因表达的变化。
总之,空间转录组学技术是一种强大的分子工具,可以提供对组织和细胞水平上基因表达谱的全面分析,从而为生物学、医学和农业领域的研究提供更深入的了解和启示。
- 1 -。
生物大数据技术中的关键基因筛选方法解析随着生物技术的迅猛发展,生物大数据的量不断增长。
这些丰富的生物大数据储存了大量的基因信息,对于分析基因功能和疾病的发生机制具有重要意义。
然而,如何从庞大的生物大数据中筛选出关键基因成为了一个挑战。
本文将重点分析生物大数据技术中的关键基因筛选方法,为基因研究和疾病治疗提供参考。
一、基因表达谱分析基因表达谱是指不同组织或条件下基因表达的量。
通过分析基因表达谱,可以找到在不同机体阶段或疾病条件下显著上调或下调的基因。
常用的基因表达谱分析方法包括基因芯片和RNA测序。
基因芯片是一种高通量的基因表达谱分析方法。
它可以同时检测上万个基因在样本中的表达水平,进而找出与特定生物过程或疾病相关的基因。
通过比较不同样本的基因表达谱,可以筛选出在不同生理或病理条件下显著变化的基因。
RNA测序是一种新兴的基因表达谱分析方法,具有更高的灵敏度和准确性。
相比之下,RNA测序可以检测到更低丰度的RNA分子,从而发现更多的潜在关键基因。
此外,RNA测序还可以确定剪接异构体和全长转录本的存在,为深入研究基因功能提供更多信息。
二、基因共表达网络构建与分析基因共表达网络是基于基因表达谱数据构建的一个图结构,其中基因是节点,而它们之间的关联是边。
通过基因共表达网络,可以识别出在相似条件下共同调控的基因模块,进而挖掘出关键的基因。
构建基因共表达网络的常用方法包括Pearson相关系数、相关网络和共表达模块分析。
Pearson相关系数可用于度量基因间的相似性,并且基于相似性构建网络。
相关网络可以通过阈值设定来筛选出显著相关的基因对,从而构建基因共表达网络。
共表达模块分析是一种从基因共表达网络中识别出功能相关基因模块的方法。
通过计算基因之间的相似度,可以使用聚类算法将基因分组到不同的模块中。
每个模块可以表示一个功能或通路,从中可以推测出关键的基因。
三、基因组关联分析基因组关联分析是一种通过比较基因组上的变异和表型差异来寻找关键基因的方法。
基因表达水平的计算和分析方法基因表达是生命活动的基础,每个细胞都依赖于基因表达来维持正常的生理功能。
随着高通量技术的发展,越来越多的基因表达数据积累,如何从这些数据中提取有价值的信息成为生物数据分析领域的重要研究方向之一。
基因表达数据分析的核心是基因表达水平的计算和分析方法。
一、基因表达水平的计算方法基因表达水平的计算方法有三种:基于芯片、基于RNA-seq和蛋白质组学分析法。
1.基于芯片的计算方法基于芯片的基因表达计算方法是通过DNA芯片技术,采用荧光信号分析技术来计算基因表达水平。
基本上分为以下步骤:首先将RNA转化为cDNA,并标记为荧光物质并打上芯片;通过荧光信号分析技术对荧光素的强度进行检测,并将强度值转换为基因表达水平。
这种方法已经被广泛应用,但是具有一定的局限性。
芯片技术复杂、成本高昂、对特定基因有选择性和检测范围有限等缺点,限制了其在大规模研究和应用中的应用。
2.基于RNA-seq的计算方法RNA-seq技术是最新的一种高通量测序技术,可同时检测所有共同的基因表达和新的转录本,以及所有SNP和突变等分子标记,分子分类和转录水平分布。
它可以使有限的生物材料得到有效和高精度的序列,并在不断更新的转录本数据库中分析和基因注释。
RNA-seq可以直接测量RNA转录本的量,并不是通过荧光信号而是通过量化RNA-seq文库中测序reads的数目来计算基因表达的水平。
可以检测很少的RNA,扩大了涉及的基因范围和研究范围,大大提高了检测效率和准确率。
3.蛋白质组学分析法蛋白质组学方法是一种更直接的基因表达水平测量方法。
蛋白质是基因表达的最终产物,是表达的直接结果,可以反映基因表达水平的真实状态。
蛋白质组学方法通过进行质谱分析,测量蛋白质的多肽序列,进而实现对基因表达水平的定量。
二、基因表达水平的分析方法基因表达水平的分析方法可以分为两个方面:一是对数据进行预处理并分析数据的结构,二是基于分析的结果进行生物学的解释和计算。
差异基因表达差异基因表达是指在不同生物体或不同组织、不同发育阶段、不同环境条件下,基因在转录和翻译过程中表达水平的变化。
这种差异使得生物体能够适应不同的环境和生理状态,并发挥出不同的功能。
差异基因表达的研究对于理解生物体的发育、适应和进化具有重要意义。
差异基因表达的研究主要通过基因表达谱分析来实现。
基因表达谱分析是指对不同样本中的基因表达情况进行比较和分析,以寻找差异表达的基因。
最常用的方法是基于高通量测序技术的RNA-seq和微阵列技术。
差异基因表达的发现不仅可以揭示不同生物体之间的差异,还可以帮助我们理解疾病的发生机制。
通过比较病人和正常人的基因表达谱,可以发现和疾病相关的差异基因。
这些差异基因可能是疾病的致病基因或潜在治疗靶点。
例如,通过对癌症组织和健康组织的基因表达谱进行比较,可以发现与癌症相关的差异基因,从而为癌症的诊断和治疗提供新的线索。
差异基因表达的研究还可以帮助我们理解基因调控网络的结构和功能。
基因调控网络是由一系列相互作用的基因和调控元件组成的复杂网络。
通过分析差异基因表达的调控网络,可以揭示基因间的相互作用关系和调控机制。
这对于理解生物体的发育和功能具有重要意义。
差异基因表达的研究不仅限于生物医学领域,还涉及到农业、生态学和进化生物学等领域。
例如,通过比较不同品种或不同环境条件下作物的基因表达谱,可以发现与产量、品质和抗性相关的差异基因,从而为作物改良提供新的思路和方法。
差异基因表达的研究对于理解生物体的发育、适应和进化具有重要意义。
通过分析差异基因表达,我们可以揭示基因调控网络的结构和功能,发现与疾病和重要农作物性状相关的差异基因。
差异基因表达的研究将为生物医学、农业和生态学等领域的科研和应用提供新的思路和方法。
基因功能的实验验证方法
一、基因敲除和回复实验
基因敲除实验是通过特定的方法将基因敲除或沉默,观察其敲除或沉默后对细胞或生物体的影响,从而验证基因的功能。
回复实验则是将敲除的基因恢复,以验证基因的缺失是导致表型改变的原因。
二、基因转录和翻译水平检测
1.转录水平检测:通过逆转录PCR(RT-PCR)等技术检测基因的转录产物,
了解基因在不同条件下的表达水平,从而验证基因的功能。
2.翻译水平检测:通过Western blot等技术检测基因的蛋白质产物,了解蛋
白质的表达水平和修饰情况,进一步验证基因的功能。
三、基因表达谱分析
基因表达谱分析是通过高通量测序等技术检测细胞或组织中所有基因的表达情况,了解基因在不同条件下的表达差异,从而筛选出与特定生物学过程相关的基因,进一步验证其功能。
四、蛋白质相互作用研究
蛋白质相互作用研究是通过特定的实验方法检测蛋白质之间的相互作用,了解蛋白质之间的相互关系和作用机制,从而验证基因的功能。
常见的蛋白质相互作用研究方法包括酵母双杂交、免疫共沉淀等。
五、生物信息学分析
生物信息学分析是通过计算机技术和算法对生物数据进行分析和挖掘,了解基因的结构、序列、表达和功能等信息,从而验证基因的功能。
常见的生物信息学分析方法包括基因注释、基因网络分析等。
分子通路三个方法摘要:1.分子通路概述2.方法一:基因表达谱分析3.方法二:高通量测序4.方法三:生物信息学分析5.总结与展望正文:分子通路是生物体内细胞调控的重要机制,通过一系列相互作用分子组成的网络来实现生物过程的调节。
在生物学研究中,揭示分子通路的研究方法不断发展。
本文主要介绍了三种研究分子通路的方法:基因表达谱分析、高通量测序和生物信息学分析。
一是基因表达谱分析。
基因表达谱是通过检测细胞在不同条件下基因的表达水平来研究分子通路的调控机制。
这种方法可以揭示特定条件下基因的激活或抑制情况,为进一步研究分子通路提供重要线索。
通过比较不同样本的基因表达谱,研究者可以发现分子通路中的关键基因和调控节点,从而揭示生物学过程的分子机制。
二是高通量测序技术。
高通量测序技术已经成为研究分子通路的重要手段。
这种方法可以大规模地检测基因、RNA和蛋白质等生物大分子的信息,为揭示分子通路提供详细的数据支撑。
高通量测序技术在转录组、蛋白质组和代谢组等多个水平上全面解析分子通路,有助于深入了解生物过程的调控机制。
三是生物信息学分析。
生物信息学方法通过对高通量测序数据进行深度挖掘,揭示分子通路中的关键节点和调控因子。
生物信息学方法包括数据挖掘、模式识别、机器学习等技术,可以有效地分析分子通路中的复杂关系。
通过生物信息学分析,研究者可以从一个全新的角度审视分子通路,并为实验研究提供理论指导。
总之,分子通路研究是一个多学科交叉的领域,基因表达谱分析、高通量测序和生物信息学分析等方法为研究分子通路提供了强大的工具。
随着技术的不断发展,未来还会有更多高效、便捷的研究方法涌现,帮助我们更好地理解生物体内的分子调控机制。
基因表达谱分析技术1微阵列技术(microarray)这是近年来发展起来的可用于大规模快速检测基因差别表达、基因组表达谱、DNA序列多态性、致病基因或疾病相关基因的一项新的基因功能研究技术。其原理基本是利用光导化学合成、照相平板印刷以及固相表面化学合成等技术,在固相表面合成成千上万个寡核苷酸“探针”(cDNA、ESTs或基因特异的寡核苷酸),并与放射性同位素或荧光物标记的来自不同细胞、组织或整个器官的DNA或mRNA反转录生成的第一链cDNA进行杂交,然后用特殊的检测系统对每个杂交点进行定量分析。其优点是可以同时对大量基因,甚至整个基因组的基因表达进行对比分析。包括cDNA芯片(cDNAmicroarray)和DNA芯片(DNAchips)。cDNA芯片使用的载体可以是尼龙膜,也可以是玻片。当使用尼龙膜时,目前的技术水平可以将20000份材料点在一张12cm×18cm的膜上。尼龙膜上所点的一般是编好顺序的变性了的双链cDNA片段。要得到基因表达情况的数据,只需要将未知的样品与其杂交即可。杂交的结果表示这一样品中基因的表达模式,而比较两份不同样品的杂交结果就可以得到在不同样品中表达模式存在差异的基因。杂交使用的探针一般为mRNA的反转录产物,标记探针使用32PdATP。如果使用玻片为载体,点阵的密度要高于尼龙膜。杂交时使用两种不同颜色的荧光标记不同的两份样品,然后将两份样品混合起来与一张芯片杂交。洗去未杂交的探针以后,能够结合标记cDNA的点受到激发后会发出荧光。通过扫描装置可以检测各个点发出荧光的强度。对每一个点而言,所发出的两种不同荧光的强度的比值,就代表它在不同样品中的丰度。一般来讲,显示出来的图像中,黄色的点表示在不同的样品中丰度的差异不大,红色和绿色的点代表在不同样品中其丰度各不相同。使用尼龙膜为载体制作cDNA芯片进行研究的费用要比玻片低,因为尼龙膜可以重复杂交。检测两种不同的组织或相同组织在不同条件下基因表达的差异,只需要使用少量的尼龙膜。但是利用玻片制作的cDNA芯片灵敏度更高,而且可以使用2种探针同时与芯片杂交,从而降低了因为杂交操作带来的差异;缺点是无法重复使用还必须使用更为复杂的仪器。Guo等(2004)将包含104个重组子的cDNA文库点在芯片上,用于检测拟南芥叶片衰老时的基因表达模式,得到大约6200差异表达的ESTs,对应2491个非重复基因。其中有134个基因编码转录因子,182个基因预测参与信号传导,如MAPK级联传导路径。Li等(2006)设计高密度的寡核苷酸tilingmicroarray方法,检测籼稻全基因组转录表达情况。芯片上包含13,078,888个36-mer寡核苷酸探针,基于籼稻全基因组shot-gun测序的序列合成,大约81.9%(35,970)的基因发生转录事件。Hu等(2006)用含有60,000寡核苷酸探针(代表水稻全部预测表达基因)的芯片检测抗旱转基因植株(过量表达SNAC1水稻)中基因的表达情况,揭示大量的逆境相关基因都是上升表达的。
2基因表达系列分析(Serialanalysisofgeneexpression,SAGE)基因表达系列分析(SAGE)是一种转录物水平上研究细胞或组织基因表达模式的快速、有效的技术,也是一种高通量的功能基因组研究方法,它可以同时将不同基因的表达情况进行量化研究(Velculescuetal.,1995)。SAGE的基本原理是:每一条mRNA序列都可以用它包含的9bp的小片段(TAG)代替,因此考查这些TAGs出现的频率就能知道每一种mRNA的丰度。首先利用生物素标记的oligo(dT)引物将mRNA反转录成双链cDNA,然后利用NlaIII酶切双链cDNA。NlaIII酶的识别位点只有4bp,因此cDNA都被切成几十bp的小片段。带有生物素标记的小片段cDNA被分离出来,平均分成2份。这2份cDNA分别跟2个接头连接,2个接头中均有一个FokI酶切位点。FokI是一种IIS型核酸内切酶,其识别位点不对称,切割位点位于识别位点下游9bp且不依赖于特异的DNA序列。FokI酶切分成2份的cDNA之后,带有部分接头序列的TAGs就被释放下来。这时将2份cDNA混合起来,进行连接反应。根据接头序列设计引物扩增连接反应的产物,然后通过NlaIII酶切PCR产物得到连接在一起的两个TAG,即ditag。将ditags串连起来,克隆到质粒载体中。一般每个克隆包含50个左右的ditags。这些克隆经过PCR扩增然后测序。因为每一个ditag之间都是以NlaIII识别位点间隔的,所以很容易对每个的ditag进行区分和计数。而每一个TAG在SAGE库中出现的频率就代表了该基因的表达水平。利用SAGE可以在短期内得到丰富的表达信息,与直接测定cDNA克隆序列方法相比,减少了大量的重复测序,从而大大节省了研究时间和费用。这种方法对正常、癌基因旁、癌组织中基因的差异表达研究方面还有独到的优点,有助于发现肿瘤特异基因。首次运用SAGE技术是分析1000个胰腺基因的表达情况(Velculescuetal.,1995),结果都显示它是一种有效的功能基因组研究方法。最早用SAGE技术进行研究的高等植物是水稻(Matsumuraetal.,1999)。运用SAGE技术检测了水稻中来源于6000个基因10000个TAGs。SAGE分析显示绝大部分高表达基因都是看家基因,这些基因的mRNA在总RNA中的比例超过1%。在无氧处理和对照的幼苗中分析了2000个左右的TAGs,结果显示多数基因的表达没有变化。有趣的是,发酵途径相关的基因同样没被检测到。根据TAG序列设计引物,成功扩增出了延伸因子EF-1a0.2kb的cDNA片段。这一事实说明利用9bp的TAG序列和4bp的NlaIII识别位点序列,可以设计引物用于长片段cDNA的获得。
3cDNA-AFLPcDNA-AFLP是从基因组AFLP方法(Vosetal.,1995)发展来的RNA指纹技术。经典的cDNA-AFLP按照标准的AFLP方法进行操作,只不过模板变成了cDNA。这一方法包含3个步骤:a.将cDNA酶切并连上载体;b.利用包含选择性碱基的引物进行扩增;c.电泳及成像。一般选择两种限制性内切酶进行cDNA的消化,这两种酶一种的酶切位点为4个碱基,另一种为6个碱基。在植物基因组的AFLP操作中,需要添加3个进行选择性扩增。因为cDNA的复杂性比基因组低,所以cDNA-AFLP中每个引物只需要2个选择性碱基。聚丙烯酰胺凝胶电泳中,最大的cDNA-AFLP产物为1000bp左右,最小将近100bp。对马铃薯的已知cDNA序列分析发现,cDNA-AFLP中常用的两种酶只能同时切割大约45%的cDNA。如果使用这种方法,几乎一半的基因的表达情况是无法被揭示出来的,因此对cDNA-AFLP方法进行了修改,在cDNA的酶切时仅仅采用单酶切,这就是单酶切的cDNA-AFLP技术(Habuetal.,1997)。1999年,Kawamoto等人进一步发展了这项技术,他们称之为iAFLP技术。利用该技术可以同时检测多个样本的基因表达差异。cDNA-AFLP技术在植物中的首次运用,是Bachem等(1996)对马铃薯基因表达差异的研究。该研究分离到两个在马铃薯块茎形成过程中差异表达的cDNA片段。这两个基因的转录都是块茎特异性的;它们在15天的块茎中高度表达,但在其它组织中表达量很低。此后无论在分离抗逆相关基因的研究也有成功的报道(Ivashutaetal.,1999)。
4差异展示PCR(DifferentialDisplayReverseTranscriptionPCR,DDRT-PCR)DDRT-PCR是基于反转录和PCR的功能基因组研究方法,最早由Liang和Pardee(1992)报道。这种方法能有效地检测真核细胞中特定基因的表达模式,因而可以用于发现和克隆新的基因。DDRT-PCR技术的基本原理是扩增反转录产物得到分子量大小不同的cDNA片段。首先利用oligo(dT)引物反转录mRNA得到cDNA,然后利用相同的oligo(dT)引物和另一随机引物进行PCR扩增。到目前为止该技术已经得到了大量的改进。为了提高特异性,可以增加随机引物的特异性。碱基数增加以后,有可能包含某一特异基因家族的保守序列,这使得检测某一类基因的表达情况成为现实。分离DDRT-PCR的产物简单易行,可以通过变性或非变性的聚丙烯酰胺凝胶电泳(Liangetal.,1995)、毛细管电泳(Georgeetal.,1997)或普通琼脂糖电泳获得。DDRT-PCR产物的标记,可以使用[33P]dATP(SimonandOppenheimer,1996)、荧光(Jonesetal.,1997)或银染(Gottschlichetal.,1997)。因为这种方法非常简便,所以它得到了广泛的应用。自从该方法被发明以来,已有许多成功的报道,应用范围包括从微生物到人类的各个物种。在植物中运用DDRT-PCR方法得到了大量的cDNA克隆,从拟南芥中得到了一些控制生物钟的基因(Krepsetal.,2000),从大麦野生型品种和无根毛的突变体中克隆了一个控制根毛形成的基因HvEXPB1(BratanichandBlanchetot,2006)。该技术在生物对逆境条件适应的研究中同样得到了广泛的应用,为了弄清楚在多系统衰竭综合症中猪圆环病毒Ⅱ型侵入时发生的细胞学事件,利用DDRT-PCR鉴定了一些淋巴细胞内受此病诱导而的靶分子,其中两个差异调节转录本显示与人的基因有一定同源性,分别是RNA剪接因子和透明质酸介导的运动受体(KwasniewskiandSzarejko,2006)。
5抑制消减杂交(SuppressionSubtractiveHybridization,SSH)SSH方法是一种简便而高效地寻找差异表达基因的新方法。该方法是以抑制PCR为基础的cDNA消减杂交方法。所谓抑制PCR,是利用非目标序列片段两端的长反向重复序列在退火时产生“锅柄”结构,无法与引物配对,从而选择性地抑制非目标序列的扩增。SSH方法既利用了消减杂交技术的消减富集,又利用抑制PCR进行了高效的复性动力学富集。其全部流程包括4个主要步骤:a.分别反转录tester和driver的mRNA得到双链cDNA,然后利用RsaI酶切cDNA,并将酶切的testercDNA加上接头。b.用大大过量的driver与tester进行两轮杂交,对杂交产物进行抑制PCR扩增。c.将PCR产物克隆进质粒载体,转化后挑取文库。d.对SSH文库进行筛选。抑制差减杂交法不仅能分离高丰度差异表达基因,而且能有效富集低丰度差异表达基因。1996年Diatchenko等人构建了第一个SSH文库,并通过与人类Y染色体的Cosmid文库杂交证实了该方法的有效性。运用该技术仅通过一轮反应既可使特异表达的基因富集1000倍以上。过去几十年来,抑制差减杂交法广泛运用于动植物、微生物研究。在植物中主要用于组织器官、发育阶段和逆境差异表达基因的筛选(Gepsteinetal.,2003;Chuetal.,2004;Zengetal.,2006)。Gepstein等(2003)利用该方法从拟南芥中得到了约800个自然衰老相关的cDNA片段,Chu等(2004)使用该技术鉴定了702个与xa13介导的抗病反应相关的ESTs。到目前为止,SSH方法得到了很多改进和完善。为了进一步消除SSH中的背景,Rebrikov等(2000)提出了基于MOS(Mirrororientationselection)的抑制消减杂交,有效的提高了筛选效率。Zeng等(2006)将SSH和cDNAMicroarray技术联合运用,从棉花中筛选到了242个体细胞胚胎形成相关的cDNA片段,进一步证实了该方法的实用性。以上几种都是高通量筛选特异表达基因的方法,但是同时给我们带来大量重复或无意的数据。带来这一问题的一个原因是因为一种mRNA能产生不止一个信号。我们构建的cDNA文库和制作的cDNA芯片,都不可避免地会包含重复克隆。而如果我们使用单酶切cDNA-AFLP,那么每一条mRNA都最终会形成不止一条条带(Habuetal.,1997)。当然随着技术地改进,cDNA-AFLP中的冗余信息有可能被彻底除去(Matzetal.,1999)。但是DDRT-PCR的特点却决定了它一定会有重复数据。因为每一条mRNA通常都会跟不止一对引物结合,所以它会带来至少两条分子量大小不同的条带。另一个更重要的原因是只有少数的基因是真正差异表达的,而我们的检测到的差异很多只是背景。例如,在拟南芥中利用cDNA芯片技术对673个基因的分析发现,只有2个基因真正在两种材料中差异表达(Kehoeetal.,1999)。运用SAGE方法对水稻幼苗1500个基因的分析显示,仅有24个在无氧处理前后表达量有显著差异(Matsumuraetal.,1999)。到目前为止,如何消除组成性表达基因造成的背景仍然是一个急待解决的难题。虽然消减杂交提供了一个消除背景的方法,但是效果仍不是十分理想。