SSR分析的原理及操作技术报告
- 格式:ppt
- 大小:1.01 MB
- 文档页数:30

ssr标记原理
SSR标记,即简单重复序列标记,是一种以特异引物PCR为基础的分子标记技术。
它利用了DNA序列中的简单重复序列,这些重复序列通常由1-6个碱基组成,形成长串重复。
由于这些重复序列在不同个体间的数量存在差异,因此能揭示比其他标记技术更高的多态性。
SSR标记的基本原理是,根据微卫星序列两端互补序列设计引物,通过PCR反应扩增微卫星片段。
由于核心序列串联重复数目不同,能够用PCR的方法扩增出不同长度的PCR产物。
将这些产物进行凝胶电泳,根据分离片段的大小决定基因型并计算等位基因频率。
SSR 标记具有一些优点,如一般检测到的是一个单一的多等位基因位点、微卫星呈共显性遗传,可鉴别杂合子和纯合子、所需DNA量少等。
在采用SSR技术分析微卫星DNA多态性时,必须知道重复序列两端的DNA序列的信息。

SSR标记的原理及应用1. 什么是SSR标记?SSR标记(Server Side Rendering,服务器端渲染)是一种将动态生成的内容直接嵌入到HTML页面中的技术。
传统的JavaScript渲染技术(如SPA单页面应用)需要在浏览器中使用JS代码动态生成页面内容,而SSR标记则在服务器端将动态内容生成后,将其直接嵌入到HTML页面中,再传输给浏览器进行展示。
2. SSR标记的原理SSR标记的原理主要分为以下几个步骤:•接收客户端请求:服务器通过监听HTTP请求,获取客户端请求的URL。
•路由解析:服务器根据URL进行路由解析,确定请求的页面和相应的数据。
•数据获取:服务器从数据库或其他数据源获取相应的数据。
•模板渲染:服务器使用模板引擎将数据填充到HTML模板中,并生成标记好动态内容的HTML页面。
•HTML页面发送:服务器将生成的HTML页面发送给客户端。
•客户端渲染:浏览器接收到HTML页面后,在展示页面之前,会解析其中包含的JavaScript文件并执行,以完成页面的交互和渲染。
3. SSR标记的优势相比于传统的客户端渲染,SSR标记具有以下几个优势:•更快的首次加载速度:SSR标记在服务器端生成带有动态内容的HTML页面,因此用户第一次请求页面时可以直接获取到完整的页面内容,无需等待JavaScript文件的加载和执行。
这可以显著减少首次加载的时间,提高用户体验。
•更好的SEO效果:由于SSR标记在服务器端生成静态HTML页面,搜索引擎爬虫可以直接读取页面内容,获取更多的有效信息,从而提高网页的收录率和排名。
•更低的服务器负载:SSR标记将页面的渲染工作放在了服务器端,相比于传统的客户端渲染,可以减轻浏览器的压力,降低服务器负载。
4. SSR标记的应用场景SSR标记在以下几个场景中具有广泛的应用:•需要更好SEO效果的网站:对于需要被搜索引擎爬虫收录的网站,使用SSR标记可以提高网站的可见性和排名,增加自然流量。

SSR:微卫星DNA又叫简单重复序列,指的是基因组中由1~6个核苷酸组成的基本单位重复多次构成的一段DNA,广泛分布于基因组的不同位置,长度一般在200bp以下。
研究表明,微卫星在真核生物的基因组中的含量非常丰富,而且常常是随机分布于核DNA中。
微卫星中重复单位的数目存在高度变异,这些变异表现为微卫星数目的整倍性变异或重复单位序列中的序列有可能不完全相同,因而造成多个位点的多态性。
如果能够将这些变异揭示出来,就能发现不同的SSR在不同的种甚至不同个体间的多态性,基于这一想法,人们发展起了SSR标记。
SSR标记又称为sequence tagged microsatellite site,简写为STMS,是目前最常用的微卫星标记之一。
由于基因组中某一特定的微卫星的侧翼序列通常都是保守性较强的单一序列,因而可以将微卫星侧翼的DNA片段克隆、测序,然后根据微卫星的侧翼序列就可以人工合成引物进行PCR扩增,从而将单个微卫星位点扩增出来。
由于单个微卫星位点重复单元在数量上的变异,个体的扩增产物在长度上的变化就产生长度的多态性,这一多态性称为简单序列重复长度多态性(SSLP),每一扩增位点就代表了这一位点的一对等位基因。
由于SSR重复数目变化很大,所以SSR标记能揭示比RFLP高得多的多态性,这就是SSR标记的原理。
? 与其它分子标记相比,SSR标记具有以下优点:(1)数量丰富,覆盖整个基因组,揭示的多态性高;(2)具有多等位基因的特性,提供的信息量高;(3)以孟德尔方式遗传,呈共显性;(4)每个位点由设计的引物顺序决定,便于不同的实验室相互交流合作开发引物。
因而目前该技术已广泛用于遗传图谱的构建〔11,12,18,19,33〕、目标基因的标定〔8,9,21,22,26〕、指纹图〔22〕的绘制等研究中。
但应看到,SSR标记的建立首先要对微卫星侧翼序列进行克隆、测序、人工设计合成引物以及标记的定位、作图等基础性研究,因而其开发费用相当高,各个实验室必须进行合作才能开发更多的标记。

SSR分析的原理及操作技术SSR(Server-Side Rendering)是一种用于开发 web 应用程序的技术,它主要通过将应用程序的初始渲染过程从客户端转移到服务器端来提供更好的性能和用户体验。
SSR 在加载时可以直接在服务器上渲染出完整的 HTML 页面,然后将其发送给客户端显示,而不是在客户端上等待JavaScript 渲染完成后再显示内容。
这种技术可以加快页面加载速度,提供更好的引擎优化(SEO)以及更好的用户体验。
1.数据请求:SSR首先会发送请求到服务器获取应用程序所需的数据,这样服务器端就能够预先获取数据并进行处理,然后将响应发送给客户端。
这样客户端在接收到HTML页面后就已经包含了所需的数据。
2.模版渲染:服务器会使用应用程序的模版引擎来渲染出初始的HTML页面。
模版引擎会根据数据和模版文件生成HTML页面,然后返回给客户端。
这样客户端接收到的HTML页面已经包含了服务器端渲染的内容,可以直接展示给用户。
SSR的操作技术主要包括以下方面:1. 选择框架:选择适合你的项目的 SSR 框架。
目前流行的 SSR 框架有 Next.js、Nuxt.js等。
这些框架为开发者提供了简化 SSR 开发流程的工具和规范。
2.路由配置:配置应用程序的路由,确保服务器能够正确地响应不同的路由请求,并加载相应的数据和渲染模版。
3. 数据获取:在服务器端获取数据的方法有多种,可以使用 AJAX请求、GraphQL、RESTful API等。
根据项目的需求选择合适的数据获取方式,并保证数据可以在服务器和客户端之间共享。
4. 模版引擎:选择适合你的项目的模版引擎,如 Handlebars、Pug 等。
模版引擎可以帮助你将数据和模版文件结合生成最终的 HTML 页面。
5. 客户端激活:在客户端接收到 HTML 页面后,需要编写JavaScript 代码来接管页面的交互和渲染。
这个阶段需要和服务器端渲染的 DOM 结构保持一致,可以使用一些库或框架来实现,如 React、Vue 等。




SSR技术1. SSR 简介说明:简单重复序列(Simple Sequence Repeat,SSR),简单重复序(SSR)也称微卫星DNA ,其串联重复的核心序列为1-6 bp,其中最常见是双核苷酸重复,即(CA) n 和(TG) n 每个微卫星DNA 的核心序列结构相同,重复单位数目10-60 个,其高度多态性主要来源于串联数目的不同。
SSR标记的基本原理:根据微卫星序列两端互补序列设计引物,通过PCR 反应扩增微卫星片段,由于核心序列串联重复数目不同,因而能够用PCR 的方法扩增出不同长度的PCR 产物,将扩增产物进行凝胶电泳,根据分离片段的大小决定基因型并计算等位基因频率。
在真核生物中,存在许多2-5bp 简单重复序列,称为“微卫星DNA”其两端的序列高度保守,可设计双引物进行PCR 扩增,揭示其多态性。
SSR具有以下一些优点:(l) 一般检测到的是一个单一的多等位基因位点;(2)微卫星呈共显性遗传,故可鉴别杂合子和纯合子;(3)所需DNA 量少。
显然,在采用SSR 技术分析微卫星DNA 多态性时必须知道重复序列两端的DNA 序列的信息。
如不能直接从DNA 数据库查寻则首先必须对其进行测序。
SSR的分类:根据SSR 核心序列排列方式的不同,可分为 3 种类型:1)完全型(perfect) ,指核心序列以不间断的重复方式首尾相连构成的DNA 。
如:ATATATATATATATATATATATATATATATATAT ;2)不完全型(imperfect) ,指在SSR 的核心序列之间有 3 个以下的非重复碱基,但两端的连续重复核心序列重复数大于 3 。
如:ATATATATGGATATATATATCGATATATATATATATATGGATATATATAT ;3)复合型(compound) ,指2 个或 2 个以上的串联核心序列由 3 个或3 个以上的连续的非重复碱基分隔开,但这种连续性的核心序列重复数不少于 5 。

ssr功能SSR(Server-Side Rendering)是一种用于网站开发的技术,它能够在服务器端将网页内容渲染成HTML,并将其发送到浏览器,从而使得用户在浏览器中能够更快地加载和渲染内容。
SSR功能的出现,给用户带来了更好的浏览体验,下面我将从其原理、优势和应用场景三个方面来详细介绍SSR功能。
首先,让我们来了解一下SSR的原理。
传统的Web应用在服务器端生成完整的HTML页面后,通过网络将其发送到浏览器,并在浏览器中进行渲染。
这种方式需要用户等待整个HTML页面加载完成后才能看到内容,对于大型网站来说,加载时间会很长,用户体验不佳。
而SSR的原理是在服务器端,将页面的首屏内容用服务器语言(比如Node.js)动态生成,并将其渲染成HTML返回给浏览器,浏览器再进行渲染。
这样,用户不需要等待整个页面加载完成,就能够快速看到首屏内容。
其次,SSR功能带来了许多优势。
首先,它能够提升网页的加载速度。
由于服务器端已经将页面渲染成HTML,所以用户在浏览器中只需要加载和渲染HTML,不需要执行JavaScript来生成页面,从而加快页面加载速度。
其次,SSR可以提升SEO效果。
由于搜索引擎(比如Google)爬取的是HTML内容,所以使用SSR可以使搜索引擎更好地抓取和索引网站的内容,提高网站的搜索排名。
另外,SSR还可以提供更好的用户体验。
由于用户能够快速看到首屏内容,所以整个页面的渲染过程会更加平滑,用户不会感到页面卡顿或闪烁。
最后,我们来看一下SSR功能的应用场景。
SSR适用于那些需要快速展示内容的网站,比如电商网站、新闻网站和社交网络平台等。
在电商网站上,用户经常需要快速查看商品信息、价格和库存等,使用SSR能够快速呈现给用户,提高购物体验。
在新闻网站上,用户需要及时获取最新的新闻内容,使用SSR能够快速加载新闻列表和正文,保证用户第一时间获取信息。
在社交网络平台上,用户需要快速查看好友的动态和留言,使用SSR能够快速展示给用户,提升用户参与度。
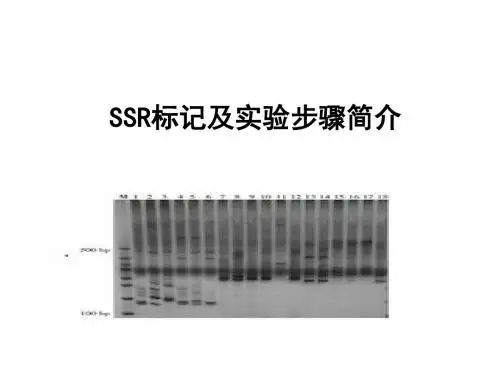

SSR分析遗传多样性SSR(Simple Sequence Repeat,简称SSR),也称为微卫星分子标记,是一类单核苷酸序列的重复结构,广泛分布在基因组中。
SSR具有高度多态性、遗传稳定性好、易于分析和重复性高等优点,因此在遗传多样性研究中得到广泛应用。
首先,选择适当材料是进行SSR分析的基础。
一般来说,选择具有代表性的材料样本,包括不同种群、地理分布范围广等特点的样本,以最大程度地反映种群的遗传多样性。
样本采集时应注意确保样本的纯度,避免杂交或污染。
同时,还要保证样本数量足够,以提高分析的准确性与可靠性。
其次,进行实验方法。
SSR分析主要包括DNA的提取、PCR扩增和基因分型三个步骤。
DNA提取是获取样本基因组DNA的关键步骤,要注意选择适当的提取方法,以保证提取的DNA质量和纯度。
PCR扩增是从样本DNA中扩增出所需的微卫星DNA序列,需要根据相关文献选择适当的引物。
在PCR扩增过程中,要注意避免扩增偏倚,并进行质控措施以确保扩增产物的准确性。
基因分型是利用PCR扩增得到的扩增产物进行电泳分析,可以采用聚丙烯酰胺凝胶电泳或毛细管电泳等方法。
电泳分析结果可用于构建遗传图谱、计算遗传相似性和种群遗传结构等。
最后,进行数据分析。
根据电泳分析结果,我们可以计算各个样本的等位基因频率和遗传多样性指数等。
等位基因频率是指不同等位基因在样本中的占比,可以用于估计种群的遗传多样性和变异程度。
遗传多样性指数包括多态信息内容(PIC)、香农信息指数(I)和Weir&Cockerham's Fst等。
PIC是一种测量位点多态性的指标,I是一种描述群体内基因多样性的指标,Fst则是一种测量种群间基因流动程度的指标。
这些指标的计算可以借助计算机软件进行。
总结来说,SSR分析遗传多样性是一种重要的分子标记技术,其研究流程包括材料选择、实验方法和数据分析等步骤。
通过SSR分析,可以揭示不同种群之间的遗传多样性特征,为种质资源鉴定、基因组定位和保护物种等方面的研究提供重要依据。
SSR技术1. SSR简介说明:简单重复序列(Simple Sequence Repeat,SSR),简单重复序(SSR)也称微卫星DNA,其串联重复的核心序列为1-6 bp,其中最常见是双核苷酸重复,即(CA) n和(TG) n每个微卫星DNA的核心序列结构相同,重复单位数目10-60个,其高度多态性主要来源于串联数目的不同。
SSR标记的基本原理:根据微卫星序列两端互补序列设计引物,通过PCR反应扩增微卫星片段,由于核心序列串联重复数目不同,因而能够用PCR的方法扩增出不同长度的PCR 产物,将扩增产物进行凝胶电泳,根据分离片段的大小决定基因型并计算等位基因频率。
在真核生物中,存在许多2-5bp简单重复序列,称为“微卫星DNA”其两端的序列高度保守,可设计双引物进行PCR扩增,揭示其多态性。
SSR具有以下一些优点:(l)一般检测到的是一个单一的多等位基因位点;(2)微卫星呈共显性遗传,故可鉴别杂合子和纯合子;(3)所需DNA量少。
显然,在采用SSR技术分析微卫星DNA多态性时必须知道重复序列两端的DNA序列的信息。
如不能直接从DNA数据库查寻则首先必须对其进行测序。
SSR的分类:根据SSR核心序列排列方式的不同,可分为3种类型:1)完全型(perfect),指核心序列以不间断的重复方式首尾相连构成的DNA。
如:ATATATATATATATATATATATATATATATATAT;2)不完全型(imperfect),指在SSR的核心序列之间有3个以下的非重复碱基,但两端的连续重复核心序列重复数大于3 。
如:ATATATATGGATATATATATCGATATATATATATATATGGATATATATAT;3)复合型(compound) ,指2个或2个以上的串联核心序列由3个或3个以上的连续的非重复碱基分隔开,但这种连续性的核心序列重复数不少于5 。
如:ATATATATATATATGGGATATATATATATA 。
苹果品种鉴定技术规程 ssr分子标记法全文苹果品种鉴定技术规程—— SSR分子标记法一、技术原理SSR(Simple Sequence Repeats)分子标记法是一种常用的遗传标记技术,也称为微卫星标记法。
该技术基于植物DNA中包含的重复序列,通过PCR扩增特定的SSR位点,进而对DNA序列进行分析,从而实现对苹果品种的鉴定。
二、实验步骤1. DNA提取:从苹果叶片或幼芽中提取基因组DNA。
2. SSR扩增反应:选择特定的SSR引物对DNA进行PCR扩增反应,得到扩增产物。
3. PCR产物分析:将PCR产品通过电泳方法分离在聚丙烯酰胺凝胶上,通过比较DNA片段迁移距离,鉴定不同苹果品种之间的差异。
三、实验材料1.离心管和PCR试管:用于提取DNA和进行PCR反应。
2. DNA提取试剂盒:用于提取DNA样品。
3. SSR引物组合:根据需要选择适宜的SSR引物。
4. PCR试剂盒:用于PCR反应。
5. DNA电泳试剂盒:用于DNA样品分离。
四、实验操作细节1. DNA提取:按照DNA提取试剂盒的说明进行操作,提取苹果叶片或幼芽中的基因组DNA。
2. SSR扩增反应:准备PCR反应体系,包括DNA模板、引物、酶和缓冲液等。
根据引物选择,设置合适数量的PCR反应管。
3. PCR条件设置:根据引物的特性,设置适当的扩增温度和周期。
4. PCR产物分析:将PCR反应产物通过电泳方法分离在聚丙烯酰胺凝胶上,根据DNA片段大小进行鉴定。
5.结果解读:根据电泳图像,比较不同苹果品种之间的DNA条带差异,判断品种间的差异和相似性。
五、结果分析1. SSR分离电泳图像:根据电泳分离的结果,分析苹果品种之间的遗传关系和差异。
2. SSR结构鉴定:通过比对已知品种的SSR位点,来反推未知品种的SSR结构和遗传关系。
3. SSR图谱构建:通过整理分析的SSR位点数据,构建苹果品种的SSR图谱,进一步研究苹果品种的遗传表达规律。
ssr分析的基本原理SSR分析的基本原理。
SSR(Server Side Rendering)是一种常见的前端渲染方式,它的基本原理是在服务器端将页面的 HTML、CSS 和 JavaScript 等资源进行渲染,然后再将渲染好的页面发送给客户端进行展示。
相比于客户端渲染,SSR 在页面加载速度、搜索引擎优化等方面有着明显的优势。
下面我们来详细了解一下 SSR 分析的基本原理。
首先,SSR 的基本原理是通过服务器端将页面进行渲染,生成完整的 HTML 页面,然后再将这个页面发送给客户端。
这样做的好处是可以让搜索引擎更好地理解页面的内容,提高页面的 SEO(Search Engine Optimization)效果,同时也可以加快页面的首次加载速度,提升用户体验。
其次,SSR 的实现原理通常是通过在服务器端使用一些特定的框架或工具来进行页面的渲染。
比如,在 React 中,可以使用 ReactDOMServer.renderToString() 方法来将组件渲染成字符串,然后将这个字符串作为 HTML 页面的内容返回给客户端。
在 Vue 中,可以使用 VueServerRenderer 来实现类似的功能。
另外,SSR 还需要考虑到一些与客户端渲染不同的问题,比如数据的获取和管理。
在客户端渲染中,通常是通过 AJAX 请求或者其他方式来获取数据,然后再将数据渲染到页面上。
而在 SSR 中,由于页面是在服务器端进行渲染的,因此需要考虑如何在服务器端获取和管理数据,以及如何将这些数据传递到客户端。
此外,SSR 还需要考虑到一些与客户端渲染不同的问题,比如数据的获取和管理。
在客户端渲染中,通常是通过 AJAX 请求或者其他方式来获取数据,然后再将数据渲染到页面上。
而在 SSR 中,由于页面是在服务器端进行渲染的,因此需要考虑如何在服务器端获取和管理数据,以及如何将这些数据传递到客户端。
综上所述,SSR 的基本原理是通过服务器端进行页面的渲染,然后再将渲染好的页面发送给客户端进行展示。
ssr分子标记技术及其在玉米种子鉴定上的应用随着现代农业的发展,种子质量的鉴定变得越来越重要。
其中,分子标记技术成为了种子鉴定的重要手段之一。
SSR分子标记技术是一种基于DNA序列多态性的分子标记技术,具有高度的稳定性、可重复性和高度的信息含量。
本文将介绍SSR分子标记技术及其在玉米种子鉴定上的应用。
一、SSR分子标记技术的基本原理SSR分子标记技术是基于DNA序列上短重复序列的多态性而开发的一种分子标记技术。
这些短重复序列通常为2-6个碱基的重复序列,如ATATAT、AGAGAG等。
在不同个体中,这些短重复序列的重复次数和排列方式不同,因此可以用作分子标记。
SSR分子标记技术的基本原理是:首先从待分析的DNA样品中提取出DNA,并使用PCR技术扩增出含有SSR位点的DNA片段。
然后,利用电泳技术将扩增出的DNA片段分离出来,并通过染色体特异性的显色剂进行染色。
最后,通过比较不同个体的DNA条带图谱,确定不同个体之间的遗传差异。
二、SSR分子标记技术在玉米种子鉴定中的应用SSR分子标记技术在玉米种子鉴定中的应用主要体现在以下几个方面:1.玉米品种的鉴定SSR分子标记技术可以通过比较不同玉米品种的DNA条带图谱,确定不同品种之间的遗传差异。
这种方法比传统的形态学鉴定方法更为准确和可靠。
2.杂交种子的鉴定杂交种子是由不同品种的玉米杂交而成的,因此杂交种子的遗传背景比较复杂。
使用SSR分子标记技术可以快速准确地鉴定杂交种子的亲本品种,有助于杂交育种的进展。
3.种子纯度的鉴定种子纯度是指种子中所含的杂质和其他品种的比例。
使用SSR分子标记技术可以准确地鉴定种子的纯度,有助于保证种子的品质和纯度。
4.种子存储的鉴定种子存储过程中,可能会发生一些突变和遗传变异,从而影响种子的品质和纯度。
使用SSR分子标记技术可以快速准确地鉴定种子存储过程中的遗传变异,有助于提高种子的品质和纯度。
三、SSR分子标记技术在玉米种子鉴定中的应用案例1.玉米品种的鉴定一项研究使用SSR分子标记技术对中国南方地区的20个玉米品种进行了鉴定。