蛋白质融合表达的标签及切割研究
- 格式:doc
- 大小:62.50 KB
- 文档页数:5


蛋⽩质表达中质粒上TEV酶切位点的设计与应⽤⼀、引⾔蛋⽩质表达是⽣物学研究中重要的技术⼿段之⼀,其中质粒作为外源基因的携带者,在蛋⽩质表达过程中发挥着关键作⽤。
TEV酶(Tobacco Etch Virus protease)作为⼀种常⽤的切割酶,被⼴泛应⽤于蛋⽩质表达系统中,能够在特定的氨基酸序列识别并切割蛋⽩质,为蛋⽩质纯化和功能研究提供了便利。
⼆、TEV酶切割位点的设计原则在质粒构建中,合理设计TEV酶切割位点⾄关重要。
设计TEV酶切割位点时应考虑以下原则:1. 特异性:选择在⽬标蛋⽩质序列中唯⼀存在的TEV酶切割位点,以避免⾮特异性切割导致的副产物。
2. 保留原⽣氨基酸序列:尽可能选择不影响⽬标蛋⽩质结构和功能的TEV酶切割位点,以确保切割后蛋⽩质仍能够正确折叠和发挥作⽤。
3. 免疫原性:避免选择可能引起免疫原性问题的TEV酶切割位点,以减少可能的影响。
三、TEV酶切割在蛋⽩质表达中的应⽤TEV酶切割在蛋⽩质表达中具有⼴泛的应⽤价值:1. 蛋⽩质纯化:TEV酶可以⾼效地切割融合标签,如His标签、GST标签等,从⽽实现对⽬标蛋⽩质的纯化。
2. 蛋⽩质亲和性纯化:利⽤TEV酶切割蛋⽩质与亲和树脂之间的连接,实现⾼纯度的蛋⽩质亲和纯化。
3. 结构研究:TEV酶切割可以⽤于去除蛋⽩质表达体系中的融合标签,以进⾏蛋⽩质结构和功能的研究。
4. 功能研究:通过TEV酶切割产⽣⽆标签的⽬标蛋⽩质,⽤于功能鉴定和活性分析。
四、实验步骤1. 质粒构建:在⽬标蛋⽩质的N-末端或C-末端引⼊包含TEV酶切割位点的多肽序列,如ENLYFQG。
2. 蛋⽩质表达:将质粒转化⾄表达宿主中,经过诱导表达⽬标蛋⽩质。
3. 蛋⽩质纯化:利⽤亲和树脂纯化体系,如Ni-NTA亲和层析,纯化蛋⽩质融合标签形式。
4. TEV酶切割:应⽤TEV酶切割蛋⽩质与融合标签之间的连接,获取⽆标签的⽬标蛋⽩质。
5. 切割后处理:通过适当的层析技术去除TEV酶和融合标签,获得纯净的⽬标蛋⽩质。

融合蛋白的作用与应用蛋白质是生命体中重要的组成部分,因此蛋白质的合成和研究一直是生物科学的热门领域。
在这个领域里,融合蛋白技术因其独特的作用和广泛的应用而备受关注。
本文章将重点介绍融合蛋白技术在生物科学研究中的作用和应用。
融合蛋白是一种人工合成的蛋白质,它是由两个或多个不同蛋白质的基因融合而成的。
融合蛋白通常由一个标签和一个目标蛋白组成。
标签可以是荧光蛋白、酶标签、His-标签等。
融合标签可以帮助研究人员追踪蛋白质的存在、位置和运动。
目标蛋白可以是任何感兴趣的蛋白质,例如转录因子、激酶、酶等。
融合蛋白技术的主要作用是实现表达和纯化目标蛋白质。
传统的表达和纯化方法通常需要在目标蛋白质中添加一些化学物质或结构,这样会影响到蛋白质的本来结构和功能。
而融合蛋白技术使得目标蛋白质可以在不添加任何结构和化学物质的情况下得到表达和纯化。
因此,融合蛋白技术可以保证目标蛋白质的结构和功能完整,从而更好的进行生物学研究。
融合蛋白技术还可以用于标记和可视化蛋白质,以便于对其进行追踪和定位。
这项技术可以帮助研究人员研究蛋白质在细胞内的运动和交互,并且提供一种高效、准确的方法来定位和证实蛋白质相互作用的位置和方式。
融合蛋白技术还可以用于对药物与蛋白相互作用进行研究,这样可以更好的理解药物的作用机理并开发更加高效、准确的药物。
例如,研究人员可以制备融合蛋白并将其融入药物。
然后,他们可以研究药物对融合蛋白的影响,从而了解药物如何与蛋白相互作用。
融合蛋白技术还可以用于优化蛋白表达和纯化方案。
蛋白表达和纯化通常是一项复杂的任务,但是融合蛋白技术可以使其更加容易。
通过使用融合蛋白来表达和纯化目标蛋白,可以使表达和纯化此目标蛋白变得更加容易和高效。
总而言之,融合蛋白技术在生物科学研究中具有广泛的应用,可以实现表达和纯化目标蛋白、标记和可视化蛋白、研究药物与蛋白相互作用和优化蛋白表达和纯化方案。
随着蛋白质研究的不断深入,融合蛋白技术将会发挥更多的作用和应用。

蛋白表达载体系统重组蛋白表达技术和重组蛋白表达载体现已广泛应用于生物学各个具体领域,尤其是体内功能研究和蛋白质的大规模生产。
GeneCopoeia的蛋白表达载体按照表达宿主的不同分为3类:B类,M类和Lv 类。
B类的表达宿主为大肠杆菌,M类的宿主为哺乳动物细胞,Lv类是慢病毒载体,宿主可以为哺乳动物细胞或原代细胞。
除了必要的复制和筛选的元件,协助表达和翻译的元件外,本文将各类载体按荧光蛋白标签、多功能标签、促溶解度标签和抗体免疫共沉淀标签四种,先将几种主流的标签功能初步介绍如下:eGFP/eCFP/eYFP/mCherry分别是增强型绿色荧光蛋白/增强型黄绿色荧光蛋白/增强型黄色荧光蛋白/单体红色荧光蛋白标签,具有不同的激发波长和发射波长,均由野生型荧光蛋白通过氨基酸突变和密码子优化而来。
就eGFP而言,相对于GFP,其荧光强度更强、荧光性质更稳定。
同时载体中构建的Kozak序列使得含有eGFP的融合蛋白在真核表达系统中表达效率更高。
mCherry是从DsRed演化来的性能最好的一个单体红色荧光蛋白,可以和GFP系列荧光蛋白共用,实现多色标记体内、外实验表明,mCherry在N端和C端融合外源蛋白时,荧光蛋白活性和被融合的目标蛋白功能相互没有明显影响。
这些荧光标签蛋白,其融合表达目的蛋白后具有以下优点:1.不用破碎组织细胞、不加任何底物,直接通过荧光显微镜就能在活细胞中发出绿色荧光,实时显示目的基因的表达情况,而且荧光性质稳定,被誉为活细胞探针。
2.其自发荧光,不需用目的基因的抗体或原位杂交技术就可推知目的基因在细胞中的定位等情况。
3.同时细胞内的其它产物不会干扰标签蛋白检测,从而使其检测效果更快速、简便、灵敏度高而且重现性好。
4.其低消耗、高灵敏度检测的特性,十分适用于高通量的药物筛选。
现eGFP 表达标签被广泛地应用于基团表达调控、转基因功能研究、蛋白在细胞中的功能定位、迁移变化及药物筛选等方面。

融合标签技术及其应用摘要:融合标签技术是一种基于报告基因的重组DNA技术,融合标签技术的发展使重组蛋白质的纯化、固定及检测更加快速、简便,并很快得到了应用。
关键词:融合标签技术;重组蛋白;应用融合标签技术是20世纪末兴起的一种基因重组技术,其主要过程是利用重组DNA技术在靶蛋白编码基因的3’端或5’端融合某种标签的编码基因,通过适宜的宿主来表达重组蛋白质[1],表达的重组蛋白质可以通过融合的标签与包被在固相基质上的特异配基结合而进行纯化。
融合标签技术的发展使重组蛋白质的纯化更加快速、简便。
近几年,随着新的融合标签系统的开发,其功能逐渐多样化,除用于蛋白质纯化外,还用于蛋白质定位和检测等。
1 融合标签的种类融合标签根据其分子量大小可分为两大类:大的蛋白质分子(或蛋白质结构域及其衍生物)和小的多肽片段。
大的蛋白质标签有GST(谷胱甘肽巯基转移酶)、SPA(葡萄球菌蛋白A)和GFP(绿色荧光蛋白)等,它的使用会增加目标蛋白的溶解性,但在蛋白结晶和抗体产生等过程中,标签必须去除。
小的多肽标签有6×His(六聚组氨酸)、HA(流感病毒血凝素表位)、c-myc(人c-myc蛋白表位)、FLAG(专为蛋白纯化和检测设计的八肽)等,多数情况下,由于多肽标签相对较小,对融合蛋白质结构影响小,不需要从融合蛋白质中切除,因而多肽标签较蛋白质标签更为常用。
迄今为止,已有文献较为详尽地报道了各种融合标签[2],其大小从几个氨基酸到蛋白质不等,相互作用类型包括酶与底物,细菌受体与血清蛋白,六聚组氨酸与金属离子,抗原与抗体等[3],表1-1列出了目前报道较多的标签融合系统。
2融合标签技术的应用2.1用于蛋白的纯化。
融合标签技术用于重组蛋白质纯化已为大量实验所证实[4]。
理论上,根据亲和纯化原理可定向设计使目标蛋白质与纯化基质之间不发生任何直接相互作用的融合标签,以避免由于这种直接相互作用造成蛋白质变性。
目前已有许多不同的标签可供利用。
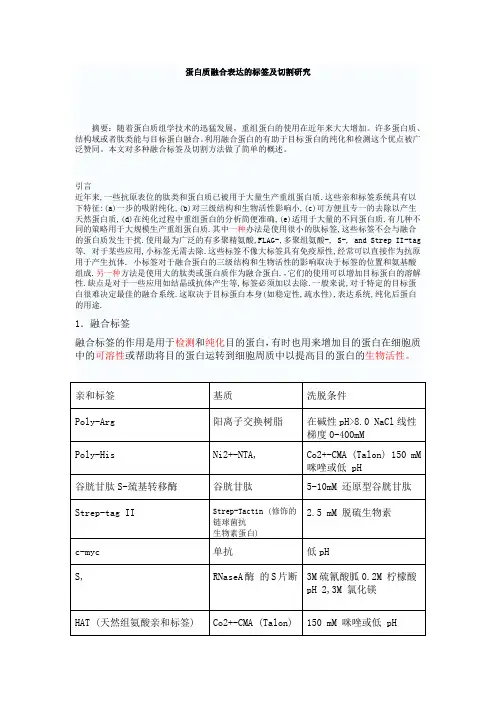
1 .1多聚精氨酸-标签(Arg-tag)精氨酸-标签通常由5或6个精氨酸组成.它已被成功用作细菌C末端标签,精氨酸是碱性最强的氨基酸,带5个精氨酸标签的蛋白质可以结合到阳离子交换树脂SP-Sephadex上, 而大部分杂蛋白不结合.结合后,带标签的蛋白质在碱性pH下运行线性NaCl梯度洗脱得到.当C末端为疏水性区域时,多聚精氨酸可能影响蛋白质的三级结构.氨酸残基的C末端序列可用羧肽酶B处理去除.这一酶促处理已被成功用于一些例子,但常常由于低的切割得率或者在期望的蛋白质序列间发生不必要的切割而受到限制.然而精氨酸标签并不常用,与第二标签联用是很有趣的蛋白质纯化工具.1.2 多聚组氨酸-标签(His-tag)已广泛采用的方法是利用固定化金属螯合层析纯化带有由多聚组氨酸残基组成的一个短的亲和标签的重组蛋白质.固定化金属螯合层析的基础是固定在基质上的过渡态金属离子(Co2+, Ni2+, Cu2+, Zn2+)与特定的氨基酸侧链之间的相互作用.组氨酸是与固定化金属离子作用最强的氨基酸,组氨酸的咪唑环作为电子供体容易与固定的金属离子形成配位键.基质材料洗涤之后,可通过调节柱缓冲液的pH或者添加游离咪唑洗脱含多聚氨基酸序列的肽类。
虽然在变性条件下系统对于带6个组氨酸标签的蛋白质纯化有效,带3个组氨酸标签的蛋白质在生理条件下可有效纯化.然而带6个组氨酸标签的蛋白质可以在天然条件下,在低或高浓度盐的缓冲液中结合到Ni2+-NTA 基质上.结合后,目标蛋白可用0.8到250 mM咪唑梯度洗脱.低浓度的咪唑(如0.8 mM)可减少带有组氨酸的宿主蛋白质的非专一吸附.6个组氨酸标签的蛋白质可用20到250mM咪唑浓度范围洗脱. 使用咪唑的缺点是咪唑的存在经常导致蛋白质的聚集.另一种用于纯化多聚组氨酸标签蛋白质的材料是TALON.这由Co2+-羧甲基天冬氨酸(Co2+-CMA)组成,并偶联到固相的树脂上.TALON使得标签蛋白在温和条件下洗脱,已有报导与Ni2+-NTA树脂相比,其非特异性蛋白结合更少,从而达到更高的洗脱产物纯度.一种酶制剂通过SDS-PAGE鉴定的纯度高于95%.1.3 谷胱甘肽S-转移酶-标签1988年首次描述了融合有谷胱甘肽S-转移酶(GST)的多肽的一步纯化.来自日本血吸虫(schisto soma japonicum),26-kDa的 GST被克隆在大肠杆菌表达载体中.融合蛋白可从未经处理的裂解液中用固定化的谷胱甘肽亲和层析加以纯化.结合的融合蛋白在非变性条件下用10mM 还原型谷胱甘肽洗脱..GST标签可用酶学分析或免疫分析很方便的检测.标签有助于保护重组蛋白免受胞外蛋白酶的降解并提高其稳定性.在大多数情况下GST融合蛋白是完全或部分可溶的.推荐采用位点专一的蛋白酶如凝血酶或Xa因子从融合蛋白切除GST标签.蛋白酶解后,GST载体和蛋白酶通过谷胱甘肽琼脂糖亲和层析去除.GST标签可位于N端或C端,可用于细菌,酵母,哺乳细胞, 和杆状病毒感染的昆虫细胞. GST融合蛋白已成为分子生物学家的基本工具.1.4 Strep-标签(Strep-tag)Strep-标签是一氨基酸肽开发来用于在链球菌抗生物素蛋白(streptavidin)柱上亲和纯化相应的融合蛋白.链球菌抗生物素蛋白(streptavidin)在特定位点第44, 45, 和47位的突变体与天然形式相比,对八肽Strep-tag II的亲和力更强,.Strep标签的蛋白质在生理缓冲液条件下结合到生物素结合区域中,并可用生物素衍生物温和洗脱.洗脱时推荐使用2.5 mM脱硫生物素.基质可以用4-羟基-偶氮苯-2-羧酸再生,这种物质在溶液中呈黄色,当结合到基质上则呈红色.结合条件是非常专一的.生物素化的蛋白质如大肠杆菌的羧基载体蛋白可以结合,但生物素或者生物素化的蛋白质则用抗生物素封闭.纯化条件是多种多样的.螯合剂,温和性去污剂,还原型去污剂,和高达1M的盐可以加到缓冲液中. Strep-标签系统适合用于研究蛋白质之间的相互作用以及当那些大的标签或者带电荷的标签无效时的特殊用途.1.5 S-标签S-标签是个融合肽标签可以通过快速灵敏的均匀分析或者在Western blot中用比色法加以检测.该系统的基础是15个氨基酸残基的S-tag与103氨基酸残基的S-蛋白质之间的强烈相互作用,这两个都来自RNaseA.S-蛋白质/S-标签复合物的Kd接近0.1μM,这取决于pH,温度和离子强度.标签由4个带正电荷残基,3个负电荷残基,3个不带电的极性残基和5个非极性残基组成.这使S -标签保持可溶.S-标签的快速分析是基于核酸降解活力的恢复.标签蛋白可以结合到S蛋白质基质上.洗脱条件较苛刻,如pH2.0的缓冲液.然而为了获得有功能的蛋白质推荐用蛋白酶切割标签.该系统可用于纯化的重组蛋白质来源包括细菌,哺乳细胞和杆状病毒感染的昆虫细胞抽提物.该系统经常与第二标签联用.RNase酶A高度灵敏的荧光底物的发现使得该系统可用于与高通量筛选联用的检测.2. 融合蛋白的切割为了对目的蛋白进行生化及功能分析,通常要从目的蛋白上去除fusion tag部分。

融合蛋白的分离纯化原理融合蛋白是指在外源表达系统中,将感兴趣的蛋白与另一个蛋白(通常具有较好的抗体或标签)融合在一起,以便在后续的蛋白分离和纯化过程中更容易识别和纯化。
以下是由融合蛋白的分离纯化原理所组成的详细解释。
1. 蛋白融合表达系统的构建和优势:融合蛋白的表达系统通常包括重组蛋白的DNA序列,它融合了目标蛋白和另一种中间蛋白(常被称为融合标签)。
通过将DNA序列导入到合适的宿主细胞中,在细胞中进行表达和产生融合蛋白。
融合蛋白具有以下优势:- 提高目标蛋白的产量和稳定性;- 改善目标蛋白的溶解和可溶性;- 方便蛋白纯化和检测。
2. 常见的融合标签:常见的融合标签包括:- 多希(histidine tag):这是最常用的标签,由6个连续的组氨酸残基组成。
利用多希标签可以简便地将融合蛋白通过亲和层析柱进行结合和纯化,例如金属螯合层析;- 谷氨酸蛋白酶切位点(glutathione S-transferase cleavage site, GST tag):GST标签可以用于纯化融合蛋白,它可以通过谷氨酸蛋白酶进行特异性切割,从而分离目标蛋白;- 绿色荧光蛋白(green fluorescent protein, GFP tag):GFP标签可以通过荧光显微镜直接检测融合蛋白,从而评估目标蛋白的表达和定位情况。
3. 整细胞和亲和层析柱的纯化方法:对于融合蛋白的纯化,可以采用整细胞和亲和层析柱的方法。
整细胞的纯化方法是将目标融合蛋白在细胞中产生,并通过裂解细胞膜和核膜释放蛋白。
然后,通过高速离心将裂解物中的纤维蛋白、DNA和RNA沉淀,获得上清液。
接下来,可以通过盐析沉淀、超滤或浓缩来富集目标蛋白。
亲和层析是利用融合蛋白与特定配体的结合来纯化蛋白。
例如,使用多希标签可以通过金属螯合层析柱选择性地结合多希蛋白,并通过改变洗脱缓冲液的组成来分离并收集目标蛋白。
4. 蛋白纯化的后续步骤:蛋白纯化的后续步骤通常包括:- 目标蛋白的浓缩和洗脱:通过离心、超滤或吸附到凝胶等方法去除污染物,从而富集目标蛋白;- 蛋白质的测定和定性:通过比色法、荧光法或质谱等方法确定目标蛋白的含量和纯度;- 蛋白质的保存和储存:通过冻干、冷冻或加入保护剂等方法保持蛋白的活性和稳定性。


重组蛋白融合标签及对应的酶切酶一、概述重组蛋白是一种人工合成的蛋白质,通常用于生物技术和药物研发领域。
在研究和应用中,为了更好地研究和分析重组蛋白,通常需要在其N端或C端连接不同的标签,以便于对其进行纯化、检测和定位等操作。
而对于大多数重组蛋白而言,酶切酶是一个重要的工具,它可以通过切割特定的肽键来去除标签或实现蛋白质的精确裁剪。
重组蛋白融合标签及对应的酶切酶是重要的研究话题。
二、重组蛋白融合标签1. His标签His标签是一种常用的重组蛋白融合标签,它通常由6个组氨酸构成,其序列为His-His-His-His-His-His。
这种标签能够与金属离子亲和层析柱上的镍离子结合,便于对融合蛋白进行纯化。
常用的His标签融合蛋白包括His-tag、GST-His-tag等。
对应的酶切酶包括Thrombin、Enterokinase等,可以在需要的时候精确切割掉His标签。
2. FLAG标签FLAG标签是另一种常用的重组蛋白融合标签,它是一个八肽序列,其序列为DYKDDDDK。
FLAG标签常用于蛋白质检测和免疫印迹等分析中。
常用的FLAG标签融合蛋白包括pET21a-FLAG、CMV-FLAG等。
对应的酶切酶为Enterokinase等,可实现对FLAG标签的精确切割。
3. GFP标签GFP标签是一种表达在绿色荧光蛋白上的重组蛋白融合标签,其序列与GFP蛋白的序列相同,能够使融合蛋白在荧光显微镜下直接观察到。
常用的GFP标签融合蛋白包括pGFP、pGFP-V-RS等。
对应的酶切酶为Bgl II等。
4. HA标签HA标签是一种可溶酶敏感的蛋白质标签,其序列为YPYDVPDYA。
HA标签常用于蛋白质的免疫检测、纯化等。
对应的酶切酶为Enterokinase等。
5. Myc标签Myc标签是另一种常用的重组蛋白融合标签,其序列为EQKLISEEDL。
Myc标签常用于免疫印迹、免疫沉淀等研究中。
对应的酶切酶为Enterokinase等。

融合标签技术在蛋白质研究中的应用摘要近年来,越来越多的科学家开始运用一种被称为融合标签的技术来研究蛋白质。
融合标签技术的快速发展给蛋白的快速纯化带来了福音。
本文主要介绍一些常用融合标签的原理、优缺点以及它们在蛋白质研究中的应用,并讨论了融合标签对蛋白研究产生的深远影响。
关键词蛋白质融合标签蛋白纯化前言蛋白质相互作用存在于每个细胞的新陈代谢过程中。
围绕着中心法则,我们可以发现,DNA复制、转录、逆转录、翻译等少不了蛋白质的参与,而且在细胞的信号转导,细胞死亡和凋亡等生命活动中,蛋白质的相互作用也都发挥了核心作用。
对蛋白质的研究,催生了结构生物学的蓬勃发展,而且,蛋白质相互作用的研究也成为了蛋白质研究不可或缺的一部分[1]。
利用蛋白质表达技术,快速、简便地进行有关活性代谢产物的高效表达而研究蛋白质生物学功能和理化性质成为一种很好的研究方法。
其中融合蛋白表达技术由于容易实现高效表达、纯化,兼有步骤简便、产物多以活性形式存在等优点,在蛋白质研究中得到了广泛的应用[2]。
蛋白融合即在蛋白序列上加上蛋白标签。
蛋白标签是指利用DNA重组技术与目的蛋白一起融合表达的一种多肽或蛋白质,以便于目的蛋白的表达、检测以及纯化[3]。
该技术的原理是通过基因操作技术,将目的基因片段与某些特定的蛋白标签串联,插入适当的载体中进行表达。
这些特定的基因因为带有标签,能够与特殊的介质进行亲和层析,使特定的蛋白能够纯化出来。
得到的融合蛋白还可以通过使用化学物质或者酶类的特定反应切除标签,从而得到纯化的目的蛋白,用于结晶或者特定功能研究。
常用的融合蛋白主要包括多聚组氨酸标记、硫氧还原蛋白、谷胱甘肽S-硫转移酶(GST)、融合了八个氨基酸的亲水性多肽、麦芽糖结合蛋白等。
根据其分子量大小,可将之分为两类:小的多肽标签,包括FLAG、His6(六聚组氨酸)、c-myc(人c-myc蛋白表位)、HA(流感病毒血凝素表面抗原决定簇)等;大的蛋白标签,包括SPA(葡萄球菌蛋白A)、GST(谷胱甘肽巯基转移酶)和GFP (绿色荧光蛋白)等。

在蛋白质功能及结构研究过程中,研究的首要任务就是利用多种方法获得纯化的具有完整结构及生物学功能并能够正确折叠的高度纯化蛋白质。
除了蛋白质的研究,具有特定生物活性的高价值蛋白质的生产也需要对蛋白质产品进行纯化。
因此,科研人员和工业生产往往大量采用多种多样的表达系统来获得高表达的蛋白质,对其纯化后进行研究或加工,这些表达系统包括原核表达系统、酵母菌表达系统、昆虫动物细胞表达系统、真核细胞表达系统等。
如何将系统中表达的目标蛋白质与其他蛋白分离,一直是表达纯化中的一个重点。
为了克服从复杂样品中纯化单一蛋白质这种困难,科学家利用生物物质,特别是酶和抗体等蛋白质,具有识别某种特定物质并与该物质分子特异性结合的能力,利用生物分子间的这种特异性结合能力而形成的亲和纯化技术。
亲和标签纯化技术已广泛应用于蛋白质,特别是重组蛋白的分离纯化中。
在重组蛋白的亲和纯化中,利用基因工程技术,将经过改造优化的亲和标签与目标蛋白融合表达,通过一步简单快速的亲和层析,直接获得纯度较高的重组融合蛋白,已成为重组蛋白纯化的一个通用方法,具有结合特异性高、纯化步骤简便、纯化条件温和、适用性广泛等优点,为蛋白质的有效纯化提供了一条解决的途径,广泛应用于蛋白质结构与功能的研究及重组蛋白纯化工艺的开发中。
自从20世纪70年代中期融合标签技术出现以来,亲和标签已成为一种重组蛋白纯化十分有效的工具,具有结合特异性高、纯化条件温和、纯化步骤简便、适用性广泛等显着优势。
通常,亲和标签定义为对特定的生物或化学配基具有高度亲和力的一段氨基酸序列。
到目前为止,已经出现了种类众多、功能各异、用途多样的亲和标签,极大地促进了对重组蛋白的有效纯化。
根据自身分子量大小的不同,亲和标签可以分为两大类:一类是结合固定化配基的短肽标签,如His-tag、FLAG-tag、Strep-tagⅡ等;另一类是识别小分子配基的蛋白标签,如GST、MBP等。
短肽标签:His-tag:His标签是目前高通量蛋白纯化最普遍使用的亲和标签,广泛用于多种重组蛋白在各种表达系统的表达与纯化中。
蛋白融合pdi标签的用途蛋白融合PDI标签的用途蛋白质折叠是生物学中一个重要的过程,它指的是蛋白质从线性氨基酸序列折叠成特定的三维结构的过程。
这个过程对于蛋白质功能的发挥非常重要,因为只有在正确的三维结构下,蛋白质才能发挥其生物学功能。
然而,蛋白质折叠的过程并不总是顺利进行,有时会出现错误的折叠或聚集,导致蛋白质失去功能或形成有害的聚集物。
为了解决这个问题,科学家们开发了一种蛋白质标签——蛋白质融合PDI标签,用于监测和促进蛋白质折叠的正确进行。
蛋白质融合PDI标签是一种蛋白质融合标签,其中PDI代表蛋白质二硫醇异构酶(protein disulfide isomerase)。
PDI是一种在内质网(endoplasmic reticulum,ER)中广泛存在的蛋白质,它在蛋白质折叠的过程中起着重要的作用。
蛋白质融合PDI标签利用了PDI的功能,将其与目标蛋白质融合,以监测和促进目标蛋白质的正确折叠。
蛋白质融合PDI标签的主要用途之一是在蛋白质表达和纯化过程中监测蛋白质折叠的质量。
蛋白质的折叠质量对于蛋白质的功能和稳定性至关重要,因此在表达和纯化过程中对折叠质量进行监测是必要的。
通过将PDI标签与目标蛋白质融合,可以利用PDI的二硫醇异构酶活性来检测目标蛋白质的折叠状态。
如果目标蛋白质折叠正确,PDI标签将保持在目标蛋白质上;而如果目标蛋白质折叠出现错误,PDI标签将与目标蛋白质分离。
通过监测PDI标签的状态,可以及时发现蛋白质折叠的问题,并采取相应的措施进行修复或改进。
除了在蛋白质表达和纯化中的应用,蛋白质融合PDI标签还可以用于研究蛋白质折叠的机制和调控。
蛋白质折叠是一个复杂的过程,涉及到许多分子和细胞机制的相互作用。
通过将PDI标签与不同的蛋白质融合,可以研究PDI在折叠过程中的作用和调控机制。
此外,还可以利用PDI标签来筛选和鉴定与蛋白质折叠有关的分子和药物,以及探索蛋白质折叠与疾病之间的关联。
在蛋白质功能及结构研究过程中,研究的首要任务就是利用多种方法获得纯化的具有完整结构及生物学功能并能够正确折叠的高度纯化蛋白质。
除了蛋白质的研究,具有特定生物活性的高价值蛋白质的生产也需要对蛋白质产品进行纯化。
因此,科研人员和工业生产往往大量采用多种多样的表达系统来获得高表达的蛋白质,对其纯化后进行研究或加工,这些表达系统包括原核表达系统、酵母菌表达系统、昆虫动物细胞表达系统、真核细胞表达系统等。
如何将系统中表达的目标蛋白质与其他蛋白分离,一直是表达纯化中的一个重点。
为了克服从复杂样品中纯化单一蛋白质这种困难,科学家利用生物物质,特别是酶和抗体等蛋白质,具有识别某种特定物质并与该物质分子特异性结合的能力,利用生物分子间的这种特异性结合能力而形成的亲和纯化技术。
亲和标签纯化技术已广泛应用于蛋白质,特别是重组蛋白的分离纯化中。
在重组蛋白的亲和纯化中,利用基因工程技术,将经过改造优化的亲和标签与目标蛋白融合表达,通过一步简单快速的亲和层析,直接获得纯度较高的重组融合蛋白,已成为重组蛋白纯化的一个通用方法,具有结合特异性高、纯化步骤简便、纯化条件温和、适用性广泛等优点,为蛋白质的有效纯化提供了一条解决的途径,广泛应用于蛋白质结构与功能的研究及重组蛋白纯化工艺的开发中。
自从20世纪70年代中期融合标签技术出现以来,亲和标签已成为一种重组蛋白纯化十分有效的工具,具有结合特异性高、纯化条件温和、纯化步骤简便、适用性广泛等显著优势。
通常,亲和标签定义为对特定的生物或化学配基具有高度亲和力的一段氨基酸序列。
到目前为止,已经出现了种类众多、功能各异、用途多样的亲和标签,极大地促进了对重组蛋白的有效纯化。
根据自身分子量大小的不同,亲和标签可以分为两大类:一类是结合固定化配基的短肽标签,如His-tag、FLAG-tag、Strep-tagⅡ等;另一类是识别小分子配基的蛋白标签,如GST、MBP等。
短肽标签:His-tag:His标签是目前高通量蛋白纯化最普遍使用的亲和标签,广泛用于多种重组蛋白在各种表达系统的表达与纯化中。
GST-NAP融合蛋白可溶性表达及柱上切割GST标签黄夏冰;康巧珍;傅国;汲振余;刘鑫【摘要】利用基因工程的方法,以实验室保存的pMAL-C2X-NAP质粒为模板,PCR 扩增NAP基因,构建重组质粒pGEX-NAP.重组质粒通过酶切和测序鉴定后转化大肠杆菌表达菌株BL21(DE3),再经IPTG低温诱导获得可溶性GST-NAP融合蛋白,最后利用谷胱甘肽琼脂糖凝胶树脂进行纯化,Prescission蛋白酶进行柱上切割去除GST标签.结果表明,pGEX-NAP重组质粒构建正确,在大肠杆菌中经IPTG低温诱导表达,可获得大量可溶性GST-NAP融合蛋白. Prescission蛋白酶柱上切割去除GST标签后,经Western Blot 验证NAP蛋白能被兔抗NAP多克隆抗体特异识别.%To obtain plenty of HP-NAP ( H. pylori neutrophil-activating protein) for research, NAP gene was amplified from pMAL-C2X-NAP vector by PCR, and the recombinant plasmid pGEX-NAP was con-structed. After verified by enzyme digestion and gene sequencing analysis, pGEX-NAP was transformed into E. coli BL21(DE3)and the soluble fusion protein GST-NAP was obtained by inducing with IPTG in low temperature. Then GST-NAP was purified with Glutathione Sefinose Resin, and cleavaged GST-tag with Prescission protease on column. In conclusion, recombinant plasmid pGEX-NAP was successfully constructed and soluble GST-NAP fusion protein was efficiently expressed in E. coli. High purity HP-NAP can be obtained by Prescission protease cleavage on column. NAP protein was recognized by Rabbit anti-NAP mAb using Western Blot.【期刊名称】《郑州大学学报(理学版)》【年(卷),期】2015(000)004【总页数】5页(P94-98)【关键词】GST-NAP;重组质粒;蛋白表达;GST标签【作者】黄夏冰;康巧珍;傅国;汲振余;刘鑫【作者单位】郑州大学生命科学学院分子免疫学实验室河南郑州450000;郑州大学生命科学学院分子免疫学实验室河南郑州450000;郑州大学生命科学学院分子免疫学实验室河南郑州450000;河南省医药科学研究院河南郑州45000;郑州大学生命科学学院分子免疫学实验室河南郑州450000【正文语种】中文【中图分类】Q786HP-NAP是幽门螺杆菌中的一种能趋化并激活中性粒细胞的可溶性蛋白,它能促进中性粒细胞黏附胃黏膜内皮,并且能够刺激中性粒细胞产生大量的氧自由基.到目前为止,HP-NAP被证实是Dps(DNA protection during starvation)家族中唯一能够激活人类白细胞的蛋白,和HP-NAP结构相似的Flp、Dlp-1和Dlp-2都没有此功能.与其他结构相似的分子比较,HP-NAP具有独特阳离子电荷簇,这可能有利于炎症细胞的激活.已有报道,HP-NAP具有通过逆转Th1/Th2平衡,抗旋毛虫感染,抗哮喘及治疗肿瘤的潜力[1-8],同时也被认为是极具潜力的侯选免疫调节剂[9].HP-NAP可从幽门螺杆菌(Helicobacter pylori,H.pylori)水抽提物中获得,然而由于H.pylori培养条件较为苛刻,无法进行H.pylori的大规模培养,且体外条件下不同菌株HP-NAP表达量差异较大,因此,大量HP-NAP的稳定获得受到较大限制[10].目前,利用基因工程的方法重组表达目的基因,能够快速简便得到大量蛋白[11].E.coli作为基因工程菌, 具有生产周期短、成本低、易线性放大等优点,且该工程菌质粒上携带有多种融合标签,有利于目标蛋白的表达纯化[12].作为E.coli原核表达系统融合标签,谷胱甘肽-S-转移酶(GST)能够增加外源蛋白的可溶性[13-16].pGEX系列载体是高效表达GST的原核表达载体,其中pGEX-6P-1带有IPTG诱导启动子, 融合蛋白产物带有GST标签,因此可通过GST层析柱简便、快速地纯化,最终可以通过Prescission蛋白酶切割GST标签快速释放目的蛋白. 本研究构建了HP-NAP的原核表达质粒pGEX-NAP, 通过诱导表达、纯化得到大量的GST-NAP,经柱上切割成功去除GST标签,得到的NAP蛋白经Western Blot验证可被兔抗NAP多克隆抗体特异识别.本研究为进一步研究HP-NAP的功能奠定基础.1.1 材料1.1.1 质粒与菌株 pMAL-C2X-NAP质粒及pGEX-6P-1质粒均为本实验室保存;E.coli DH5α、BL21(DE3)感受态购自于郑州鼎国生物有限公司.1.1.2 试剂 Taq DNA聚合酶、T4 DNA连接酶、限制性内切酶EcoRⅠ、SalⅠ、质粒DNA小量提取试剂盒、DNA marker购自于宝生物工程(大连)有限公司;还原型谷胱甘肽、谷胱甘肽琼脂糖树脂购自于上海生工生物工程公司;低相对分子质量蛋白marker购自于北京全式金有限公司;异丙基硫代半乳糖苷(IPTG)、丙烯酰胺、甲叉丙烯酰胺等购自于郑州鼎国生物有限公司.1.2 方法1.2.1 pGEX-NAP重组质粒的构建首先,以本实验室构建并保存的pMAL-C2X-NAP质粒为模板扩增出HP-NAP基因,扩增HP-NAP基因的所用引物为:F: 5′-GGAGAATTCATGAAAACATTTGAAATTTTA-3′(含有EcoRⅠ酶切位点),R: 5′-AAGTCGACTTAAGCTAAATGGGCTTGCAGC-3′(含有SalⅠ酶切位点).然后将扩增得到的HP-NAP片段和空质粒载体pGEX-6P-1用EcoRⅠ、SalⅠ双酶切,酶切产物胶回收后用T4 DNA连接酶于16 ℃过夜连接.最后将连接产物转化到DH5a感受态细胞中,取少量菌液涂布到含氨苄的LB固体培养基上,37 ℃培养16 h后挑取单菌落,接种于含氨苄的LB液体培养基中,37 ℃振荡培养过夜,次日提取质粒.经EcoRⅠ、SalⅠ双酶切电泳验证,结果正确的质粒送去测序.1.2.2 GST-NAP融合蛋白的诱导表达将重组质粒pGEX-NAP转化到BL21感受态细胞中,37 ℃倒置培养过夜.次日挑取单菌落,接种于5 mL LB培养基中(含Amp 100 μg/mL),37 ℃,200 rpm活化过夜.然后按1%比例接种于500 mL LB培养基中(含A mp 100 μg/mL),37 ℃,230 rpm扩大培养3 h.最后加入诱导剂IPTG 至终浓度0.5 mmol/L,15 ℃过夜培养.取200 μL菌液,离心弃上清,SDS-PAGE电泳分析.1.2.3 GST-NAP融合蛋白的纯化将诱导表达后的菌液用50 mL离心管分装,4 000 rpm离心5 min,弃上清.用5 mL GST binding buffer(含有1% Triton X-100的PBS)重悬沉淀,-20 ℃冻存过夜.室温解冻后用40 mL GST binding buffer重悬,冰浴条件下使用300 W短脉冲超声处理(超声处理10 s,间隔15 s)重复20~30次至菌液透明.将超声破碎后的菌体于4 ℃、9 000 rpm离心20 min,取上清置于另一无菌的50 mL离心管中,沉淀用5 mL GST binding buffer重悬,分别对超声上清和沉淀物进行可溶性及超声破碎效果分析.取1 mL谷胱甘肽琼脂糖树脂加入到3 mL层析柱中,制备简易重力亲和层析柱.使用10 mL GST binding buffer平衡层析柱,加入菌体上清25 mL,经10 mL GST binding buffer洗涤后,使用12 mL Elution buffer(10 mmol/L还原型谷胱甘肽,50 mmol/L Tris-HCl,pH 8.0)洗脱得到GST-NAP,并用PBS进行透析.纯化后的蛋白通过SDS-PAGE验证.1.2.4 Prescission protease柱上切割GST-NAP融合蛋白在1 mL层析柱中加入400 μL GST树脂,将纯化透析后的GST-NAP按照纯化步骤进行平衡、上样、洗涤.配制酶切反应体系(PP酶10 μL,10×cleavage buffer 50 μL,ddH2O 440μL)加入到层析柱中,4 ℃反应16 h后,分多管收集流出液,并用Elution buffer 洗脱蛋白.流出液及洗脱蛋白通过SDS-PAGE验证.1.2.5 Western Blot鉴定NAP蛋白将流出液经SDS-PAGE分离后,在110 V、65 min条件下转至硝酸纤维膜(NC)上,用1%BSA室温封闭1.5 h;使用TBST(含0.9% NaCl的10 mmol/L Tris溶液加入适量Tween-20,PH 7.4)以1∶8 000稀释的兔抗NAP抗体常温孵育2 h后,TBST洗膜3次,每次10 min;再用TBST以1∶8 000稀释的羊抗兔IgG常温孵育1 h,TBST洗膜3次,每次10 min;最后进行增强化学发光(ECL)显色反应曝光.2.1 HP-NAP基因的扩增以pMAL-C2X-NAP质粒为模板,使用带有酶切位点的引物进行PCR扩增,得到了含有EcoRⅠ和SalⅠ酶切位点的435 bp片段,与目标条带大小一致(图1).2.2 重组质粒pGEX-NAP的酶切鉴定利用EcoRⅠ和SalⅠ酶切位点将HP-NAP片段克隆到pGEX-6P-1质粒中,转化到DH5α感受态细胞,挑取阳性克隆菌落,摇菌提取质粒,用SalⅠ和EcoR I双酶切进行鉴定,如图2所示结果.经双酶切后得到两条条带,分别为5 000 bp(载体pGEX-6P-1)和435 bp(目的基因NAP).将重组质粒测序并在Genbank数据库中使用同源性分析软件BLAST比对测序结果,目的片段序列与数据库中的HP-NAP基因序列完全一致.2.3 GST/NAP融合蛋白的表达、鉴定及可溶性分析构建好的重组质粒pGEX-NAP转化BL21,挑取单菌落,扩培后提取质粒,用NAP特异性引物进行PCR鉴定.对于鉴定正确的菌株,用IPTG对菌株进行15 ℃低温诱导表达,收集菌体进行超声破碎,SDS-PAGE结果显示在43 kD处可见明显的蛋白条带,其与目标蛋白GST-NAP融合蛋白大小一致(图3).同时,菌体经超声破碎后离心,对其沉淀物和上清进行SDS-PAGE,在43 kD处均出现条带,且在同等上样条件下,上清中的目标蛋白条带更为明显.结果显示,在15 ℃诱导条件下,GST-NAP融合蛋白主要以可溶的形式存在,如图4所示.2.4 GST-NAP融合蛋白纯化取1 mL谷胱甘肽琼脂糖树脂加入到3 mL层析柱中,制备简易重力亲和层析柱.使用10 mL GST binding buffer平衡层析柱,加入2.3中制备的菌体上清25 mL,经10 mL GST binding buffer洗涤后,使用12 mL洗脱液洗脱.对洗脱液进行SDS-PAGE鉴定,显示在43 kD处有唯一一条蛋白条带(图5).结果表明,经亲和层析得到了纯化的GST-NAP融合蛋白.2.5 Prescission protease柱上切割GST-NAP融合蛋白在1 mL层析柱中加入400 μL树脂,按照纯化步骤进行平衡、上样(纯化透析后的GST-NAP)、洗涤,然后加入PP酶切反应体系,4 ℃反应16 h后,分多管收集流出液,使用Elution buffer洗脱蛋白.流出液及洗脱蛋白经SDS-PAGE验证,17 kD的NAP单体存在于流出液中,洗脱蛋白为26 kD的GST标签(图6).2.6 Western Blot 鉴定NAP蛋白将流出液经SDS-PAGE分离后,进行转膜、封闭、孵育一抗、洗膜、孵育二抗、洗膜、ECL曝光处理得到Western Blot结果.兔抗NAP抗体与流出液中的NAP蛋白特异性结合,表明PP酶柱上切割成功获得NAP蛋白(图7).由于目前大量获取HP-NAP较为困难,不利于研究应用.因而,本研究尝试用简便、高效的大肠杆菌表达载体pGEX-6P-1构建重组载体pGEX-NAP,通过诱导表达、纯化得到大量的GST-NAP,经Prescission蛋白酶柱上切割GST标签后,成功得到HP-NAP.在诱导蛋白表达过程中,当使用37 ℃诱导表达时,在0.1 mmol/L,0.2 mmol/L,0.3 mmol/L,0.4 mmol/L和0.5 mmol/L IPTG浓度下, GST-NAP融合蛋白均为不可溶的包涵体蛋白.为了避免后续的变复性工作,尝试了低温诱导,当诱导温度降至15 ℃时诱导产物绝大部分为可溶的形式,并且在0.5 mmol/L IPTG条件下可溶性蛋白表达量最高.随后对纯化后的GST-NAP用Prescission蛋白酶进行柱上切割去除GST标签.Prescission蛋白酶能够识别pGEX-6P-1载体表达的蛋白序列Leu-Glu-Val-Leu-Phe-Gln-Gly-Pro,并且特异性地在Glu和Gly残基之间进行切割[17].Prescission蛋白酶在4 ℃具有最大活性,因此选择在低温下进行切割.最初尝试对结合在1 mL树脂上的GST-NAP进行直接切割,但并未成功,推断可能是由于酶切体系较小导致柱上切割不理想.随后尝试用小体积的树脂和大于树脂体积的酶切体系进行切割,最终实现GST-NAP的柱上切割.本研究提供了一种简便快速获取HP-NAP的方法,HP-NAP蛋白的表达纯化成功将为下一步研究提供重要的实验材料.[1] Satin B,Del P G,Della Bianca V,et al.The neutrophil-activating protein (HP-NAP) of Helicobacter pylori is a protective antigen and a majorvirulence factor [J].The Journal of Experimental Medicine,2000,191(9): 1467-1476.[2] Amedei A,Cappon A,Codolo G,et al.The neutrophil-activating protein of Helicobacter pylori promotes Th1 immune responses [J].The Journal of Clinical Investigation,2006,116(4): 1092-1101.[3] Del P G,Chiumiento L,Amedei A,et al.Immunosuppression of Th2 responses in Trichinella spiralis infection by Helicobacter pylori neutrophil-activating protein [J].The Journal of Allergy and Clinical Immunology,2008,122(5): 908-913.[4] Amedei A,Codolo G,Del P G,et al.The effect of Helicobacter pylorion asthma and allergy [J].Journal of Asthma and Allergy,2010,3: 139-147.[5] de Bernard M,D′Elios M M.The immune modulating activity of the Helicobacter pylori HP-NAP: Friend or foe? [J].Toxicon: Official Journal of the International Society on Toxinology,2010,56(7): 1186-1192.[6] Codolo G,Mazzi P,Amedei A,et al.The neutrophil-activating protein of Helicobacter pylori down-modulates Th2 inflammation in ovalbumin-induced allergic asthma[J].Cellular Microbiology,2008,10(11): 2355-2363.[7] Codolo G,Fassan M,Munari F,et al.HP-NAP inhibits the growth of bladder cancer in mice by activating a cytotoxic Th1 response [J].Cancer Immunology,Immunotherapy: CII,2012,61(1): 31-40.[8] Ramachandran M,Yu Di,Wanders A,et al.An infection-enhanced oncolytic adenovirus secreting H.pylori neutrophil-activating protein with therapeutic effects on neuroendocrine tumors [J].Molecular Therapy: the Journal of the American Society of Gene Therapy,2013,21(11): 2008-2018.[9] Fu Huawen.Helicobacter pylori neutrophil-activating protein: From molecular pathogenesis to clinical applications [J].World Journal of Gastroenterology,2014,20(18): 5294-5301.[10] 张帅清.如何提高幽门螺杆菌培养阳性率[J].中国卫生检验杂志,2007,17(4):759-760.[11] 李莉,贾新成,秦广雍,等.酵母海藻糖合成酶基因植物表达载体的构建[J].郑州大学学报:理学版,2003,35(1): 49-51.[12] 吴玉荣,吴海涛,曹萌,等.重组PET30a/hsBLyS质粒在BL21(DE3)菌株中的稳定性观察[J].信阳师范学院学报:自然科学版,2005,18(2):165-168.[13] Smith D B,Johnson K S.Single-step purification of polypeptides expressed in Escherichia coli as fusions with glutathione S-transferase [J].Gene,1988,67(1): 31-40.[14] 段沁江.谷胱甘肽S-转移酶与肝脏病变研究的新进展[J].环境卫生学杂志,1995,22(1): 8-11.[15] Wang Changsen,Castro A F,Wilkes D M,et al.Expression and purification of the first nucleotide-binding domain and linker region of human multidrug resistance gene product: comparison of fusions to glutathione S-transferase,thioredoxin and maltose-binding protein [J].Biochemical Journal,1999,338(1): 77-81.[16] 赵战勤,薛云,赵刚,等.重组百日咳杆菌黏附素C端蛋白的免疫原性[J].河南科技大学学报:自然科学版,2010,31(5):76-79.[17] Walker P A,Leong L E,Ng P W P,et al.Efficient and rapid affinity purification of proteins using recombinant fusionproteases[J].Biotechnology,1994,12(6): 601-605.。
蛋白融合表达蛋白融合表达是一种将外源蛋白基因与其他基因的一部分结合在一起进行表达的技术。
通过这种方法,外源蛋白可以作为融合蛋白的一部分被表达出来。
在蛋白融合表达中,通常会将外源蛋白基因与一个或多个其他基因的片段进行连接,形成一个融合基因。
这个融合基因会被转录和翻译,从而产生一个包含外源蛋白和其他基因片段的融合蛋白。
融合蛋白中的其他基因片段可以是一个特定的蛋白结构域、标签或信号序列等。
这种技术有许多优点。
首先,它可以提高目的蛋白的表达效率。
由于外源蛋白与其他基因片段的融合,可能会改变蛋白的稳定性、溶解性和活性,从而提高蛋白的表达量。
其次,融合表达可以帮助蛋白正确折叠,特别是对于一些容易形成错误折叠或聚集的蛋白来说,融合表达可以提供一个稳定的框架,促进正确的蛋白构象形成。
此外,可溶性表达也是融合表达的一个重要优势,因为一些蛋白在单独表达时可能难以溶解或形成包涵体,而融合表达可以增加蛋白的溶解性。
另外,融合表达中的蛋白标签,如His 标签等,为纯化蛋白提供了便利,使其更容易通过亲和层析等方法进行纯化。
最后,融合表达还可以使表达的蛋白更加稳定,减少蛋白的降解和失活。
在实际应用中,研究人员可以根据需要选择不同的基因片段进行融合,以实现特定的功能或研究目的。
例如,常用的融合标签包括His 标签、GST 标签、FLAG 标签等,它们可以用于蛋白的检测、纯化和定位等。
此外,融合表达还可以用于构建蛋白复合物、进行蛋白功能研究以及生产重组蛋白等领域。
总的来说,蛋白融合表达是一种强大的生物技术手段,它为蛋白研究和应用提供了多种可能性。
通过合理选择融合伙伴和优化表达条件,研究人员可以获得高表达、高纯度和具有特定功能的融合蛋白,推动各个领域的研究进展。
1 .1多聚精氨酸-标签(Arg-tag)精氨酸-标签通常由5或6个精氨酸组成.它已被成功用作细菌C末端标签,精氨酸是碱性最强的氨基酸,带5个精氨酸标签的蛋白质可以结合到阳离子交换树脂SP-Sephadex上, 而大部分杂蛋白不结合.结合后,带标签的蛋白质在碱性pH下运行线性NaCl梯度洗脱得到.当C末端为疏水性区域时,多聚精氨酸可能影响蛋白质的三级结构.氨酸残基的C末端序列可用羧肽酶B处理去除.这一酶促处理已被成功用于一些例子,但常常由于低的切割得率或者在期望的蛋白质序列间发生不必要的切割而受到限制.然而精氨酸标签并不常用,与第二标签联用是很有趣的蛋白质纯化工具.1.2 多聚组氨酸-标签(His-tag)已广泛采用的方法是利用固定化金属螯合层析纯化带有由多聚组氨酸残基组成的一个短的亲和标签的重组蛋白质.固定化金属螯合层析的基础是固定在基质上的过渡态金属离子(Co2+, Ni2+, Cu2+, Zn2+)与特定的氨基酸侧链之间的相互作用.组氨酸是与固定化金属离子作用最强的氨基酸,组氨酸的咪唑环作为电子供体容易与固定的金属离子形成配位键.基质材料洗涤之后,可通过调节柱缓冲液的pH或者添加游离咪唑洗脱含多聚氨基酸序列的肽类。
虽然在变性条件下系统对于带6个组氨酸标签的蛋白质纯化有效,带3个组氨酸标签的蛋白质在生理条件下可有效纯化.然而带6个组氨酸标签的蛋白质可以在天然条件下,在低或高浓度盐的缓冲液中结合到Ni2+-NTA 基质上.结合后,目标蛋白可用0.8到250 mM咪唑梯度洗脱.低浓度的咪唑(如0.8 mM)可减少带有组氨酸的宿主蛋白质的非专一吸附.6个组氨酸标签的蛋白质可用20到250mM咪唑浓度范围洗脱. 使用咪唑的缺点是咪唑的存在经常导致蛋白质的聚集.另一种用于纯化多聚组氨酸标签蛋白质的材料是TALON.这由Co2+-羧甲基天冬氨酸(Co2+-CMA)组成,并偶联到固相的树脂上.TALON使得标签蛋白在温和条件下洗脱,已有报导与Ni2+-NTA树脂相比,其非特异性蛋白结合更少,从而达到更高的洗脱产物纯度.一种酶制剂通过SDS-PAGE鉴定的纯度高于95%.1.3 谷胱甘肽S-转移酶-标签1988年首次描述了融合有谷胱甘肽S-转移酶(GST)的多肽的一步纯化.来自日本血吸虫(schisto soma japonicum),26-kDa的 GST被克隆在大肠杆菌表达载体中.融合蛋白可从未经处理的裂解液中用固定化的谷胱甘肽亲和层析加以纯化.结合的融合蛋白在非变性条件下用10mM 还原型谷胱甘肽洗脱..GST标签可用酶学分析或免疫分析很方便的检测.标签有助于保护重组蛋白免受胞外蛋白酶的降解并提高其稳定性.在大多数情况下GST融合蛋白是完全或部分可溶的.推荐采用位点专一的蛋白酶如凝血酶或Xa因子从融合蛋白切除GST标签.蛋白酶解后,GST载体和蛋白酶通过谷胱甘肽琼脂糖亲和层析去除.GST标签可位于N端或C端,可用于细菌,酵母,哺乳细胞, 和杆状病毒感染的昆虫细胞. GST融合蛋白已成为分子生物学家的基本工具.1.4 Strep-标签(Strep-tag)Strep-标签是一氨基酸肽开发来用于在链球菌抗生物素蛋白(streptavidin)柱上亲和纯化相应的融合蛋白.链球菌抗生物素蛋白(streptavidin)在特定位点第44, 45, 和47位的突变体与天然形式相比,对八肽Strep-tag II的亲和力更强,.Strep标签的蛋白质在生理缓冲液条件下结合到生物素结合区域中,并可用生物素衍生物温和洗脱.洗脱时推荐使用2.5 mM脱硫生物素.基质可以用4-羟基-偶氮苯-2-羧酸再生,这种物质在溶液中呈黄色,当结合到基质上则呈红色.结合条件是非常专一的.生物素化的蛋白质如大肠杆菌的羧基载体蛋白可以结合,但生物素或者生物素化的蛋白质则用抗生物素封闭.纯化条件是多种多样的.螯合剂,温和性去污剂,还原型去污剂,和高达1M的盐可以加到缓冲液中. Strep-标签系统适合用于研究蛋白质之间的相互作用以及当那些大的标签或者带电荷的标签无效时的特殊用途.1.5 S-标签S-标签是个融合肽标签可以通过快速灵敏的均匀分析或者在Western blot中用比色法加以检测.该系统的基础是15个氨基酸残基的S-tag与103氨基酸残基的S-蛋白质之间的强烈相互作用,这两个都来自RNaseA.S-蛋白质/S-标签复合物的Kd接近0.1μM,这取决于pH,温度和离子强度.标签由4个带正电荷残基,3个负电荷残基,3个不带电的极性残基和5个非极性残基组成.这使S -标签保持可溶.S-标签的快速分析是基于核酸降解活力的恢复.标签蛋白可以结合到S蛋白质基质上.洗脱条件较苛刻,如pH2.0的缓冲液.然而为了获得有功能的蛋白质推荐用蛋白酶切割标签.该系统可用于纯化的重组蛋白质来源包括细菌,哺乳细胞和杆状病毒感染的昆虫细胞抽提物.该系统经常与第二标签联用.RNase酶A高度灵敏的荧光底物的发现使得该系统可用于与高通量筛选联用的检测.2. 融合蛋白的切割为了对目的蛋白进行生化及功能分析,通常要从目的蛋白上去除fusion tag部分。
亲和标签的存在可能影响待研究蛋白的重要特性或功能.很早就已建立了数种对融合蛋白进行位点特异性裂解的方法。
化学裂解如溴化氰(Met↓)等,不但便宜且有效,往往还可以在变性条件下进行反应。
但由于裂解位点的特异性低和可能对目的蛋白产生的不必要修饰,使这个方法渐渐被酶解法取代。
酶解的方法相对来说反应条件较温和,更重要的是,普遍用于此用途的蛋白酶都具有高度的特异性。
位点特异性蛋白酶(例如凝血酶、肠激酶、Xa因子和烟草蚀刻病毒(TEV)等)通常被用来切割融合蛋白。
凝血酶在这三种中是特异性活性最高的,能够有效切质量比仅为1:2000的蛋白。
Xa因子似乎对于切点周围的序列很敏感,经常会出现特异性位点切割不理想却发生别的位点被切割的情况。
肠激酶的专一性是上述三种中最好的,但由于切割效率低(通常要求质量比达到1:10)而显得比较昂贵。
在质量比比较高的酶切反应进行完后,通常要通过色谱法处理。
比较方便可行的方法是采用生物素化凝血酶结合链亲和素琼脂糖一起使用。
尽管没有一种蛋白酶完美无缺,凝血酶还算的上是活性高、专一性好的典型。
蛋白的标签切割后不能降低蛋白质的活力. 另一个需要考虑的问题是:切割完成之后,是否需要去除蛋白酶。
切割后蛋白酶的去除对于使用带亲和标签的重组蛋白酶或者生物素化的蛋白酶较为容易.生物素化的蛋白酶可通过Strep -tag/Strep-Tactin亲和层析直接加以纯化.1.肠激酶由于专一识别5个氨基酸多肽(D-D-D-D-K-X1)并在赖氨酸的羧基上切割,是N末端融合通常选择的蛋白酶,在其他残基的不规则切割发生水平较低,这取决于蛋白质底物的构象(Choi et al.2001). 肠激酶轻链的分子量为26.3kDa.一个单位定义为在23℃下切割50ug 融合蛋白在8小时内达到95%切割率所需的酶量.生化分析已经表明切割效率取决于D4K识别位点下游的X1 氨基酸残基.2.TEV 蛋白酶是位点专一的蛋白酶,具有7个氨基酸残基的识别位点,序列为 E-XX-Y-X-Q-S, 切割在两个保守的谷氨酰胺和丝氨酸之间进行. X可以是各种氨基酸,但不是全部氨基酸都可行.最佳的切割序列为 E-N-L-Y-F-Q-S; .当TEV蛋白酶识别位点在两个结构域之间是结果最理想.当切割不理想时,插入增加结构灵活性的小连接肽可改善效率. 高专一性,多种底物的活力以及低温下的有效切割使TEV蛋白酶成为从融合蛋白移除标签的理想工具.3.凝血酶是一种广泛用于切割标签的蛋白酶.切割可在20到37℃之间切割0.3到16小时.与肠激酶相反,Xa因子和凝血酶切割后,在目标蛋白C末端增加了两个氨基酸.凝血酶最佳的切割位点是X4-X3-P-R[K]-X1'-X2',这里X4 和 X3 是疏水氨基酸而X1'和 X2'是非酸性氨基酸.一些经常使用的识别位点是L-V-P-R-G-S,L-V-P-R-G-F,和MY-P-R-G-N在X4-X3-P-R-G-X2'之间切割比在X4-X3-P-K-L-X2'更有效.其他识别位点是 X2-R[K]-X1', 这里X2 或者 X1'是甘氨酸.例如A-R-G和G-K-A,这里切割发生在第二个残基后.在凝血酶切割位点和N末端标签之间插入五个甘氨酸残基可改善切. 通过这一"甘氨酸连接肽"只需较少酶量就可达到完全切割,而且也可以避免可能发生的错误切割.有效的消化缓冲液是纯的Tris缓冲液,pH8.0.在缓冲液中存在的NaCl具有抑制作用.凝血酶可从切割产物中用p-氨基琼脂糖亲和纯化,或者superose-12 FPLC 柱凝胶过滤或者苯甲脒琼脂糖移去.4.在标签和目标蛋白之间的Xa因子识别位点可作为有效的工具完全去除N末端的亲和标签.Xa因子在四氨基酸肽I-E[D]-G-RX1的羧基切割, 这里X1可以是除了精氨酸和脯氨酸以外的其他氨基酸.切割可以在4到25℃之间进行.绝大多数X a因子的分子量大约43 kDa,由两个二硫键连接的多肽组成,一条27 kDa,一条 1 6 kDa.在 SDS-PAGE,还原的肽链具有30 kDa 和20 kDa的表观分子量. 通过位点专一的蛋白酶如Xa因子切除标签有时候是无效的,有Xa因子切割非专一的报导.原因可能是融合蛋白的不溶性或者变性剂的存在.可通过引入5个氨基酸的多聚甘氨酸区域来增加切割.丹磺酰氯-谷氨酸-甘氨酸-精氨酸-氯甲基酮在室温下1分钟内可使Xa因子活性不可逆失活95%.虽然由于切割需要更长的孵化时间而且效率较低而使Xa因子越来越少使用,但仍有使用Xa因子的成功报导。
裂解融合蛋白以除去fusion tagIMPACT系统的推出是融合表达系统的一个重大突破。
该系统最大的优点是表达的融合蛋白无需蛋白酶裂解即可实现目的蛋白与fusion tag的精确切割。
IMPACT(Intein Mediated Purif ication with an Affinity Chitin-binding Tag)系统利用一个来源于枯草杆菌的5kDa大小的几丁质结合域(chitin binding domain,用于亲和纯化)和一个来源于酵母intein蛋白质组成一个双效的fusion tag,再与克隆到多克隆位点的目的基因融合表达。
Intein是一个蛋白质剪接元件,类似于基因组中的内含子intron在RNA的剪接中所起的重要作用,intein在较低的温度和还原条件下发生自身介导的N端裂解,可以释放出与之相连的目的蛋白。