基因突变数据库介绍
- 格式:ppt
- 大小:7.14 MB
- 文档页数:22

TCGA数据库介绍TCGA(The Cancer Genome Atlas)是一个国际合作的项目,旨在通过全面研究多种人类癌症的基因组变异,进一步加深对癌症的认识。
TCGA项目由美国国立卫生研究院(NIH)和美国癌症研究所(NCI)联合发起,自2024年启动以来,已经成为全球最大的癌症基因组学项目之一TCGA项目收集并分析人体内约20种癌症的基因组数据,并将其公开发布在TCGA数据库中。
这些癌症类型包括但不限于结直肠癌、肺癌、乳腺癌、子宫内膜癌、肝癌和前列腺癌等。
通过对这些癌症样本的深度测序和分析,TCGA数据库提供了广泛的基因信息、表达谱、临床特征等数据,为研究人员和医疗专业人员提供了宝贵的资源。
TCGA数据库中的数据主要包括两个方面:基因组数据和临床数据。
基因组数据包括基因突变信息、DNA甲基化信息、基因拷贝数变异信息等。
这些信息可以帮助研究人员深入了解癌症发生和发展的分子机制,发现潜在的治疗靶点。
临床数据包括患者的生存数据、治疗信息、病理学特征等。
这些数据可以帮助医疗专业人员制定个性化治疗方案,改善癌症患者的生存率和生活质量。
TCGA数据库的数据共享政策使得全球的研究人员和医疗专业人员都可以自由访问和使用这些宝贵的数据资源。
研究人员可以利用这些数据开展各种癌症基因组学研究,寻找新的治疗方案和预测指标。
医疗专业人员可以利用这些数据指导临床决策,提供更好的个体化治疗。
除了数据本身,TCGA项目还提供了许多工具和平台,帮助用户更好地分析和解释数据。
例如,TCGA数据包含了丰富的调查表和数据字典,以帮助用户理解和使用数据。
此外,TCGA还提供了一系列的分析工具和软件,如GARFIELD、Firehose、DAVID等,以帮助用户进行数据挖掘和进一步分析。
TCGA数据库的影响已经超出了癌症研究领域。
许多其他研究和临床领域的学者也开始利用TCGA数据库进行多种疾病的基因组学研究,如心血管疾病、神经系统疾病等。

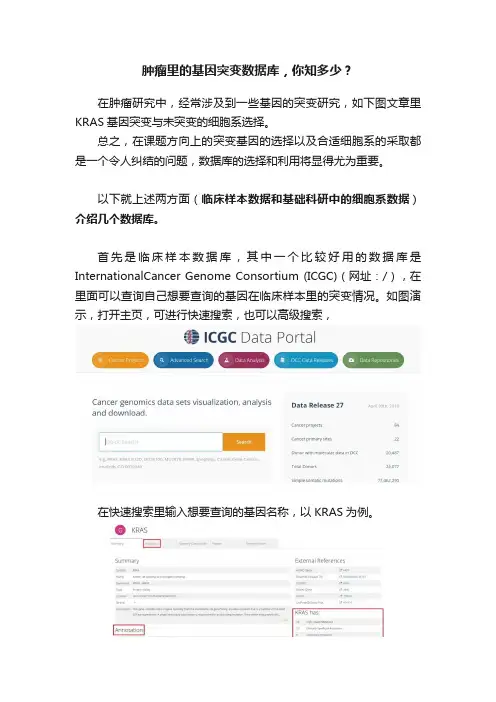
肿瘤里的基因突变数据库,你知多少?在肿瘤研究中,经常涉及到一些基因的突变研究,如下图文章里KRAS基因突变与未突变的细胞系选择。
总之,在课题方向上的突变基因的选择以及合适细胞系的采取都是一个令人纠结的问题,数据库的选择和利用将显得尤为重要。
以下就上述两方面(临床样本数据和基础科研中的细胞系数据)介绍几个数据库。
首先是临床样本数据库,其中一个比较好用的数据库是InternationalCancer Genome Consortium (ICGC)(网址:/),在里面可以查询自己想要查询的基因在临床样本里的突变情况。
如图演示,打开主页,可进行快速搜索,也可以高级搜索,在快速搜索里输入想要查询的基因名称,以KRAS为例。
可以看到KRAS的摘要,一些基本信息和注释,注释包括参与的反应通路和GO注释,同时也可以看到KRAS在临床样本中突变起了很大作用,但还没有靶向药物。
我们重点关注突变,点击Mutations,可以看到在很多临床项目和不同肿瘤类型中的KRAS突变情况,如果突变的多且还没有研究过,或许还有研究意义。
页面往下拉,还可以查看哪些位点突变的多,这个是很多其他数据库难以做到的,可以看到,在KRAS G12D这个位点突变的情况最多。
当然,这个数据库还有其他功能,如上KRAS在胰腺癌中突变最多,我们可以在高级搜索中选择胰腺癌,查看基因,可以看到KRAS 排名第一,继续点击最右红框里的光标,可看到突变与未突变生存曲线的比较,ICGC这个数据库还有其他功能,大家可以自行探索一下。
第二个问题,在基础科研做突变基因研究,肯定离不开细胞,就如第一张截图文献里的细胞系选取,他们是怎么做到的,难道一个个测序?那肯定不是,有几个数据库可以了解一下。
第一个,CCLE(CancerCell Line Encyclopedia),翻译过来就是癌细胞百科全书,够牛逼了。
网址:/ccle,里面包含了很多癌细胞系的基因表达和突变的信息。

tcga数据库使用方法TCGA(The Cancer Genome Atlas)数据库是一个重要的公共数据资源,为研究人员提供了大量的癌症基因组数据。
本文将介绍如何使用TCGA数据库进行数据获取和分析,以帮助读者更好地利用这一资源。
1. TCGA数据库简介TCGA数据库是由美国国立癌症研究所(NCI)和国立人类基因组研究所(NHGRI)联合推出的一个综合性癌症基因组学数据库。
该数据库整合了来自世界各地的研究机构共享的癌症基因组数据,包括肿瘤组织样本和正常对照样本的基因表达、突变、甲基化等数据。
2. 数据获取要使用TCGA数据库,首先需要访问官方网站(www . tcga . nih . gov)。
在网站主页上,你可以找到关于TCGA项目的详细信息,包括参与机构、数据类型等。
3. 数据筛选在进入TCGA数据库后,你可以根据自己的研究需求进行数据筛选。
首先,选择你感兴趣的癌症类型,例如乳腺癌、肺癌等。
其次,根据不同的研究目的,你可以在筛选条件中选择不同的数据类型,比如基因表达数据、突变数据、甲基化数据等。
另外,你还可以根据样本类型(肿瘤组织、正常对照组织等)和患者特征(性别、年龄等)进行筛选。
4. 数据下载在完成数据筛选后,你可以选择下载符合条件的数据。
TCGA数据库提供了多种数据下载方式,包括整个癌症类型的数据包或特定基因的数据。
你可以选择合适的下载方式,并按照指引完成下载过程。
5. 数据分析在获取到TCGA数据库的数据后,你可以使用各种生物信息学工具对数据进行分析。
例如,你可以使用R语言中的Bioconductor包、Python中的pandas库等进行数据处理和统计分析。
根据具体的研究需求,你可以进行差异表达分析、生存分析、通路分析等。
6. 结果解释在完成数据分析后,你需要解释和讨论你的结果。
根据研究问题的不同,你可以从不同的角度对结果进行解读。
你可以参考相关文献和数据库,验证你的结果是否与已有研究相符,并给出你自己对结果的解释。

数据库新生突变研究,突变速率不容忽视mirDNMRActionFree一个追求高品质生活异想天开的浪漫疯子,一只研究精神疾病与脑的遗传的科研汪...生活不该只停留在想法而无实际行动,做一个勇敢的行动者,去尝试去努力。
此公众号旨在分享本汪在科研及生活中的一些实践报告、学习笔记,期待与你的交流与合作。
戳这里ActionFree公众号原文,请多关注哦~ActionFree新生突变(de novo mutation 又叫新发突变)是指父母中没有的,但在孩子新发生的突变,它在散发性遗传疾病的研究中扮演重要角色。
有研究证明,人类基因组上平均每代人自发发生74个新生单核苷酸突变和3个新生插入缺失,而这些突变中只有极少数会致病。
从如此多的新生突变中准确找到致病突变并筛选致病基因具有重要意义。
以往的研究通过寻找在基因中频发的突变(即在多个家系中出现的突变)筛选致病基因。
然而,新生突变可能会随机地在某个基因中出现多次。
而且,出现这种现象的概率会随着基因长度的增长而增大。
因此,仅仅基于频发突变筛选致病基因存在极大的假阳性,并且缺乏统计方法的支持。
也有研究采用case-control的方式筛选致病基因。
然而,由于在某些基因上新生突变的分布极其稀少,仍然难以应用统计学方法进行研究。
并且,大规模地对control进行测序耗资巨大。
如果对每个基因的背景突变速率有个准确的估计,则可以在不对control 进行测序的前提下准确地筛选致病基因。
实际上,目前已经有多个研究应用不同的方法计算了每个基因的背景突变速率,并且应用该背景值找到了候选致病基因。
mirDNMR数据库(/mirdnmr/index.php)收集了人类新生突变的背景突变速率。
该数据库可供用户浏览和搜索不同基因的背景突变速率,同时还可以基于背景突变速率筛选候选致病基因。
该数据库还可供用户搜索不同人群的突变频率,包括:ExAC、ESP6500、UK10K、1000G和dbSNP。


ncbi突变命名规则(实用版)目录1.NCBI 的概述2.基因突变的命名规则3.突变类型的分类4.突变命名的实例分析5.突变命名规则的实际应用正文【1.NCBI 的概述】CBI(National Center for Biotechnology Information)是美国国家生物技术信息中心,负责收集、存储和传播生物学和医学研究所需的各种信息资源。
在基因研究领域,NCBI 建立了一个基因序列数据库,为科研人员提供基因相关的数据和资源。
【2.基因突变的命名规则】基因突变是指基因序列发生的改变,可能导致生物体表现出不同的表型。
为了方便描述和交流,NCBI 对基因突变进行了命名规则的制定。
基因突变的命名主要由三部分组成:基因名称、突变位点以及突变类型。
【3.突变类型的分类】突变类型主要分为以下几类:(1)点突变(Point Mutation):是指基因序列中的一个碱基被另一个碱基替换,导致氨基酸序列发生改变。
(2)插入突变(Insertion):是指基因序列中插入了一个或多个额外的碱基,导致氨基酸序列发生改变。
(3)删除突变(Deletion):是指基因序列中删除了一个或多个碱基,导致氨基酸序列发生改变。
(4)移位突变(Frame Shift):是指基因序列中的一段碱基发生移位,导致阅读框发生改变,进而使氨基酸序列发生改变。
(5)倒位突变(Inversion):是指基因序列中的一段碱基序列发生倒置,导致氨基酸序列发生改变。
【4.突变命名的实例分析】以一个点突变为例,假设在基因名为“ABC”的基因上,发生了一个 C 碱基替换 G 碱基的突变,突变位点为第 100 个碱基,那么该突变的命名应为“ABC:c.100G>C”。
【5.突变命名规则的实际应用】突变命名规则在基因研究和遗传病诊断中有着广泛的应用。
科研人员可以通过突变命名快速了解基因突变的类型和位点,便于开展进一步的研究。
同时,对于遗传病的诊断,突变命名有助于医生和患者准确地了解病情,以便采取更有效的治疗措施。

tcga数据库使用方法TCGA(The Cancer Genome Atlas)数据库是一个公共数据资源,为研究人员提供了丰富的肿瘤基因组学数据。
本文将简要介绍TCGA数据库的使用方法,并向读者展示如何利用该数据库获取和分析数据。
一、什么是TCGA数据库TCGA数据库是由美国国立卫生研究院(NIH)与癌症学会(ACS)合作建立的一个肿瘤基因组学资源。
该数据库集结了来自全球多个研究机构的数据,包括肿瘤样本的临床信息、基因组数据、转录组数据等。
这些数据可供科学家们使用,以加深对癌症的理解并推动治疗的发展。
二、访问TCGA数据库想要使用TCGA数据库,首先需要访问TCGA的官方网站(https:///)。
在该网站上,你需要创建一个账户才能获得全面的数据访问权限。
创建账户后,你就可以开始使用TCGA数据库。
三、查找和下载数据在登录后,你可以使用TCGA网站提供的搜索功能来查找你感兴趣的数据。
你可以按照肿瘤类型、患者的临床特征、数据类型等多个维度进行筛选。
在找到合适的数据后,你可以将它们添加到购物车,并下载相应的数据文件。
四、解析和分析数据下载完数据后,你可以使用多种生物信息学工具和软件来解析和分析这些数据。
这些工具和软件可以帮助你寻找肿瘤的基因突变、基因表达情况以及表观遗传学改变等。
其中一些工具还提供了数据可视化的功能,以帮助你更好地理解和呈现你的研究结果。
五、数据共享和合作TCGA数据库鼓励科学家之间的数据共享和合作。
你可以将你的研究结果上传到TCGA数据库,与其他研究人员分享你的发现和数据。
这种合作可以促进知识交流和科学进步。
TCGA数据库作为一个全球性的公共资源,为癌症研究者提供了重要的数据支持。
通过充分利用这个数据库,我们能够更好地研究癌症的发生机制、诊断方法以及潜在的治疗策略。
希望本文所提供的TCGA数据库的使用方法对于你的研究工作有所帮助。
这篇文章介绍了TCGA数据库的使用方法,包括访问数据库、查找和下载数据、解析和分析数据,以及数据共享和合作等内容。

TCGA数据库介绍TCGA(The Cancer Genome Atlas)是由美国国立癌症研究所(NCI)和美国国立人类基因组研究所(NHGRI)共同发起的一个大型国际性癌症基因组计划。
该计划的目标是通过对人类癌症进行全面的基因组学分析,以帮助科学家更好地理解癌症的发生机制,识别潜在的治疗靶点,并为个性化医疗提供关键信息。
TCGA数据库提供了多种类型的基因组数据,包括基因组测序数据、表达谱数据、DNA甲基化数据、蛋白质表达数据等。
每个样本都经过详细的基因组学分析,使得科学家可以探索癌症的发生机制、转录组表达变化、基因突变和表达、DNA甲基化等方面的信息。
除了数据规模之外,TCGA数据库的另一个显著特点是其数据的多样性。
由于TCGA采集了全球范围内的癌症样本,包括不同类型的癌症和不同种族、性别和年龄的患者,因此其数据库中的数据具有一定的代表性和覆盖性。
这使得科学家在比较不同类型的癌症、寻找特定变异或基因表达的相关性时具有更高的可靠性。
TCGA数据库对于癌症研究以及相关领域的研究有着重要的意义。
首先,它为癌症研究提供了宝贵的资源和参考。
科学家可以利用TCGA数据库中的数据与自己的研究进行验证和比较,进一步加深对癌症的认识。
其次,TCGA数据库还为研究人员提供了一个共享和交流的平台。
任何人都可以访问TCGA数据库并使用其中的数据进行自己的研究,促进了全球范围内的合作和共同进展。
最后,TCGA数据库的开放性和透明度也为临床医生和患者提供了一个参考资源,帮助他们做出更准确的医疗决策和制定个性化的治疗方案。
然而,需要注意的是,TCGA数据库也存在一些限制和挑战。
首先,由于大规模基因组数据的复杂性和多样性,对于非专业研究人员来说,理解和解释TCGA数据可能是一项挑战。
其次,基因组数据的分析和解释需要一定的专业知识和技能,并且需要使用适当的分析工具和软件进行处理。
此外,由于TCGA数据库只包含了限定数量和类型的癌症数据,所得到的研究结果可能并不适用于所有类型的癌症或个体患者。

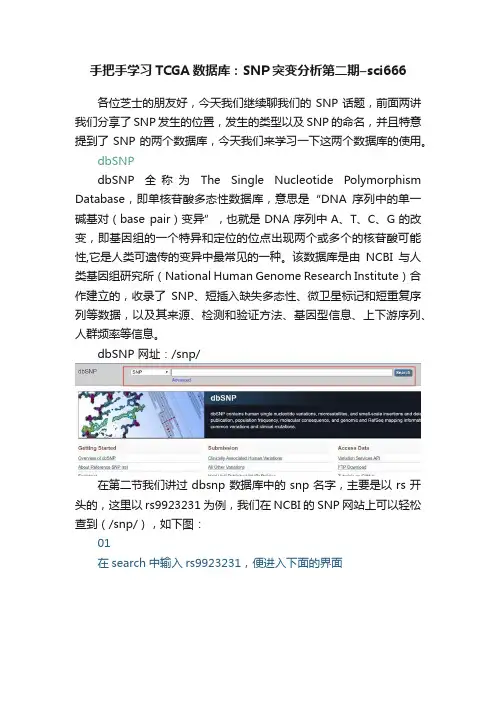
手把手学习TCGA数据库:SNP突变分析第二期–sci666各位芝士的朋友好,今天我们继续聊我们的SNP话题,前面两讲我们分享了SNP发生的位置,发生的类型以及SNP的命名,并且特意提到了SNP的两个数据库,今天我们来学习一下这两个数据库的使用。
dbSNPdbSNP 全称为The Single Nucleotide Polymorphism Database,即单核苷酸多态性数据库,意思是“DNA序列中的单一碱基对(base pair)变异”,也就是DNA序列中A、T、C、G的改变,即基因组的一个特异和定位的位点出现两个或多个的核苷酸可能性,它是人类可遗传的变异中最常见的一种。
该数据库是由NCBI与人类基因组研究所(National Human Genome Research Institute)合作建立的,收录了SNP、短插入缺失多态性、微卫星标记和短重复序列等数据,以及其来源、检测和验证方法、基因型信息、上下游序列、人群频率等信息。
dbSNP 网址:/snp/在第二节我们讲过dbsnp数据库中的snp名字,主要是以rs开头的,这里以rs9923231为例,我们在NCBI的SNP网站上可以轻松查到(/snp/),如下图:01在search中输入rs9923231,便进入下面的界面主要有下列信息:分别是Variant type(变异类型)、Alleles (等位基因)、Chromosome (染色体位置)、Gene (位于基因的名字)、Functional Consequence(功能结果)、Clinical significance (临床价值)、Validated(验证类型)、Global MAF(MAF格式文件注释)、HGVS:(HGVS数据库注释)02继续点击rs9923231,便出现下面的界面你会发现跳入到新的界面,便是对该位点的详细介绍,这个时候看到一个Switch to class site界面,点击进去发现进入到的是经典的站点,如下:这个提示我们该站点将会停止使用,并推荐我们进入新站点,即我们最开始看到的,那我们就在新站点学习一下该网站使用。
⽣物数据库介绍——NCBINCBI(National Center for Biotechnology Information,美国国家⽣物技术信息中⼼)除了维护GenBank核酸序列数据库外,还提供数据分析和检索资源。
NCBI资源包括Entrez、Entrez编程组件、MyNCBI、PubMed、PudMed Central、PubReader、Gene、the NCBI Taxonomy Browser、BLAST、Pimer-Blast、COBALT、RefSeq、UniGene、HomoloGene、ProtEST、dbMHC、dbSNP、dbVar、Epigenomics、the Genetic Testing Registry、Genome和相关⼯具、⽐对查看器、跟踪存档、Sequence Read Archive、BioProject、BioSample、ClinVar、MedGen、HIV-1/⼈类蛋⽩质相互作⽤数据库、Gene Expression Omnibus、Probe、Online Mendelian Inheritance in Animals、the Molecular Modeling Database、the Conserved Domain Database、the Conserved Domain Architecture Retrieval Tool、Biosystem、Protein Clusters and thePubChem suite of small molecule databases,所有这些资源可以在NCBI主页找到。
Databases⼀个提供有关基因组组装结构,装配名称和其他元数据,统计报告以及基因组序列数据链接等信息的数据库。
⼀个有关培养物、动植物样本和其他⾃然样本的精选元数据集。
记录显⽰样本状态,有关馆藏的机构的信息,以及NCBI中相关数据链接。
基因突变数据库介绍ppt xx年xx月xx日contents •引言•基因突变数据库概述•基因突变数据库的应用•基因突变数据库的未来发展•结论目录01引言介绍基因突变数据库的意义01基因突变数据库是生物信息学研究的重要方向之一,对于基因组学、遗传学和医学等领域的研究具有重要意义。
02基因突变会导致遗传性疾病和癌症等多种疾病,而基因突变数据库可以记录和分析这些突变,为研究这些疾病的原因和机制提供支持。
03基因突变数据库也可以帮助科学家更好地理解基因的多样性和演化,为药物研发和个性化医疗等方面提供参考。
基因突变数据库的应用前景基因突变数据库在医学和生物技术领域具有广泛的应用前景。
同时,基因突变数据库也可以为新药研发提供重要的参考信息,帮助科学家更快地找到潜在的治疗方法。
例如,通过分析基因突变数据库,可以帮助医生更好地诊断和治疗遗传性疾病和癌症等疾病。
此外,基因突变数据库还可以应用于农业和生态学等领域,为作物改良和环境保护等方面提供支持。
本次介绍将包括基因突变数据库的基本概念、发展历程、现状和未来趋势等方面。
通过详细了解基因突变数据库的背景和应用,可以更好地了解其重要性和价值,为相关领域的研究和应用提供参考。
本次介绍的概要02基因突变数据库概述基因突变是指DNA分子中发生碱基对的替换、增添和缺失,导致基因结构改变的现象。
基因突变是生物进化的重要驱动力,也是生物多样性的重要来源。
基因突变的基本概念基因突变的常见方式包括点突变、插入突变、缺失突变、倒位和易位等。
基因突变的常见原因包括DNA复制错误、环境因素影响、化学物质诱导、辐射诱导等。
基因突变的方式和原因基因突变数据库的建立需要收集、整理和注释大量的基因突变数据。
基因突变数据库的建立及分类基因突变数据库可以分为突变数据库和疾病相关数据库两类。
突变数据库主要收集基因突变类型及其频率等信息,用于研究突变与生物进化、物种分化等方面的关系;而疾病相关数据库则主要收集与特定疾病相关的基因突变信息,用于研究疾病发生、发展和治疗等方面的规律。
手把手学习TCGA数据库:SNP突变分析第七期结果如下:接着问题来了,如果我们想对指定的基因进行突变频率可视化呢?该怎么操作呢?这个时候我们需要借助oncoplot的genes参数,比如我们向可视化TTN,IDH2,TET2,NRAS,TP53,SMC3这几个基因,那命令行则如下:结果如下:你会发现指定的基因排序是按照突变频率由高到低进行排列,如果你不想改变基因的排列顺序,则可以增加一个参数,如下:结果如下:除此之外,我们还可以使用oncostrip函数进行可视化任意基因,如下:转换和颠换数据可视化前面在讲解SNP基础的时候,我们讲到转换则是嘌呤变嘌呤或者嘧啶变嘧啶,颠换则是异型碱基的置换,一个嘌呤被另外一个嘧啶替换或一个嘧啶被另外一个嘌呤置换,即嘌呤变嘧啶,或者嘧啶变嘌呤。
那么在maf文件里面这些信息也是可以进行展示的,需要借助titv 函数将snp分类为转换和转换,并进行展示。
命令如下:结果如下:Ti代表转换,Tv代表颠换,我们可以发现Ti是Tv的3倍,一般情况下发生转换和颠换频率是2:1。
看到这,我们好像没把临床数据加载进来,这个时候我们可以将临床数据进行加载进去,还是同样的操作,同样采用内置的急性髓性白血病为例子,读进数据如下:可以看到我们同样借助read.maf函数进行操作,只不过在clinicalData参数后面赋值了laml.clin而已,这个时候的laml对象则涵盖了maf文件同时还包括了临床数据。
这个时候我们在进行绘制瀑布图,如下:可以看到我们是可视化了两个临床特征,分别为FAB_classification和Overall_Survival_Status,结果如下:Ok,今天的教程主要是带大家继续采用maftools对maf文件进行处理,希望大家能到学会如何使用maftools,谢谢大家。
·end·。
KEGG数据库使用方法详解KEGG(Kyoto Encyclopedia of Genes and Genomes)是一个整合了基因组、生物信息、代谢途径和疾病等多种数据的数据库。
它提供了一种系统的方法来理解生物分子行为和生物系统的功能。
本文将详细介绍KEGG数据库的使用方法。
首先,KEGG数据库提供了基因组和基因信息的查询功能。
用户可以通过基因ID、基因名或其他基因标识符来特定的基因,并获得与该基因相关的信息,如基因的序列、结构和功能注释等。
此外,用户还可以查询特定物种的基因组信息,如基因组大小、基因组组成和染色体标记等。
第二,KEGG数据库提供了生物信息学工具来预测和分析基因的功能和相互作用。
例如,KEGG可以根据基因或蛋白质的序列信息,通过基因注释和蛋白质相似性等方法,预测基因的功能和相互作用。
此外,KEGG 还提供了一系列的生物信息学工具,用于分析基因的表达模式、基因家族和进化关系等。
这些工具可以帮助研究人员更好地理解基因在细胞、组织和器官水平上的功能。
第三,KEGG数据库还提供了代谢途径和信号传导网络的详细信息。
用户可以查询特定物种的代谢途径和信号传导网络的图表,并查看其中包含的基因、蛋白质和化合物等分子的详细信息。
此外,用户还可以通过KEGG数据库来预测和分析代谢途径的调控机制、信号传导通路的调控网络等。
这些功能可以帮助研究人员更好地理解代谢途径和信号传导网络在生物系统中的作用和调控机制。
第四,KEGG数据库提供了疾病和药物的相关信息。
用户可以查询特定疾病或药物的相关信息,并了解其与基因、代谢途径和信号传导网络的关联。
此外,KEGG还提供了一些工具来预测和分析疾病和药物的相互作用,如药物的靶点和作用机制等。
这些功能可以帮助研究人员更好地理解疾病的发生和药物的治疗机制。
最后,KEGG数据库还提供了一些工具和资源来帮助研究人员进行数据分析和可视化。
例如,KEGG提供了一些图形化工具来展示基因、代谢途径和信号传导网络的关系。
人类基因突变及疾病相关数据库> 资源 > 生物大数据 >人类基因突变及疾病相关数据库1.HGMD人类基因变异数据库(HMGD)收集公开发表的引起人类遗传疾病的胚系突变信息。
范围限定在导致明确遗传表型的突变,体细胞突变和线粒体突变也列入其中。
HGMD检索界面主要以文本为基础,目标检索依赖正常的基因的HUGO命名知识。
2.HGBASE(Gwas central)人类遗传双等位基因序列数据库(HGBASE)是人类基因从启动子到转录终点,即基因及其前后所发现的所有单核苷酸多态性和其他变化的数据库。
3.OMIM人类孟德尔遗传在线(OMIM)是以人类孟德尔遗传与疾病(MIM)为基础的人类基因及其相关突变的在线目录。
可用于查找疾病相关基因及位点,并涵盖了不同程度的综合性疾病的资料。
4. KMDB/MutationView/MutationView/jsp/index.jspKeio Mutation Databases, 提供人类疾病相关基因突变,涵盖眼、耳、心脏、肿瘤、自身免疫性疾病、肌肉及血液等方面疾病基因。
5.KinMutBase酪氨酸激酶区域突变导致疾病的数据库,同时可链接其他突变数据库。
6. Atlas Chromosomes in CancerAtlas of Genetics and Cytogenetics,提供癌症和癌症倾向疾病的生物学和分子方面信息。
7.dbSNP人类单核苷酸多态性数据库(dbSNP)是由NCBI与人类基因组研究所合作建立的,关于单碱基替换以及短片段插入、删除多态性的资源库。
8.TGDB肿瘤基因家族数据库(TGDBs)包含了有关肿瘤的一系列基因数据,如:原癌基因和抑癌基因。
基因信息包括:原癌基因的激活、调节的机制、在不同癌症类型中的相关频率以及染色体的定位。
有关蛋白的信息包括:该蛋白存在于何种细胞类型,亚细胞的定位,DNA序列,配体的结合,在发育过程中的作用等等。