NCBI序列比对方法与实例操作
- 格式:ppt
- 大小:2.09 MB
- 文档页数:15


ncbi序列比对步骤嘿,朋友们!今天咱就来聊聊 ncbi 序列比对那档子事儿。
你知道不,这 ncbi 序列比对就像是给基因们来一场相亲大会!咱得让它们找到最合适的那个“伴儿”。
首先呢,咱得把咱手头的序列准备好,就像给基因们打扮得漂漂亮亮的,准备去见心仪的对象。
这可不能马虎,得仔细着呢!然后呢,就到了关键的一步,打开 ncbi 的大门,就像走进了一个神奇的基因世界。
在那里面,各种序列都在等着和咱的宝贝序列来个亲密接触。
接着,按照指示一步步操作,选择合适的比对工具,这就好比给基因们选一个最合适的约会场所。
这可不能瞎选,得选个能让它们尽情展示自己的地方。
在比对的过程中,你就想象基因们在那里面欢快地交流、互动,看看谁和谁最合拍。
有时候啊,可能会出现一些不太理想的结果,就好像相亲的时候遇到了不太对眼的,但别灰心,咱继续找呀!等啊等,终于等到比对结果出来了。
这时候可得瞪大眼睛好好瞅瞅,看看哪些基因是真正的“天作之合”。
这感觉,就像是看到了自己的基因宝贝找到了真爱一样开心。
要是结果不太满意,咱也别着急上火,再重新来一遍呗,就当是给基因们多几次相亲的机会。
哎呀,这 ncbi 序列比对啊,真的是既有趣又充满挑战。
就像我们在生活中寻找自己的位置一样,需要耐心和细心。
咱可不能小瞧了这每一个步骤,一个不小心可能就错过了最佳的比对结果。
所以啊,每一步都得认真对待,就像对待生活中的每一个选择一样。
总之呢,ncbi 序列比对就是一场基因的奇妙之旅,让我们一起在这个神奇的世界里探索吧,说不定会有很多意想不到的惊喜等着我们呢!你说是不是?。

NCBI在线BLAST使用方法与结果详解BLAST(Basic Local Alignment Search Tool)是一套在蛋白质数据库或DNA数据库中进行相似性比较的分析工具。
BLAST程序能迅速与公开数据库进行相似性序列比较。
BLAST结果中的得分是对一种对相似性的统计说明。
BLAST 采用一种局部的算法获得两个序列中具有相似性的序列。
Blast中常用的程序介绍:1、BLASTP是蛋白序列到蛋白库中的一种查询。
库中存在的每条已知序列将逐一地同每条所查序列作一对一的序列比对。
2、BLASTX是核酸序列到蛋白库中的一种查询。
先将核酸序列翻译成蛋白序列(一条核酸序列会被翻译成可能的六条蛋白),再对每一条作一对一的蛋白序列比对。
3、BLASTN是核酸序列到核酸库中的一种查询。
库中存在的每条已知序列都将同所查序列作一对一地核酸序列比对。
4、TBLASTN是蛋白序列到核酸库中的一种查询。
与BLASTX相反,它是将库中的核酸序列翻译成蛋白序列,再同所查序列作蛋白与蛋白的比对。
5、TBLASTX是核酸序列到核酸库中的一种查询。
此种查询将库中的核酸序列和所查的核酸序列都翻译成蛋白(每条核酸序列会产生6条可能的蛋白序列),这样每次比对会产生36种比对阵列。
下面是具体操作方法1,进入在线BLAST界面,可以选择blast特定的物种(如人,小鼠,水稻等),也可以选择blast所有的核酸或蛋白序列。
不同的blast程序上面已经有了介绍。
这里以常用的核酸库作为例子。
2,粘贴fasta格式的序列。
选择一个要比对的数据库。
关于数据库的说明请看NCBI在线blast数据库的简要说明。
一般的话参数默认。
3,blast参数的设置。
注意显示的最大的结果数跟E值,E值是比较重要的。
筛选的标准。
最后会说明一下。
4,注意一下你输入的序列长度。
注意一下比对的数据库的说明。
5,blast结果的图形显示。
没啥好说的。
6,blast结果的描述区域。
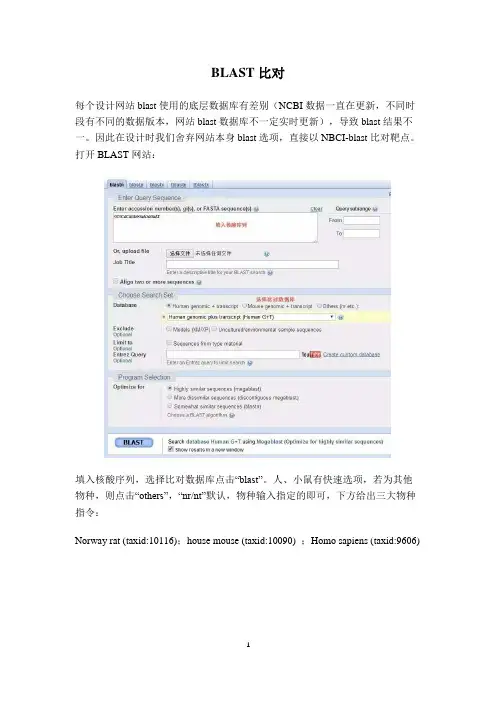
BLAST比对
每个设计网站blast使用的底层数据库有差别(NCBI数据一直在更新,不同时段有不同的数据版本,网站blast数据库不一定实时更新),导致blast结果不一。
因此在设计时我们舍弃网站本身blast选项,直接以NBCI-blast比对靶点。
打开BLAST网站:
填入核酸序列,选择比对数据库点击“blast”。
人、小鼠有快速选项,若为其他物种,则点击“others”,“nr/nt”默认,物种输入指定的即可,下方给出三大物种指令:
Norway rat (taxid:10116);house mouse (taxid:10090) ;Homo sapiens (taxid:9606)
结果界面,Max Score列数值除以2表示匹配的碱基数;完全匹配的全部都是TP53基因的15个转录本,所以,靶点位于同源区;其次,非完全靶向的Max Score最大值为30.2,也就是跟基因SIPA1L2实际结合15个碱基,错配四个,符合特异性原则,该靶点blast结果OK。
以上即靶点设计及比对的流程,该方法同样适合非编码RNA,选择多个网站设计的共有靶点以及设计2~3个靶点进行验证,更加有利于筛选出有效靶点。

实验六序列相似性的比对和搜索一、实验目的1.能够熟练使用NCBI网站的BLAST系列工具,通过NCBI中的BLAST功能,对所提供的基因组序列或蛋白质序列进行相似性比对,找到在GenBank中与之相似的序列,推测所比对序列的功能。
2.能够熟练掌握用Clustalx软件进行双序列和多序列比对。
3.学会使用EMBL上的Clustalw工具进行比对。
二、实验内容及操作步骤(一)BLAST的使用1.Blastn:进入NCBI主页下载关于AY125911、AF513548、AF525146、AF492473、AY497910、AY497911等核酸序列或其它你感兴趣的核酸序列(Fasta格式)。
1)进入/BLAST/;2)选择Nucleotide→Nucleotide-nucleotide BLAST (blastn)进行核酸相似性数据库搜索;3)在search对话框中粘贴入下载的相关核酸序列(Fasta格式);4)调整各参数值,直到获得最佳比对;5)点击进行比对;6)点击Format!对结果进行格式化,可在下面的选项中自行设计结果的显示方式;7)查看比对结果,看在数据库中找到的序列与你的序列是否相似或相同。
2.Blastp:进入NCBI主页下载某一蛋白质序列(Fasta格式),如cytochrome oxidase, peroxidase, SOD (Superoxide Dimutase)。
1)选择Protein→Protein-protein BLAST (blastp)进行蛋白质相似性数据库搜索;2)在search对话框中粘贴入下载的蛋白质序列(Fasta格式);3)调整各参数值,直到获得最佳比对;4)点击进行比对;5)点击Format!对结果进行格式化,可自行设计结果的显示方式;6)查看比对结果,看在数据库中找到的序列与你的序列是否相似或相同。
3.Bl2seq:进入NCBI主页下载某两条核酸或蛋白质序列(Fasta格式)1)进入/BLAST/;2)点击Special目录下的Align two sequences (bl2seq);3)将两条序列分别输入Sequence 1和Sequence 1区域;4)点Align进行比对;5)根据结果查看bl2seq是否允许插入空位。

NCBI在线BLAST使用方法与结果详解NCBI在线BLAST(Basic Local Alignment Search Tool)是一种广泛使用的生物信息学工具,用于比对和分析DNA、RNA或蛋白质序列。
它可以对已知和未知序列进行,找到与查询序列相似的序列,并提供有关相似性和功能的信息。
使用NCBI在线BLAST可以分为四个主要步骤:选择BLAST程序,输入查询序列,选择目标数据库,解析和分析结果。
第一步:选择BLAST程序NCBI提供了多种BLAST程序可供选择,包括BLASTN(DNA对DNA的比对)、BLASTP(蛋白质对蛋白质的比对)、BLASTX(DNA对蛋白质的比对)等。
根据实际需求选择相应的BLAST程序。
第二步:输入查询序列在查询序列的文本框中输入待比对的序列。
可以输入单个序列,也可以上传包含多个序列的文件。
如果输入的序列是DNA或RNA序列,需要选择相应的序列类型。
此外,还可以选择是否使用掩码序列或低复杂性筛选来优化比对结果。
第三步:选择目标数据库用户可以选择目标数据库来与查询序列相似的序列。
NCBI提供了多个常用的数据库,如nr(非冗余蛋白质数据库)、nt(核酸数据库)等。
此外,还可以选择特定的物种数据库来限制比对范围。
第四步:解析和分析结果在BLAST运行完成后,会生成一个结果页面,其中包含了比对结果的详细信息。
结果页面包括比对统计信息、序列比对图、E值、分数等。
通过分析这些信息,可以了解查询序列与目标数据库中的序列之间的相似性和可能的功能。
此外,NCBI在线BLAST还提供了一些高级选项,例如使用特定的算法或参数来进行比对、设置比对阈值、选择比对输出格式等。
这些选项可以根据实际需求进行调整。
总结起来,使用NCBI在线BLAST可以通过选择BLAST程序、输入查询序列、选择目标数据库以及解析和分析结果来比对和分析序列。
通过权衡算法和参数选择,在特定数据库中找到与查询序列相似的序列,从而获得有关其相似性和功能的信息。

一步一步教你使用NCBI 查找DNA、mRNA、cDNA、Protein、promoter、引物设计、BLAST序列比对等最近看到很多战友在论坛上询问如何查询基因序列、如何进行引物设计、如何使用BLAST 进行序列比对……,这些问题在 NCBI 上都可以方便的找到答案。
现在我就结合我自己使用 NCBI的一些经历(经验)跟大家交流一下 BCBI 的使用。
希望大家都能发表自己的使用心得,让我们共同进步!我分以下几个部分说一下 NCBI 的使用:Part one 如何查找基因序列、mRNA、PromoterPart two 如何查找连续的 mRNA、cDNA、蛋白序列Part three 运用 STS 查找已经公布的引物序列Part four 如何运用 BLAST 进行序列比对、检验引物特异性特别感谢本版版主,将这个帖子置顶!从发帖到现在,很多战友对该帖给与了积极的关注,在此向给我投票的(以及想给我投票却暂时不能投票的)各位战友表示真诚的感谢,谢谢各位战友!请大家对以下我发表的内容提出自己的意见。
关于NCBI 其他方面的使用也请水平较高的战友给予补充First of all,还是让我们从查找基因序列开始。
第一部分利用Map viewer 查找基因序列、mRNA 序列、启动子(Promoter)下面以人的 IL6(白细胞介素 6)为例讲述一下具体的操作步骤1.打开Map viewer 页面,网址为:/mapview/index.html 在 search 的下拉菜单里选择物种,for 后面填写你的目的基因。
操作完毕如图所示:2.点击“GO”出现如下页面:3.在步骤二图示的右下角有一个Quick Filter,下面是让你选择的几个复选框,在Gene 前面的小方框里打勾,然后点击Filter. 出现下图:说明一下:1、染色体的红色区域即为你的目的基因所处位置。
2、下面参考序列给出了三个,是不同的部门做出来的,经我验证,序列有微小的差异,但总体来说基本相同。

一步一步教你使用 NCBI 查找DNA、mRNA、cDNA、...最近看到很多战友在论坛上询问如何查询基因序列、如何进行引物设计、如何使用BLAST 进行序列比对……,这些问题在 NCBI 上都可以方便的找到答案。
现在我就结合我自己使用 NCBI的一些经历(经验)跟大家交流一下 BCBI 的使用。
希望大家都能发表自己的使用心得,让我们共同进步!我分以下几个部分说一下 NCBI 的使用:Part one 如何查找基因序列、mRNA、PromoterPart two 如何查找连续的 mRNA、cDNA、蛋白序列Part three 运用 STS 查找已经公布的引物序列Part four 如何运用 BLAST 进行序列比对、检验引物特异性特别感谢本版版主,将这个帖子置顶!从发帖到现在,很多战友对该帖给与了积极的关注,在此向给我投票的(以及想给我投票却暂时不能投票的)各位战友表示真诚的感谢,谢谢各位战友!请大家对以下我发表的内容提出自己的意见。
关于NCBI 其他方面的使用也请水平较高的战友给予补充First of all,还是让我们从查找基因序列开始。
第一部分利用Map viewer 查找基因序列、mRNA 序列、启动子(Promoter)下面以人的 IL6(白细胞介素 6)为例讲述一下具体的操作步骤1.打开Map viewer 页面,网址为:/mapview/index.html在 search 的下拉菜单里选择物种,for 后面填写你的目的基因。
操作完毕如图所示:2.点击“GO”出现如下页面:3.在步骤二图示的右下角有一个Quick Filter,下面是让你选择的几个复选框,在Gene前面的小方框里打勾,然后点击Filter. 出现下图:说明一下:1、染色体的红色区域即为你的目的基因所处位置。
2、下面参考序列给出了三个,是不同的部门做出来的,经我验证,序列有微小的差异,但总体来说基本相同。
尽管你分别点击后,序列代码、序列代码等有所差异,但碱基基本一致,不影响大家研究分析序列。

NCBI在线BLAST使用方法与结果详解BLAST(Basic Local Alignment Search Tool)是一套在蛋白质数据库或DNA数据库中进行相似性比较的分析工具。
BLAST程序能迅速与公开数据库进行相似性序列比较。
BLAST结果中的得分是对一种对相似性的统计说明。
BLAST 采用一种局部的算法获得两个序列中具有相似性的序列。
Blast中常用的程序介绍:1、BLASTP是蛋白序列到蛋白库中的一种查询。
库中存在的每条已知序列将逐一地同每条所查序列作一对一的序列比对。
2、BLASTX是核酸序列到蛋白库中的一种查询。
先将核酸序列翻译成蛋白序列(一条核酸序列会被翻译成可能的六条蛋白),再对每一条作一对一的蛋白序列比对。
3、BLASTN是核酸序列到核酸库中的一种查询。
库中存在的每条已知序列都将同所查序列作一对一地核酸序列比对。
4、TBLASTN是蛋白序列到核酸库中的一种查询。
与BLASTX相反,它是将库中的核酸序列翻译成蛋白序列,再同所查序列作蛋白与蛋白的比对。
5、TBLASTX是核酸序列到核酸库中的一种查询。
此种查询将库中的核酸序列和所查的核酸序列都翻译成蛋白(每条核酸序列会产生6条可能的蛋白序列),这样每次比对会产生36种比对阵列。
下面是具体操作方法1,进入在线BLAST界面,可以选择blast特定的物种(如人,小鼠,水稻等),也可以选择blast所有的核酸或蛋白序列。
不同的blast程序上面已经有了介绍。
这里以常用的核酸库作为例子。
2,粘贴fasta格式的序列。
选择一个要比对的数据库。
关于数据库的说明请看NCBI在线blast数据库的简要说明。
一般的话参数默认。
3,blast参数的设置。
注意显示的最大的结果数跟E值,E值是比较重要的。
筛选的标准。
最后会说明一下。
4,注意一下你输入的序列长度。
注意一下比对的数据库的说明。
5,blast结果的图形显示。
没啥好说的。
6,blast结果的描述区域。


N C B I在线B L A S T使用方法与结果详解BLAST(Basic Local Alignment Search Tool)是一套在蛋白质数据库或DNA数据库中进行相似性比较的分析工具。
BLAST程序能迅速与公开数据库进行相似性序列比较。
BLAST结果中的得分是对一种对相似性的统计说明。
BLAST 采用一种局部的算法获得两个序列中具有相似性的序列。
Blast中常用的程序介绍:1、3、4、5、NCBI1blast 2,粘贴blast 3,blast4,注意一下你输入的序列长度。
注意一下比对的数据库的说明。
5,blast结果的图形显示。
没啥好说的。
6,blast结果的描述区域。
注意分值与E值。
分值越大越靠前了,E值越小也是这样。
7,blast结果的详细比对结果。
注意比对到的序列长度。
评价一个blast结果的标准主要有三项,E值(Expect),一致性(Identities),缺失或插入(Gaps)。
加上长度的话,就有四个标准了。
如图中显示,比对到的序列长度为1405,看Identities这一值,才匹配到1344bp,而输入的序列长度也是为1344bp(看上面的图),就说明比对到的序列要长一点。
由Qurey(起始1)和Sbjct(起始35)的起始位置可知,5'端是是多了一段的。
有时也要注意3'端的。
附:来源于网络E值(Expect):表示随机匹配的可能性,E值越大,随机匹配的可能性也越大。
E值接近零或为零时,具本上就是完全匹配了。
一致性(Identities):或相似性。
匹配上的碱基数占总序列长的百分数。
缺失或插入(Gaps):插入或缺失。
用"—"来表示。
来源于网络。
NCBI在线BLAST使用方法与结果详解NCBI(National Center for Biotechnology Information)是一个包含大量基因组学、生物信息学等相关数据和工具的数据库。
其中,BLAST (Basic Local Alignment Search Tool)是一种常用的序列比对工具,可用于在数据库中搜索相似序列。
一、BLAST简介BLAST是一种基于序列比对的方法,可用于确定一给定序列与数据库中序列的相似性。
其工作原理是将查询序列与数据库中的序列进行比对,并生成一个比对得分来衡量它们之间的相似程度。
通过BLAST的结果,可以获得序列的匹配位置、长度、相似性等信息,从而帮助研究人员进行更深入的生物学研究。
二、使用方法1. 打开NCBI网站首先,打开浏览器,输入NCBI的网址(https:///),进入NCBI的官方网站。
2. 进入BLAST页面在NCBI的主页上,找到“BLAST”或“BLAST and Alignments”选项,并点击进入BLAST页面。
3. 输入查询序列在BLAST页面上,找到“Enter Query Sequence”或“Enter accession number, gi, or FASTA sequence”等文本框,将需要查询的序列输入其中。
可以直接复制粘贴序列,或选择上传文件的方式输入。
4. 选择数据库在BLAST页面上,找到“Choose Search Set”或“Database”等选项,选择需要比对的数据库。
NCBI提供了多个数据库,如“nr”(非冗余蛋白数据库)、“nt”(非冗余核酸数据库)等,根据研究需要选择合适的数据库。
5. 设置参数根据需要,可以通过“Algorithm parameters”等选项来设置比对参数,如设置匹配的阈值、比对的方式等。
6. 运行BLAST设置完成后,点击“BLAST”或“Run BLAST”等按钮运行BLAST。
1从NCBI上下载某个基因在其他物种的序列比如,下载caveolin基因在其他物种的序列NCBI地址:在search一栏的下拉列表中选择Nucleotide,for后面的一栏中输入自己要查询的基因。
完毕,点击GO确认。
可得到一下结果:每一条记录分别是某个物种的caveolin的序列,以第10条记录为例,称为GenBank 登录号。
为拉丁文的人类的字母,表示物种,表示基因名称(caveolin基因家族共有3个主要基因,分别称为1,2,3)表示此序列为cDNA,不含含子。
下图中的NEXT表示翻页,查看剩余的记录。
打开第10条记录可看到下图:现在你需要保存下来得就是上面的这一串(碱基)核酸序列。
复制黏贴(包括上面表示顺序的数字)到TXT文本中备用。
打开DNAMAN软件,左上角点击file-new,出现下图:可以把先前从NCBI下载的序列(保存到TXT文本中得)复制到箭头指示处,得到:并按照上图左上角file-save as(注意此文件得保存名称为保存的此物中得名称),已上是DNAMAN软件中seq序列格式的保存方法。
2 序列编辑和比对(DNAMAN软件)你们实验PCR得到的序列只是某个基因上的一部分,所以为了进行不同物种间的比对,要把下载下来的其他物种的某个基因的序列进行删减,以使两段基因是大约相同长度的片段进行比对。
以人类caveolin1基因为例说明一下。
按照1,2,3得顺序依次打开,得到下图:点击上图中的1,你会得到下图,点击2是清楚所有刚才选进比对的序列(为了重新选择序列),3是有选择的删除某个序列。
当然,把你的所有准备的序列保存好以后,从查找围这个下拉列表中寻找你要比对的序列。
可以按住ctrl点击你要比对的几个序列(同时选中)选完点击打开。
再点下图中得确定键。
得到下图:找好这两个物种重合的那个核苷酸的序号(前后两段都是),然后打开你保存的seq格式的序列,数出刚才比对重合部分的后端的碱基数,把这个碱基后面的序列删掉,再用此方法把比对重合部分前段得序列删掉,保存。
最近看到很多战友在论坛上询问如何查询基因序列、如何进行引物设计、如何使用BLAST 进行序列比对……,这些问题在 NCBI 上都可以方便的找到答案。
现在我就结合我自己使用 NCBI的一些经历(经验)跟大家交流一下 BCBI 的使用。
希望大家都能发表自己的使用心得,让我们共同进步!我分以下几个部分说一下 NCBI 的使用:Part one 如何查找基因序列、mRNA、PromoterPart two 如何查找连续的 mRNA、cDNA、蛋白序列Part three 运用 STS 查找已经公布的引物序列Part four 如何运用 BLAST 进行序列比对、检验引物特异性特别感谢本版版主,将这个帖子置顶!从发帖到现在,很多战友对该帖给与了积极的关注,在此向给我投票的(以及想给我投票却暂时不能投票的)各位战友表示真诚的感谢,谢谢各位战友!请大家对以下我发表的内容提出自己的意见。
关于NCBI 其他方面的使用也请水平较高的战友给予补充First of all,还是让我们从查找基因序列开始。
第一部分利用Map viewer 查找基因序列、mRNA 序列、启动子(Promoter)下面以人的 IL6(白细胞介素 6)为例讲述一下具体的操作步骤1.打开Map viewer 页面,网址为:/mapview/index.html 在 search 的下拉菜单里选择物种,for 后面填写你的目的基因。
操作完毕如图所示:2.点击“GO”出现如下页面:3.在步骤二图示的右下角有一个Quick Filter,下面是让你选择的几个复选框,在Gene 前面的小方框里打勾,然后点击Filter. 出现下图:说明一下:1、染色体的红色区域即为你的目的基因所处位置。
2、下面参考序列给出了三个,是不同的部门做出来的,经我验证,序列有微小的差异,但总体来说基本相同。
尽管你分别点击后,序列代码、序列代码等有所差异,但碱基基本一致,不影响大家研究分析序列。
(一)DNA序列比对分析一)两个DNA序列的相似性比对分析1.进入NCBI主页()2. 点击所有资源(All Resources)3.点击工具栏目(Tool)点击进入。
5. 找到Specialized BLAST,点击Align two (or more) sequences using BLAST (bl2seq)6. 将序列1复制、黏贴到Enter Query Sequence框中。
7. 将序列2复制、黏贴到Enter Subject Sequence框中。
8. 在Program Selection下面,选择Highly similar sequences (megablast) (高度相似序列)或其他分析程序9、点击BLAST按钮,进行比对分析。
10、观察、记录和分析两序列比对结果。
二)待查DNA序列与国际基因数据库中已有DNA序列的相似性比对分析。
1、进入NCBI主页()2、点击所有资源(all resources)3.点击工具栏目(Tool)点击进入。
5.在Basic BLAST下,找到nucleotide blast(Search a nucleotide database using a nucleotide query),点击进入。
6. 将DNA序列复制、粘贴到Enter Query Sequence下面的方框中。
7. 在Choose Search Set栏目下,选择others数据库8. 选择Highly similar sequences (megablast)或其他程序。
9. 按BLAST按钮。
10. 观察、记录和分析DNA序列与国际基因库已有序列的比对结果。
(二)DNA序列开放阅读框的分析1.进入NCBI主页()2.点击所有资源(all resources (A-Z) )3.点击工具栏目(Tool)4. 找到Open Reading Frame Finder (ORF Finder),点击进入。
NCBI序列比对方法与操作实例一、序列比对方法概述1. 序列比对的概念序列比对是指通过对两个或多个生物序列进行比较分析,找到它们之间的相似性和差异性。
序列比对是生物信息学中的重要工具之一,可以帮助研究人员理解DNA、RNA、蛋白质等生物分子的结构和功能,进而推动生物医药和生物科学领域的发展。
2. 序列比对的意义在生物学研究中,通过对不同生物序列进行比对分析,可以揭示它们之间的进化关系、基因结构、功能和调控机制等重要信息,有助于揭示生物系统的内在规律。
序列比对还可以在分子生物学实验设计、基因工程、疾病诊断、新药开发等方面发挥重要作用。
3. 序列比对的方法常用的序列比对方法包括全局比对、局部比对和多序列比对等,其中全局比对适用于寻找整个序列间的相似段,局部比对适用于寻找两个序列中的部分匹配段,多序列比对则适用于比较多个序列之间的相似性和差异性。
二、NCBI序列比对工具介绍1. NCBI数据库NCBI(National Center for Biotechnology Information)是美国国家生物技术信息中心,是全球生物学信息资源的重要提供者之一。
NCBI数据库中包含大量生物信息数据,包括基因组序列、蛋白质序列、原始文献、生物信息学工具等。
2. NCBI序列比对工具NCBI提供了一系列用于序列比对的工具,其中包括BLAST(Basic Local Alignment Search Tool)、BLAT(BLAST-Like Alignment Tool)、ClustalW、MAFFT等。
这些工具可以帮助研究人员进行序列比对分析,找到感兴趣的生物序列在数据库中的同源序列或相似序列。
三、NCBI序列比对操作实例以BLAST工具为例,介绍NCBI序列比对的操作步骤。
1. 打开NCBI全球信息湾打开NCBI全球信息湾(),在全球信息湾首页的搜索栏中输入“BLAST”,进入BLAST工具的页面。
2. 输入查询序列在BLAST工具的页面中,选择适当的数据库,粘贴或上传待比对的查询序列,可以选择标准蛋白数据库、EST数据库、基因组数据库等作为比对的对象。
序列比对及建树步骤1.以细菌、病毒或寄生虫为例,参考分类生物学资料,从GenBank中查询相关序列,详述Blast寻找、CLUSTAL比对、建树及种系发育过程以隐孢子虫actin基因为例做一叙述:1.1 Blast: 登录NCBI主页,打开Blast搜索引擎,将测得的一个已知的actin序列输入,下载了12条隐孢子虫序列,另外下载一条恶性疟原虫actin序列作为外群。
所获得的14条序列改为FAST格式,用TXT文件保存。
1.2 cluxtal 比对用软件clustalx1.83比对软件进行比对。
1.3 比对的精制对比对结果可以进行一些简单的调整,删去目的序列比对效果最差的开头和结尾部分。
可以用word文档打开比对所生成的aln.文件,在word文档下进行剪切。
然后将剪切的文档再用ClustalX软件进行比对,并生成Phylip格式文件。
1.4 使用Phylip软件建树以neighbour-jioning方法为例做一叙述。
1.4.1 先导树将生成的PHY文件(*.phy)拷贝到Phylip软件包目录下,最好修改成比较简单的文件名,比如修改成1或a等(比较方便下边的输入运行)。
运行DNADIST.EXE子软件,输入文件(比如1),打回车后弹出软件界面,打D可以选择不同的模型,在此选用Kimura 2-parameter模型。
生成的outfile文件可以再修改成简单的文件名,比如修改成2。
打开neighbor.exe子程序,输入文件2,打回车后运行完毕会生成两个文件,将文件outtree另存为.tre文件格式,即为所生成的先导树。
1.4.2 验证树1.4.2.1打开seqboot.exe输入文件名:输入你用CLASTAL X生成的PHY文件(*.phy)。
R为bootstrap的次数,一般为1000 (设你输入的值为M,即下两步DNADIST.EXE、NEIGHBOR.EXE中的M值也为1000)。
odd number: (4N+1)(eg: 1、5、9…)修改好了打回车y得到outfile(在phylip文件夹内)改名为21.4.2.2 打开Dnadist.EXE输入2修改M值,再按D,然后输入1000(M值)打回车y得到outfile(在phylip文件夹内)改名为31.4.2.3 打开Neighboor.EXE输入3M=1000(M值)打回车Y得到outfile和outtree(在phylip文件夹内),改outtree为4,outfile删除或另存1.4.2.4 打开consense.exe输入4打回车y得到outfile和outtree(在phylip文件夹内),Outfile可以改为*.txt文件,用记事本打开阅读或删除。