生物信息学作业1实验2
- 格式:doc
- 大小:37.00 KB
- 文档页数:2

实验1基因组序列组装(软件CAP3的使用)一、实验目的1.了解基因组测序原理和主要策略;2.掌握CAP3序列组装软件的使用方法。
二、实验原理基因组测序常用的两种策略是克隆法(clone-based strategy)和全基因组鸟枪法(whole genome shotgun method)。
克隆法先将基因组DNA打成大的片段,连到载体上,构建DNA文库;再对每一个大片段(克隆)打碎测序。
序列组装时先组装成克隆,再组装成染色体。
克隆测序法的好处在于序列组装时可以利用已经定位的大片段克隆, 所以序列组装起来较容易, 但是需要前期建立基因组物理图谱, 耗资大, 测序周期长。
全基因组鸟枪法测序无需构建各类复杂的物理图谱和遗传图谱,采用最经济有效的实验设计方案,直接将整个基因组打成不同大小的DNA片段构建Shotgun文库,再用传统Sanger测序法或Solexa等新一代测序技术对文库进行随机测序。
最后运用生物信息学方法将测序片段拼接成全基因组序列。
该方法具有高通量、低成本优势。
序列组装时,先把把单条序列(read)组装成叠连群(contig)、再把叠连群组装成“支架”(scaffold),最后组装成染色体。
本实验将练习在Linux环境下用CAP3软件组装流感病毒基因组。
1.CAP3序列组装程序简介Huang Xiaoqiu. 和 Madan,A. 开发的一套用于序列拼接的软件,此软件适用于小的数据集或 EST 拼接,它有如下特征:1. 应用正反向信息更正拼接错误、连接contigs。
2. 在序列拼接中应用 reads 的质量信息。
3. 自动截去 reads5`端、3`端的低质量区。
4. 产生 Consed 程序可读的ace 格式拼接结果文件。
5. CAP3 能用于Staden软件包的中的GAP4 软件。
2.下载此软件可以免费下载,下载地址:http:///download.html。
填写基本信息表格,即可下载。

生物信息学实验作业试验一一.找到编码拟南芥(arabidopsis)phyA(光敏色素A)基因的核酸序列编号, 并记录查找过程。
GI:224576211步骤1.进入NCBI主页2.搜索arabidopsis phyA3.Arabidopsis thaliana phytochrome A (PHYA) gene, partial cds4.VERSION:GI:224576211二.以phyA为检索词,在pubmed数据库中分别检索在题目和关键词字段中含有该检索词的文献,记录检索出的条目数目。
Results: 614三.仔细阅读所查询核酸序列在NCBI和EMBL数据库中格式的解释,理解各字段的含义,并比较NCBI 与EMBL中序列格式的异同。
实验二一.分析你感兴趣核酸序列的分子质量、碱基组成。
Composition 35 A; 25 C; 35 G; 15 T; 0 OTHERPercentage: 32% A; 23% C; 32% G; 14% T; 0%OTHERMolecular Weight (kDa): ssDNA: 34.26 dsDNA: 67.8二.列出你所分析核酸序列(或部分序列)的互补序列、反向序列、反向互补序列、DNA双链序列和RNA 序列。
R S1 ACTACTCGAG AAGCAGCGAC AGAGGCGTTA GCCCGCTCAG CAGACTGGCA GTTCTCTACC61 GACAAAAAAG AGGTAGGAGG CACAGTAATG ATACAGGCGT AGCAGGAGGGC S1 CCCTCCTGCT ACGCCTGTAT CATTACTGTG CCTCCTACCT CTTTTTTGTC GGTAGAGAAC61 TGCCAGTCTG CTGAGCGGGC TAACGCCTCT GTCGCTGCTT CTCGAGTAGTR C S1 TGATGAGCTC TTCGTCGCTG TCTCCGCAAT CGGGCGAGTC GTCTGACCGT CAAGAGATGG61 CTGTTTTTTC TCCATCCTCC GTGTCATTAC TATGTCCGCA TCGTCCTCCCD DNA S1 GGGAGGACGA TGCGGACATA GTAATGACAC GGAGGATGGA GAAAAAACAG CCATCTCTTGCCCTCCTGCT ACGCCTGTAT CATTACTGTG CCTCCTACCT CTTTTTTGTC GGTAGAGAAC61 ACGGTCAGAC GACTCGCCCG ATTGCGGAGA CAGCGACGAA GAGCTCATCATGCCAGTCTG CTGAGCGGGC TAACGCCTCT GTCGCTGCTT CTCGAGTAGTRNA S1 GGGAGGACGA UGCGGACAUA GUAAUGACAC GGAGGAUGGA GAAAAAACAG CCAUCUCUUG61 ACGGUCAGAC GACUCGCCCG AUUGCGGAGA CAGCGACGAA GAGCUCAUCA三.列出核酸序列的限制性酶切位点分析结果(酶及识别位点)。

实验一文献检索和浏览各大生物分子数据库一、实验目的1、学习文献检索方法2、了解生物信息学常用数据库的结构二、实验内容本实验通过登陆GenBank、EMBL、DDBJ三个国际上权威的核酸序列数据库、GDB基因组数据库、人类基因组数据库Ensembl、表达序列标记数据库dbEST、序列标记位点数据库dbSTS,以及PIR、SWISS-PROT、TrEMBL蛋白质序列数据库、蛋白质数据仓库UniProt、生物大分子数据库 PDB等,了解各数据库的结构,。
三、实验仪器、设备及材料计算机(联网)四、实验原理建立生物分子数据库的动因是由于生物分子数据的高速增长,而另一方面也是为了满足分子生物学及相关领域研究人员迅速获得最新实验数据的要求。
生物分子信息分析已经成为分子生物学研究必备的一种方法。
数据库及其相关的分析软件是生物信息学研究和应用的重要基础,也是分子生物学研究必备的工具。
国际上权威的核酸序列数据库有三个,分别是美国生物技术信息中心(NCBI)的GenBank (/web/Genbank/index/html)、欧洲分子生物学实验室的EMBL-Bank(简称EMBL,/embl/index/html)及日本遗传研究所的DDBJ (http://www.ddbj.nig.ac.jp/)。
三个数据库中的数据基本一致,仅在数据格式上有所差别,对于特定的查询,三个数据库的响应结果一样GDB(/)是一个出现较早的基因组数据库。
目前GDB包含对下述三种对象的描述:(1)人类基因组区域,包括基因、克隆、PCR标记物、断点、细胞遗传学标记、易碎位点、 EST、综合区域、contigs、重复等;(2)人类基因组图谱,包含细胞遗传学图谱、连接图谱、辐射混合图谱、contig 图谱、集成图谱,所有这些图谱都可以被直观地显示出来;(3)人类基因组中的变化,包括基因突变和基因多态性,加上等位基因频率数据。
Ensembl (/)是一个综合性基因组数据库,Ensembl包括所有公开的人类基因组DNA序列,通过注释形成的关于序列的特征。

生物信息学实验作业试验一一.找到编码拟南芥(arabidopsis)phyA(光敏色素A)基因的核酸序列编号, 并记录查找过程。
GI:224576211步骤1.进入NCBI主页2.搜索arabidopsis phyA3.Arabidopsis thaliana phytochrome A (PHYA) gene, partial cds4.VERSION:GI:224576211二.以phyA为检索词,在pubmed数据库中分别检索在题目和关键词字段中含有该检索词的文献,记录检索出的条目数目。
Results: 614三.仔细阅读所查询核酸序列在NCBI和EMBL数据库中格式的解释,理解各字段的含义,并比较NCBI 与EMBL中序列格式的异同。
实验二一.分析你感兴趣核酸序列的分子质量、碱基组成。
Composition 35 A; 25 C; 35 G; 15 T; 0 OTHERPercentage: 32% A; 23% C; 32% G; 14% T; 0%OTHERMolecular Weight (kDa): ssDNA: 34.26 dsDNA: 67.8二.列出你所分析核酸序列(或部分序列)的互补序列、反向序列、反向互补序列、DNA双链序列和RNA 序列。
R S1 ACTACTCGAG AAGCAGCGAC AGAGGCGTTA GCCCGCTCAG CAGACTGGCA GTTCTCTACC61 GACAAAAAAG AGGTAGGAGG CACAGTAATG ATACAGGCGT AGCAGGAGGGC S1 CCCTCCTGCT ACGCCTGTAT CATTACTGTG CCTCCTACCT CTTTTTTGTC GGTAGAGAAC61 TGCCAGTCTG CTGAGCGGGC TAACGCCTCT GTCGCTGCTT CTCGAGTAGTR C S1 TGATGAGCTC TTCGTCGCTG TCTCCGCAAT CGGGCGAGTC GTCTGACCGT CAAGAGATGG61 CTGTTTTTTC TCCATCCTCC GTGTCATTAC TATGTCCGCA TCGTCCTCCCD DNA S1 GGGAGGACGA TGCGGACATA GTAATGACAC GGAGGATGGA GAAAAAACAG CCATCTCTTGCCCTCCTGCT ACGCCTGTAT CATTACTGTG CCTCCTACCT CTTTTTTGTC GGTAGAGAAC61 ACGGTCAGAC GACTCGCCCG ATTGCGGAGA CAGCGACGAA GAGCTCATCATGCCAGTCTG CTGAGCGGGC TAACGCCTCT GTCGCTGCTT CTCGAGTAGTRNA S1 GGGAGGACGA UGCGGACAUA GUAAUGACAC GGAGGAUGGA GAAAAAACAG CCAUCUCUUG61 ACGGUCAGAC GACUCGCCCG AUUGCGGAGA CAGCGACGAA GAGCUCAUCA三.列出核酸序列的限制性酶切位点分析结果(酶及识别位点)。

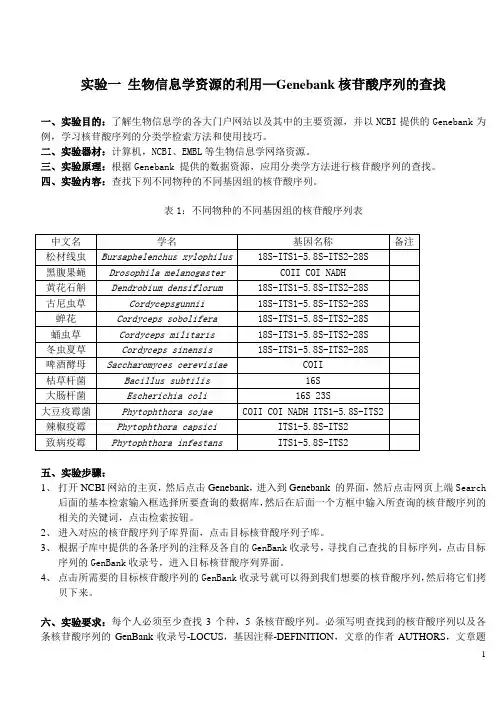
实验一生物信息学资源的利用—Genebank核苷酸序列的查找一、实验目的:了解生物信息学的各大门户网站以及其中的主要资源,并以NCBI提供的Genebank为例,学习核苷酸序列的分类学检索方法和使用技巧。
二、实验器材:计算机,NCBI、EMBL等生物信息学网络资源。
三、实验原理:根据Genebank 提供的数据资源,应用分类学方法进行核苷酸序列的查找。
四、实验内容:查找下列不同物种的不同基因组的核苷酸序列。
表1:不同物种的不同基因组的核苷酸序列表五、实验步骤:1、打开NCBI网站的主页,然后点击Genebank,进入到Genebank 的界面,然后点击网页上端Search后面的基本检索输入框选择所要查询的数据库,然后在后面一个方框中输入所查询的核苷酸序列的相关的关键词,点击检索按钮。
2、进入对应的核苷酸序列子库界面,点击目标核苷酸序列子库。
3、根据子库中提供的各条序列的注释及各自的GenBank收录号,寻找自己查找的目标序列,点击目标序列的GenBank收录号,进入目标核苷酸序列界面。
4、点击所需要的目标核苷酸序列的GenBank收录号就可以得到我们想要的核苷酸序列,然后将它们拷贝下来。
六、实验要求:每个人必须至少查找3个种,5条核苷酸序列。
必须写明查找到的核苷酸序列以及各条核苷酸序列的GenBank收录号-LOCUS,基因注释-DEFINITION,文章的作者AUTHORS,文章题目-TITLE,文章所发表的期刊-JOURNAL。
七、实验结果:查找的核苷酸序列基本情况表1LOCUS JN054403 894 bp DNA linear PLN01-NOV-2011DEFINITION Phytophthora melonis strain NN-1 18S ribosomal RNA gene, partial sequence; internal transcribed spacer 1, 5.8S ribosomal RNA gene, and internal transcribed spacer 2, complete sequence; and 28Sribosomal RNA gene, partial sequence.AUTHORS Wu,Y.G., Huang,S.L., Fu,G., Hu,C.J. and Lu,S.F.TITLE Identification of the causal agent of wax gourd blight in South ChinaJOURNAL UnpublishedORIGIN1 tgggattccc accctagaac tttccacgtg aaccgtatca acaagtagtt gggggcctgc 61 tctgtgtggc tagctgtcga tgtcaaagtc ggcgactggc tgctatgtgg cgggctctat 121 catggcgatt ggtttgggtc ctcctcgtgg ggaactggat catgagccca ccttttaaac 181 ccattcttga ttactgaata tactgtgggg acgaaagtct ctgcttttaa ctagatagca 241 actttcagca gtggatgtct aggctcgcac atcgatgaag aacgctgcga actgcgatac 301 gtaatgcgaa ttgcaggatt cagtgagtca tcgaaatttt gaacgcatat tgcacttccg 361 ggttagtcct gggagtatgc ctgtatcagt gtccgtacat caaacttggc tctcttcctt 421 ccgtgtagtc ggtggatgga gacgccagac gtgaggtgtc ttgcggcgcg gccttcgggc481 tgcctgcgag tcccttgaaa tgtactgaac tgtacttctc tttgctcgaa aagcgtgacg 541 ttgttggttg tggaggctgc ctgtatggcc agtcggcgac cagtttgtct gctgcggcgt 601 ttaatggagg agtgttcgat tcgcggtatg gttggcttcg gctgaacaat gcgcttattg 661 gatgcttttc ctgctgtggt ggtatgggct ggtgaaccgt agttgtgcga ggcttggctt 721 ttgaaccggc ggtgttgtag cgaagtagag tggcggcttc ggctgtcgag ggtcgatcca 781 tttgggaact ctgtgttgtc tctgcggctt gctgtggagg tagcatctca attggacctg 841 atatcaggca agattacccg ctgaacttaa gcatatcata aacgcggagg act2LOCUS HM596011 530 bp DNA linear PLN01-JUL-2011DEFINITION Ophiocordyceps sinensis culture-collection ARSEF:6282 clone C 18S ribosomal RNA gene, partial sequence; internal transcribed spacer 1, 5.8S ribosomal RNA gene, and internal transcribed spacer 2,complete sequence; and 28S ribosomal RNA gene, partial sequence. AUTHORS Chan,W.H.TITLE Direct SubmissionJOURNAL Submitted (28-JUN-2010) Depatment of Biology, The ChineseUniversity of Hong Kong, Shatin, Hong Kong 852, ChinaORIGIN1 tctccgttgg tgaaccagcg gagggatcat tatcgagtca ccactcccaa accccctgcg 61 aacaccacag cagttgcctc ggcgggaccg ccccggcgcc ccagggcccg gaccagggcg 121 cccgccggag gacccccaga ccctcctgtc gcagtggcat ctctcagtca agaagcaagc 181 aaatgaatca aaactttcaa caacggatct cttggttctg gcatcgatga agaacgcagc 241 gaaatgcgat aagtaatgtg aatcgcagaa ttcagtgaac catcgaatct ttgaacgcac 301 attgcgcccg ccagcactct ggcgggcatg cctgtccgag cgtcatctca accctcgagc 361 cccccgcctc gcggcggcgg ggcccggcct tgggggtcac ggccccgcgc cgccccctaa 421 acgcagtggc gaccccgccg cggctcccct gcgcagtagc tcgctgagaa cctcgcaccg 481 ggagcgcgga ggcggtcacg ccgtgaaacc accacaccct ccagttgacc3LOCUS HQ114254 711 bp DNA linear PLN31-AUG-2011DEFINITION Dendrobium densiflorum voucher PS2528MT01 18S ribosomal RNA gene, partial sequence; internal transcribed spacer 1, 5.8S ribosomal RNA gene, and internal transcribed spacer 2, complete sequence; and 28S ribosomal RNA gene, partial sequence.AUTHORS Yao,H., Gao,T. and Chen,S.-L.TITLE Direct SubmissionJOURNAL Submitted (10-AUG-2010) Institute of Medicinal Plant Development, Chinese Academy of Medical Sciences, Peking Union Medical College, No. 151 Malianwa North Road, Haidian District, Beijing 100193,ChinaORIGIN1 tttccgtagg tgaacctgcg gaaggatcat tgtcgagacc aaaataaatc gagcgatttg61 gagaaccggt caaaataagc ggtgattatt atttccgtga tgaacgccat cccagtcgtt121 acctcatccc cttagggtcg aggatgcgag taaggatgga tgaacactca agccggcgca181 gcatcgcgcc aagggaaata tcgaaacatg agcccttaaa tgggtttggt ggaatggggt241 gctgttgcac gccatatgga ttgacatgac tctcggcaat ggatatctcg gctcacgcat301 cgatgaagag cgcagcgaaa tgcgatacgt ggtgcgaatt gcagaatccc gcgaaccatc361 gagtctttga acgcaagttg cgcccgaggc caactggcca agggcacgtt tgcctgggcg421 tcaagcgtta tgtcgcttcg tgtcaactcc atcccgtcga tgtatgggct ggcgaaggct481 cggatgtgca gagtggctca tcgtgcccct cggtgcggtg agctgaagag cgggtcatca541 tctcgttggc tgcgaacgat aaggggtgga ttaaagcgag gcctatgtta ttgtgtcgtg601 tatgcccgag agaagattat acatactcag gagatcccaa atcatgcgtc gatcaaagga661 tggcgcttgg aatgcgaccc caggatgggc gaggccaccc gctgagttta a4LOCUS AJ966733 585 bp DNA linear PLN11-APR-2008DEFINITION Saccharomyces sp. CECT 11011 mitochondrial partial COII gene forcytochrome c oxidase, subunit II.AUTHORS Gonzalez,S.S., Barrio,E. and Querol,A.TITLE Molecular characterization of new natural hybrids of Saccharomyces cerevisiae and S. kudriavzevii in brewingJOURNAL Appl. Environ. Microbiol. 74 (8), 2314-2320 (2008)ORIGIN1 aatattatgt tttatttatt agttatttta ggtttagtat cttgaatgtt atatactatt61 gtaataacat attcaaaaaa ccctattgct tataaatata ttaaacatgg acaaactatt121 gaagttattt gaacaatttt cccagcagta gtattattaa ttattgcttt cccatcattt181 attttattat atttatgtga tgaagttatt tcaccagcta taactattaa agctattgga241 tatcaatgat attgaaaata tgaatattct gattttatta atgatagtgg tgaaactgtt301 gaatttgaat catatgttat tcctgatgaa ttattagaag aaggtcaatt aagattatta361 gatactgata cttctatagt tgtacctgta gatacacata ttagatttgt tgtaacagct421 gctgatgtta ttcatgattt cgctatccca agtttaggta ttaaagttga tgctactcct481 ggtagattaa atcaagtttc tgctttaatt caaagagaag gtgttttcta tgggcaatgc541 tcagagttgt gcgggctggg acatgccaac ataccaatta aaatt5LOCUS Y09069 459 bp mRNA linear INV18-APR-2005DEFINITION D.melanogaster mRNA for NADH-ubiquinone oxidoreductase acyl-carrier subunit, splice variant.AUTHORS Ragone,G., Caizzi,R., Moschetti,R., Barsanti,P., De Pinto,V. and Caggese,C.TITLE The Drosophila melanogaster gene for the NADH:ubiquinoneoxidoreductase acyl carrier protein: developmental expressionanalysis and evidence for alternatively spliced formsJOURNAL Mol. Gen. Genet. 261 (4-5), 690-697 (1999)ORIGIN1 atgtcgttca cacagatcgc gcgcagctgc agtcgactgg cggccacttt ggccccaagg61 agggtcgcct ccggcattct catccaatca caggcctcca ggatgatgca caggatcgcc121 gtgccatcga tgaccagcca gttgagccaa gagtgccgtg gtcgctggca aacgcaattg181 gtgcgcaaat actcggcgaa accgccgctc tcgctgaagc tgatcaatga gcgcgtcttg241 cttgtgctca agctctacga caagatcgat cccagcaagc tcaacgttga gtcgcacttc301 atcaacgact tgggactgga ttccttggac cacgtggagg tcatcatggc catggaggac361 gagttcggtt tcgagatccc cgactctgat gccgagaagc tgcttaaacc tgccgacatt421 attaagtacg tcgccgacaa ggaggatgtg tacgagtaa实验二序列相似性搜索软件—BLAST的使用一、实验目的:掌握序列相似性查询工具—BLAST使用方法和技巧,理解与序列相似性查询相关的几个基本概念。

生物信息学实验一简介:生物信息学实验一是生物信息学实验课程的第一部分,旨在介绍生物信息学的基本概念、工具和技术,以及生物信息学在生物学研究中的应用。
本实验将引导学生通过实际操作,学习并掌握生物信息学的基本原理和操作技巧。
实验设备和材料:- 计算机或笔记本电脑- 生物信息学软件(例如NCBI BLAST、UCSC Genome Browser等)- 相关数据库和工具(例如GenBank、KEGG等)实验目的:1. 了解生物信息学的基本概念和应用领域;2. 学习生物信息学的常用工具和技术;3. 掌握生物序列分析、基因注释和比对等基本操作;4. 学会使用生物信息学软件和数据库进行数据查询和分析;5. 培养科学研究的数据处理和解读能力。
实验步骤:1. 确定研究对象:选择一个感兴趣的生物学问题或基因序列进行研究。
2. 数据获取:使用生物信息学工具和数据库,获取与研究对象相关的生物序列数据。
3. 序列分析:使用生物信息学软件对序列数据进行分析,包括碱基组成、氨基酸序列、启动子分析等。
4. 基因注释:通过比对算法和数据库,对序列进行基因功能注释,确定基因的命名、结构和功能信息。
5. 比对分析:使用比对工具进行序列比对,比较两个或多个序列之间的相似性和差异性。
6. 数据解读:根据分析结果,结合相关文献和知识,对实验数据进行解读和分析,得出科学结论。
实验注意事项:1. 在进行实验前,先了解所要使用的工具和软件的基本操作方法和原理;2. 实验过程中注意数据安全和保密,不得将数据泄露或用于非科研目的;3. 在进行数据分析和解读时,务必准确、客观地进行,不得造假或歪曲实验结果;4. 注意数据的备份和存储,以防止数据丢失或损坏;5. 尊重他人的研究成果和知识产权,合理引用和参考相关文献。
实验结果与讨论:本实验所得的结果可以根据具体的研究对象和实验数据来展开讨论和分析。
例如,如果研究对象是某个基因序列,可以讨论其结构和功能,与其他基因的关联性,以及在哪些生物过程中有重要作用等。

生物信息学实验讲义广东药学院生命科学与生物制药学院二○一一年三月目录实验1. 生物信息学数据库与软件搜索 (1)实验2.核酸序列的检索 (2)实验3. 核酸序列分析 (3)实验4.多重序列比对及系统发生树的构建 (5)实验5. PCR 引物设计及评价 (7)实验6.蛋白质序列分析和结构预测 (9)实验一生物信息学数据库和软件的搜索【实验目的】熟练掌握上网搜索生物信息学数据库和软件的方法及技能。
【实验内容】1、搜索生物信息学数据库或者软件数据库是生物信息学的主要内容,各种数据库几乎覆盖了生命科学的各个领域。
核酸序列数据库有GenBank, EMBL, DDB等,蛋白质序列数据库有SWISS-PROT, PIR, OWL, NRL3D, TrEMBL等,蛋白质片段数据库有PROSITE, BLOCKS, PRINTS等,三维结构数据库有PDB, NDB, BioMagResBank, CCSD等,与蛋白质结构有关的数据库还有SCOP, CATH, FSSP, 3D-ALI, DSSP等,与基因组有关的数据库还有ESTdb, OMIM, GDB, GSDB等,文献数据库有Medline, Uncover等。
另外一些公司还开发了商业数据库,如MDL等。
生物信息学数据库覆盖面广,分布分散且格式不统一, 因此一些生物计算中心将多个数据库整合在一起提供综合服务,如EBI的SRS(Sequence Retrieval System)包含了核酸序列库、蛋白质序列库,三维结构库等30多个数据库及CLUSTALW、PROSITESEARCH等强有力的搜索工具,用户可以进行多个数据库的多种查询。
2、搜索生物信息学软件生物信息学软件的主要功能有:分析和处理实验数据和公共数据,加快研究进度,缩短科研时间;提示、指导、替代实验操作,利用对实验数据的分析所得的结论设计下一阶段的实验;寻找、预测新基因及预测其结构、功能;蛋白高级结构预测。

(一)生物信息学数据库实验目的:了解生物信息学的各大门户网站,了解数据库的内容及结构,理解各数据库注释的含义。
1、分别读取人CDK4的核酸序列及蛋白质序列,保存FASTA格式序列,熟悉数据库记录的flatfile格式,看懂其中的注释。
在NCBI数据库中读取人CDK4的核酸序列,步骤入下:(1)选择核酸(Nucleotide)将CDK4输入搜索栏中,点击Search。
(2)在Top Organisms中选择人(Homo sapients)(3)在数据库出现的数据中选择合适的核酸序列,选择FASTA可以使序列以FASTA 的格式显示出来。
GenBank形式则显示该序列的详细信息。
(4)保存的FASTA格式序列如下>gi|345525417|ref|NM_000075.3| Homo sapiens cyclin-dependent kinase 4 (CDK4), mRNACACCTCCTGTCCGCCCCTCAGCGCATGGGTGGCGGTCACGTGCCCAGAACGTCCGGCGTTCGCCCCG CCCTCCCAGTTTCCGCGCGCCTCTTTGGCAGCTGGTCACATGGTGAGGGTGGGGGTGAGGGGGCCTCTCTAG CTTGCGGCCTGTGTCTATGGTCGGGCCCTCTGCGTCCAGCTGCTCCGGACCGAGCTCGGGTGTATGGG(5) 在NCBI数据库中读取人CDK4的蛋白质序列,步骤入下:选择蛋白质(Protein)将CDK4输入搜索栏中,点击Search。
选择CDK4[Homo sapiens]的FASTA格式2、2BXI练习使用Jmol浏览蛋白质的三维结构。
()先进入PDB,再查看。
无法访问此网站3、练习使用Pubmed文献数据库(1)Pubmed检索运算符逻辑与:AND;逻辑或:OR;逻辑非:NOT。
注:当当一个检索表达式中同时含有三个运算符时,运算顺序从左至右,括号可以改变运算顺序。

实验二在NCBI上进行Blast比对、查询实验目的:熟悉序列比对的数学基础,掌握在NCBI网页上进行BLAST比对、查询技能。
设备、软件:装有WinXP、Win2k或Win7操作系统的计算机,同时要求装有两个网页浏览器(IE8、360极速浏览器)。
实验内容:在应用方面,BLAST分为三个方向,BLAST Assemble Genomes(在指定的基因组里鉴定同源基因,从而在基因组上实现定位),Basic BLAST(常规BLAST,即在数据库里搜索亲缘性的序列)和Specialized BLAST(对DNA、蛋白质的序列进行特殊BLAST,以期获得特殊的结构域、引物、抗体、SNP、表达谱、转录谱等),在这三组BLAST中,最常用的是Basic BLAST,它也是实现咨询序列与数据库中所有序列比较的BLAST。
通过简单的BLAST练习两条短序列的比对,熟悉两条字符串比对的原理;通过提交序列在数据库中进行BLAST在线比对,掌握在NCBI网页上进行BLAST比对、查询功能。
实验步骤:一、两条序列的比对1、先将如下两条序列进行FASTA格式处理CTGTGCGGA TTCTTGTGGCTTTGGCCCTATCTTTTCTATGTCCAAGCTGTGCCCATCCAA和CTGTGCGGATTCTTGTGGCTTTGGCCCTATCTTTTCTATGTCCAAGCTGTGCCCATCCAA AA 处理后为:>123CTGTGCGGA TTCTTGTGGCTTTGGCCCTATCTTTTCTATGTCCAAGCTGTGCCCATCCAA >456CTGTGCGGA TTCTTGTGGCTTTGGCCCTATCTTTTCTATGTCCAAGCTGTGCCCATCCAA AA2、打开IE,进入/,后打开BLAST链接(在主页的右上角的popular resources区域的第一个即是),或直接进入BLAST页面(/)。

生物信息学实验报告班级::学号:日期:实验一核酸和蛋白质序列数据的使用实验目的了解常用的序列数据库,掌握基本的序列数据信息的查询方法。
教学基本要求了解和熟悉NCBI 核酸和蛋白质序列数据库,可以使用BLAST进行序列搜索,解读BLAST 搜索结果,可以利用PHI-BLAST 等工具进行蛋白质序列的结构域搜索,解读蛋白质序列信息,可以在蛋白质三维数据库中查询相关结构信息并进行显示。
实验容提要在序列数据库中查找某条基因序列(BRCA1),通过相关一系列数据库的搜索、比对与结果解释,回答以下问题:1. 该基因的基本功能?2. 编码的蛋白质序列是怎样的?3. 该蛋白质有没有保守的功能结构域 (NCBI CD-search)?4. 该蛋白质的功能是怎样的?5. 该蛋白质的三级结构是什么?如果没有的话,和它最相似的同源物的结构是什么样子的?给出示意图。
实验结果及结论1. 该基因的基本功能?This gene encodes a nuclear phosphoprotein that plays a role in maintaining genomic stability, and it also acts as a tumor suppressor. The encoded protein combines with other tumor suppressors, DNA damagesensors, and signal transducers to form a large multi-subunit protein complex known as the BRCA1-associated genome surveillance complex (BASC). This gene product associates with RNA polymerase II, and through the C-terminal domain, also interacts with histone deacetylase complexes. This protein thus plays a role in transcription, DNA repair of double-stranded breaks, and recombination. Mutations in this gene are responsible for approximately 40% of inherited breast cancers and more than 80% of inherited breast and ovarian cancers. Alternative splicing plays a role in modulating the subcellular localization and physiological function of this gene. Many alternatively spliced transcript variants, some of which are disease-associated mutations, have been described for this gene, but the full-length natures of only some of these variants has been described. A related pseudogene, which is also located on chromosome 17, has been identified. [provided by RefSeq, May 2009]2. 编码的蛋白质序列是怎样的?[Homo sapiens]1 mdlsalrvee vqnvinamqk ilecpiclel ikepvstkcd hifckfcmlk llnqkkgpsq61 cplcknditk rslqestrfs qlveellkii cafqldtgle yansynfakk ennspehlkd121 evsiiqsmgy rnrakrllqs epenpslqet slsvqlsnlg tvrtlrtkqr iqpqktsvyi181 elgsdssedt vnkatycsvg dqellqitpq gtrdeislds akkaacefse tdvtntehhq241 psnndlntte kraaerhpek yqgssvsnlh vepcgtntha sslqhenssl lltkdrmnve301 kaefcnkskq pglarsqhnr wagsketcnd rrtpstekkv dlnadplcer kewnkqklpc361 senprdtedv pwitlnssiq kvnewfsrsd ellgsddshd gesesnakva dvldvlnevd421 eysgssekid llasdpheal ickservhsk svesniedki fgktyrkkas lpnlshvten481 liigafvtep qiiqerpltn klkrkrrpts glhpedfikk adlavqktpe minqgtnqte541 qngqvmnitn sghenktkgd siqneknpnp ieslekesaf ktkaepisss isnmelelni601 hnskapkknr lrrksstrhi halelvvsrn lsppnctelq idscssseei kkkkynqmpv661 rhsrnlqlme gkepatgakk snkpneqtsk rhdsdtfpel kltnapgsft kcsntselke721 fvnpslpree keekletvkv snnaedpkdl mlsgervlqt ersvesssis lvpgtdygtq781 esisllevst lgkaktepnk cvsqcaafen pkglihgcsk dnrndtegfk yplghevnhs 841 retsiemees eldaqylqnt fkvskrqsfa pfsnpgnaee ecatfsahsg slkkqspkvt 901 feceqkeenq gknesnikpv qtvnitagfp vvgqkdkpvd nakcsikggs rfclssqfrg 961 netglitpnk hgllqnpyri pplfpiksfv ktkckknlle enfeehsmsp eremgnenip 1021 stvstisrnn irenvfkeas ssninevgss tnevgssine igssdeniqa elgrnrgpkl 1081 namlrlgvlq pevykqslpg snckhpeikk qeyeevvqtv ntdfspylis dnleqpmgss 1141 hasqvcsetp ddllddgeik edtsfaendi kessavfsks vqkgelsrsp spfththlaq 1201 gyrrgakkle sseenlssed eelpcfqhll fgkvnnipsq strhstvate clsknteenl 1261 lslknslndc snqvilakas qehhlseetk csaslfssqc seledltant ntqdpfligs 1321 skqmrhqses qgvglsdkel vsddeergtg leennqeeqs mdsnlgeaas gcesetsvse 1381 dcsglssqsd ilttqqrdtm qhnliklqqe maeleavleq hgsqpsnsyp siisdssale 1441 dlrnpeqsts ekavltsqks seypisqnpe glsadkfevs adsstsknke pgversspsk 1501 cpslddrwym hscsgslqnr nypsqeelik vvdveeqqle esgphdltet sylprqdleg 1561 tpylesgisl fsddpesdps edrapesarv gnipsstsal kvpqlkvaes aqspaaahtt 1621 dtagynamee svsrekpelt astervnkrm smvvsgltpe efmlvykfar khhitltnli 1681 teetthvvmk tdaefvcert lkyflgiagg kwvvsyfwvt qsikerkmln ehdfevrgdv 1741 vngrnhqgpk raresqdrki frgleiccyg pftnmptdql ewmvqlcgas vvkelssftl 1801 gtgvhpivvv qpdawtedng fhaigqmcea pvvtrewvld svalyqcqel dtylipqiph 1861 shy3. 该蛋白质有没有保守的功能结构域 (NCBI CD-search)?有保守的供能结构域。
生物信息学实验一生物信息学实验一: DNA序列比对一、引言DNA序列比对是生物信息学中的基础操作之一。
DNA序列比对可以通过比较两个或多个DNA序列之间的相似性和差异性,进而揭示序列之间的进化关系、基因功能以及潜在的生物学意义。
本实验旨在介绍DNA序列比对的基本原理、常见比对工具以及实验操作步骤。
二、实验原理1. 基本原理DNA序列比对是指将两个或多个DNA序列在相同参考框架下进行对比,以确定序列之间的相似性和差异性。
基于比对结果,可以推断序列中的保守区域、突变位点、插入缺失等信息。
2. 比对方法常见的DNA序列比对方法包括全局比对和局部比对。
全局比对适用于两个序列长度相似且整体结构相似的情况,例如比对同一基因的两个亚型。
而局部比对适用于两个序列之间存在较大差异的情况,例如比对基因组中的编码区域。
3. 比对工具生物信息学领域中有许多常用的DNA序列比对工具,如BLAST (Basic Local Alignment Search Tool)、ClustalW和MUSCLE等。
每个工具都有其独特的优势和适用范围,根据具体的研究目的和样本特点选择合适的比对工具。
三、实验步骤1. 收集序列数据在进行DNA序列比对实验前,首先需要收集待比对的DNA序列数据。
可以从公共数据库(如GenBank)或实验室已有的数据中获取所需序列,并保存为FASTA格式。
2. 选择比对工具根据比对的目的和序列特点,选择合适的比对工具。
例如,对于全局比对,可以选用BLAST工具;对于局部比对,可以选择ClustalW或MUSCLE工具。
3. 导入序列数据将收集到的DNA序列导入所选择的比对工具中。
一般来说,比对工具能够接受FASTA格式的输入。
确保正确导入所有待比对的序列,并设置比对参数。
4. 进行比对运行选定的比对工具,开始进行DNA序列比对。
比对过程可能需要花费一定的时间,具体时间取决于比对工具的算法和序列的长度。
5. 分析比对结果比对完成后,可以获取比对结果。
研究生《医学生物信息学》作业班级:专业:姓名:一、实验目的:(1)掌握中文文献全文的检索和获得方法。
(2)掌握Pubmed数据库文献的检索和交大图书馆英文数据库全文的获得方法。
(3)掌握核酸序列搜索的方法。
(4)掌握核酸序列相似性分析的方法。
(5)掌握PCR引物设计软件的原理、使用及特点。
(6)掌握蛋白质序列搜索的方法。
(7)掌握蛋白质序列分析常用软件的使用方法。
二、研究背景:AIB1基因为近年来发现的p160类固醇受体转录共激活因子SRC-1家族成员,是新定义的一个原癌基因[1]。
该基因表达的蛋白在许多生物学过程中发挥重要作用,如细胞生长,增殖,分化,性成熟,女性生殖功能等[2]。
近年发现,该基因的表达异常与多种肿瘤的发生发展有关,以在乳腺癌中研究最多。
AIB1基因的高表达与乳腺癌的发生和发展有关[3]。
AIB1蛋白通过与雌激素受体相互作用,能强烈地增强雌激素受体的促进靶基因转录的效应,进而引起细胞增殖和肿瘤形成,此外,AIB1蛋白还在多条信号传导通路中发挥作用[4]。
AIB1基因(amplified in breast cancer1)又称为ACTR,TRAM1,RAC3,SRC3,NCoA3,P/CIP等。
本人选择其为研究对象。
三、实验方法、步骤及结果:1.在中国知网(CNKI)中查找中文文献:2.在PubMed中查找英文文献:登陆NCBI主页,网址:/guide/,选择gene数据库4. 使用NCBI网站中的BLAST工具进行序列比对登陆/,选择核酸序列比对nucleotide BLAST,界面显示如下,输入登录号,AF012108,点击“BLAST”。
结果如下:共有2条核苷酸序列和2条基因组序列和其匹配:第一条核苷酸序列为“Homo sapiens nuclear receptor coactivator 3 (NCOA3), transcript variant 2, mRNA”,登录号:NM_006534。
广州大学学生实验报告开课学院及实验室:生命科学学院计机楼407 2013年11月 29日1页MEPKRIREGYLVKRGSVFNTWKPMWVVLLEDGIEFYKKKSDNSPKGMIPLKGSTLTSPCQDFGKRMFVFK ITTTKQQDHFFQAAFLEERDSWVRDTKKAIKCIEGGQKFARKSTRRSIRLPETVDLGALYLSMKDIEKGI>MOUSEMEPKRIREGYLVKKGSVFNTWKPMWVVLLEDGIEFYKKKSDNSPKGMIPLKGSTLTSPCQDFGKRMFVLK ITTTKQQDHFFQAAFLEERDAWVRDIKKAIKCIEGGQKFARKSTRRSIRLPETIDLGALYLSMKDPEKGI>CanisMEPKRIREGYLVKRGSVFNTWKPMWVVLLEDGIEFYKKKSDNSPKGMIPLKGSTLTSPCQDFGKRMFVFK ITTTKQQDHFFQAAFLEERDSWVRDTKKAIKCIEGGQKFARKSTRRSIRLPETVDLGALYLSMKDIEKGI>Gallus gallusMEREPMRIREGYLVKKGSMFNTWKPMWVVLLEDGIEFYKRKSDNSPKGMIPLKGSTINSPCQDFGKRMFV FKLTAAKQQDHFFQASYLEERDAWVRDIKKAIQCIDGGQRFARKSTRKSIRLPETINLSALYLSMKDPEK>Danio rerioMEPTTIREGYLVKKGTVLNSWKAVWVVLKDDAIEFFKKKTDRNAKGMIPLKGATLTSPCQDFSKRALVFK VSTAKNQDHYFQATHLEEREHWVKDIRRAITCLQGGKKFARKSTRRSIRLPESVNLSELYVCMKDPDRGV>chimpanzeeMEPKRIREGYLVKRGSVFNTWKPMWVVLLEDGIEFYKKKSDNSPKGMIPLKGSTLTSPCQDFGKRMFVFK ITTTKQQDHFFQAAFLEERDAWVRDMKKAIKCIEGGQKFARKSTRRSIRLPETIDLGALYLSMKDTEKGI3、以上构建系统进化树的方法为N-J法,请总结采用蛋白质序列构建系统进化树与采用DNA序列构建系统进化树所选用的程序的区别。
实验一计算机网上操作基本技能训练学时数:3目的要求:1、掌握windows常用操作2、熟练掌握上网操作基本方法及技能。
3、掌握利用网络进行资料搜集的多种方法实验内容:一、windows xp/2000的操作1、熟悉界面以及内含的各种窗口2、进行文件夹以及文件操作3、应用程序的应用4、进行图文编排5、网络配置6、自我安装windows 2000作业:1、在D盘建立一个以自己名字命名的文件夹2、在Word中创建一个如何进行网络配置以及安装windows xp/2000的文档,并以恰当文件名保存到自己的文件夹中二、熟练掌握上网操作基本方法及技能1、熟悉Internet Exporer 的基本使用方法及相关技巧,熟悉Internet Exporer网络配置。
2、利用outlook express或其它邮件软件收发E-mail,掌握免费电子邮箱的申请方法,并且,能收发电子邮件。
3、掌握网上软件下载及安装方法。
4、掌握WINDOWS 系统的升级方法和安装安全补丁5、用IE或netscape等浏览工具浏览、搜索各类信息6、运用FlashGet 或网络蚂蚁等下载工具进行网络资料的下载以及运用各种上传工具上传资料到网络7、利用Winzip或Winrar等压缩工具进行文件的压缩与解压8、学习使用Telnet、ftp9、在网上自主学习了解与生物信息学相关的计算机语言,如C语言、Perl语言作业:1、请一个自已的免费电子邮箱,并发一封电子邮件到master@2、从网络上下载任意一个软件,并安装到计算机上。
3、用WINDOWS UPDATE 升级和打补丁。
4、用FTP获取一个蛋白质结构分析软件比如rasmol,下载后保存到你的文件夹中,运用其进行蛋白质结构分析。
5、载一个有关C语言的教程,并保存到你的文件夹中,进行参考学习。
生物信息学实验二目的要求1.掌握序列检索的操作方法;2.熟悉GenBank数据库序列格式及其主要字段的含义;3.了解EBML数据库序列格式及其主要字段的含义;4.熟悉GenBank数据库序列格式的FASTA序列格式显示与保存;5.熟悉分子生物学软件的搜索与下载。
生物信息学实验实验一生物信息数据库的检索一.实验目的:1.了解生物信息学的各大门户网站以及其中的主要资源。
2.了解主要数据库的内容及结构,理解各数据库注释的含义。
3.以PubMed为例,学会文献数据库的基本查询检索方法。
二.实验内容:(1)国际与国内的生物信息中心国际NCBI、EBI、ExPASy,EMBL、SIB、TIGR以及国内CBI、BioSino网站的熟悉及内容的了解。
核酸序列数据库:genbank/EMBL-bank/DDBJNCBI网址:/EBI网址:/EMBL网址:/embl蛋白质序列数据库:Swiss Prot 、ExPASy网址:/Uniprot网址:/蛋白质结构数据库:PDB网址:/pdb/(2)数据库内容、结构与注释的浏览分别读取The spike protein of SARS-Corona Virus在NCBI中的核酸序列、SWISS-PROT蛋白质序列以及PDB蛋白质结构序列,熟悉数据库记录的结构,学会看懂其中的注释。
核酸序列:SWISS-PROT蛋白质序列:PDB蛋白质结构序列:其PDB文件见附件SARS-Corona Virus.PDB文件分别读取Heamagglutinin Genes of H9N2 Subtype Influenza A Viruses(禽流感H9N2亚型HA基因)在NCBI中的核酸序列、SWISS-PROT蛋白质序列以及PDB蛋白质结构序列,熟悉数据库记录的结构,学会看懂其中的注释。
核酸序列:SWISS-PROT蛋白质序列PDB蛋白质结构序列其PDB文件见附件H9N2.PDB文件(3)文献信息的查找与管理有效地使用NCBI PubMed提供的各种主要功能,查询并下载相关课题或研究方向的论文文摘与文献全文。
查询Influenza A Viruses分子进化研究方向的文章。
三.实验要求:(1)以其中的一个信息中心网站为例,列举其中的主要资源(数据库、网上分析、生物计算、数据下载等)。
生物信息学实验实验一生物信息数据库的使用一、实验目的了解NCBI、EMBL、SWISS-PROT、PDB数据库的结构掌握NCBI、EMBL数据库检索系统ENTREZ、SRS、CN3D的操作方法,掌握文献、序列的快速高效检索方法FASTA的操作方法一、实验目的掌握GenBank数据库序列格式及其主要字段的含义了解EBML数据库序列格式及其主要字段的含义掌握GenBank数据库序列格式的FASTA序列格式显示与保存二、实验内容及操作步骤内容一:登陆NCBI、EMBL、SWISS-PROT、PDB数据库主页,打开数据库的SITE MAP页面,了解各网站的结构网址:NCBI: EMBL: SWISS-PROT: /sprot/PDB: /pdb/FASTA3 :/fasta33/index.html二、实验内容及操作步骤内容二:使用Entrez信息查询系统检索与禽流感相关的文献,并阅读感兴趣文献的摘要或全文调用Internet浏览器并在其地址栏输入Entrez网址(/Entrez)进入Entrez 主页选择pubmed文献数据库→在输入栏内输入关键词“Avian Influenza”→点击go查询练习使用AND OR BUT逻辑词来限定关键词,如Avian Influenza AND human infect等查询人感染禽流感的相关记录,比较查询结果二、实验内容及操作步骤内容三:使用Entrez信息查询系统检索与禽流感相关的核酸序列,链接提取其中一条感兴趣的序列内容,阅读序列格式的解释,理解其含义二、实验内容及操作步骤进入Entrez Home页面→选择Nucleotide数据库→在Search后的输入栏中选择Nucleotide→在输入栏内输入关键词Avian Influenza→点击go查询选择一条感兴趣的核酸序列,点击该序列与数据库的超链接,阅读序列格式的解释,理解其含义,如LOCUS、DEFINITION、ACCESSION等二、实验内容及操作步骤3. 在NCBI上找苯丙氨酸解氨酶基因序列1,打开NCBI,选择核苷酸(Nucleotide)数据库,填上Phenylalanine ammonia-lyase,点击GO,搜索二、实验内容及操作步骤2、我们来看结果,总共有1022个,结果太多二、实验内容及操作步骤3、这个时候我们可以再想办法缩少范围,比方你要找的是豆科的,我们来大豆(soybean)来作例子。
上海师范大学实验报告
实验二
一、实验原理
答:利用Blast全球联网数据库,对输入的序列进行生物信息学分析,给出与输入序列相关性最大的对应的基因信息,比较两者的同源性。
二、操作步骤
答:(1)先打开网址/
(2)点击右边的Blast链接,打开Blast数据库,进入Blast界面
(3)在Basic Blast中选择nucleotide blast
(4)在对话框中输入核苷酸序列,在choose search set下的Database选项中选择Others (nr etc.)
(5)把网页拉到最下方,点击Blast按钮
(6)在Descriptions 栏下找到Max ident 百分率最高的序列名称
(7)再往下拉,找到Alignments项下第一个序列,可以找到输入序列相关信息
(8)点击Accession,即能找到更多输入序列的相关信息。
1.
tttcactcca tagttactcc ccaggtga
1.1它属于哪类生物?
答:属于Hepatitis C virus (丙型肝炎病毒)
1.2它属于哪类基因?
答:属于non-structural protein 5B gene
1.3它在该基因的什么位置?
答:它在该基因的第749-776这个位置。
1.4它与你搜索到的序列的同源性(Identities)是多少?
答:同源性100%
2.(1)ccacccactg aaactgcaca gacaaatttg tacataagag
1.1它属于哪类生物?
答:属于Influenza A virus (A/chicken/Iran261/01(H9N2)) hemagglutinin (HA) gene (A型流感病毒,A型伊朗型261鸡流感病毒,H9N2病毒,血细胞凝集素抗原基因为依据)
1.2它属于哪类基因?
答:属于ssRNA negative-strand viruses Orthomyxoviridae (单链RNA,负义链病毒,正粘病毒科)
1.3它在该基因的什么位置?
答:它在该基因的第1-40这个位置
1.4它与你搜索到的序列的同源性(Identities)是多少?
答:100%
attgaacgct ggcggcaggc ctaacacatg caagtcgagc ggatgaagag agcttgctct
ctgattcagc ggcggacggg tgagtaatgc ctaggaatct gcctggtagt gggggacaac
gtttcgaaag gaacgctaat accgcatacg tcctacggga gaaagcaggg gaccttcggg
ccttgcgcta tcagatgagc ctaggtcgga ttagctagtt ggtgaggtaa tggctcacca
aggcgacgat ccgtaactgg tctgagagga tgatcagtca cactggaact gagacacggt
ccagactcct acgggaggca gcagtgggga atattggaca atgggcgaaa gcctgatcca
gccatgccgc gtgtgtgaag aaggtcttcg gattgtaaag cactttaagt tgggaggaag
ggcattaacc taatacgtta gtgttttgac gttaccgaca gaataagcac cggctaactc
tgtgccagca gccgcggtaa tacagagggt gcaagcgtta atcggaatta ctgggcgtaa
agcgcgcgta ggtggtttgt taagttggat gtgaaagccc cgggctcaac ctgggaactg
cattcaaaac tgacaagcta gagtatggta gagggtggtg gaatttcctg tgtagcggtg
aaatgcgtag atataggaag gaacaccagt ggcgaaggcg accacctgga ctgatactga
cactgaggtg cgaaagcgtg gggagcaaac aggattagat accctggtag tccacgccgt
aaacgatgtc aactagccgt tggggacctt gagtctttag tggcgcagct aacgcattaa
gttgaccgcc tggggagtac ggccgcaagg ttaaaactca aatgaattga cgggggcccg
cacaagcggt ggagcatgtg gtttaattcg aagcaacgcg aagaacctta ccaggccttg
acatccaatg aactttccag agatggattg gtgccttcgg gaacattgag acaggtgctg
catggctgtc gtcagctcgt gtcgtgagat gttgggttaa gtcccgtaac gagcgcaacc
cttgtcctta gttaccagca cgtaatggtg ggcactctaa ggagactgcc ggtgacaaac
cggaggaagg tggggatgac gtcaagtcat catggccctt acggcctggg ctacacacgt
gctacaatgg tcggtacaga gggttgccaa gccgcgaggt ggagctaatc ccacaaaacc
gatcgtagtc cggatcgcag tctgcaactc gactgcgtga agtcggaatc gctagtaatc
gcgaatcaga atgtcgcggt gaatacgttc ccgggccttg tacacaccgc ccgtcacacc
atgggagtgg gttgcaccag aagtagctag tctaaccttc ggggggac
2.1它属于哪类生物?
答:Pseudomonas fluorescens gene for 16S rRNA, partial sequence, strain: KU-7.(荧光假单胞菌的16SRNA 基因,
2.2它属于哪类基因?
答:属于编码16S rRNA的基因,来源于荧光假单胞菌基因组
2.3它在该基因的什么位置?
答:它在该基因的第1-1428这个位置。
2.4它与你搜索到的序列的同源性(Identities)是多少?
答:同源性100%
四、实验心得:
在输完核苷酸序列之后,一定不能忘记在Database选项中选择Others (nr etc.),不然结果会有很大差异。