高通量测序 名词解释
- 格式:doc
- 大小:39.50 KB
- 文档页数:11

高通量测序原理高通量测序(high-throughput sequencing)是一种快速且高效的基因测序技术,它通过对DNA或RNA样本进行大规模并行测序,能够同时获得大量的基因序列信息。
下面介绍高通量测序的原理。
高通量测序的核心技术之一是DNA片段的扩增。
首先,需要将DNA或RNA样本提取出来,并根据需要进行富集和净化处理。
然后,将样本DNA或RNA分解成较短的片段,通常为几百到几千碱基对。
接下来,为每个片段的两端连接适配体(adapter),适配体中含有特定序列,用于测序和扩增引物的结合。
在测序之前,需要将这些片段通过PCR(聚合酶链反应)进行扩增,形成DNA文库。
文库中的每个片段都带有两端适配体并连接了PCR引物。
最后,将文库进行测序。
高通量测序技术主要有两种方法:SBS(测序by合成)和SMRT(单分子实时测序)。
下面分别介绍它们的原理:1. SBS(Sequencing by Synthesis):这是目前应用最广泛的高通量测序技术。
其原理是通过单个DNA聚合酶复制 DNA的过程,依次加入四种具有不同荧光发射特性的可逆终止核苷酸(dNTPs)。
每次加入一个dNTP后,检测其是否被聚合到待测序片段上,并记录其信号。
然后,将其去除,以便加入下一个dNTP。
重复这个过程,直到测序结束。
通过检测每个位置的荧光信号,就可以获得该位置的碱基信息。
2. SMRT(Single-molecule Real Time sequencing):这种技术利用了DNA聚合酶的优异性质,实现了单分子级别的DNA测序。
SMRT测序使用了一种称为“ZMW”的奇特结构,即零模式波导孔(Zero-mode waveguide)。
在这种结构中,只有非常小的体积(约为20nm)被激光所照亮,并记录荧光信号。
通过DNA聚合酶复制过程,加入了与待测DNA碱基互补的荧光标记的dNTPs,并记录下其荧光信号。
通过不断加入dNTPs,观察荧光信号的变化,就可以获得DNA测序信息。

高通量测序相关名词内部编号:(YUUT-TBBY-MMUT-URRUY-UOOY-DBUYI-0128)高通量测序相关名词高通量测序时,在芯片上的每个反应,会读出一条序列,是比较短的,叫read,它们是原始数据;有很多reads通过片段重叠,能够组装成一个更大的片段,称为contig;多个contigs通过片段重叠,组成一个更长的scaffold;一个contig被组成出来之后,鉴定发现它是编码蛋白质的基因,就叫singleton;多个contigs组装成scaffold之后,鉴定发现它编码蛋白质的基因,叫unigene。
测序深度(Sequencing Depth):测序得到的碱基总量(bp)与基因组大小(Genome)的比值,它是评价测序量的指标之一。
测序深度与基因组覆盖度之间是一个正相关的关系,测序带来的错误率或假阳性结果会随着测序深度的提升而下降。
重测序的个体,如果采用的是双末端或Mate-Pair方案,当测序深度在10~15X以上时,基因组覆盖度和测序错误率控制均得以保证。
什么是高通量测序高通量测序技术(High-throughput sequencing,HTS)是对传统Sanger测序(称为一代测序技术)革命性的改变, 一次对几十万到几百万条核酸分子进行序列测定, 因此在有些文献中称其为下一代测序技术(next generation sequencing,NGS )足见其划时代的改变, 同时高通量测序使得对一个物种的转录组和基因组进行细致全貌的分析成为可能, 所以又被称为深度测序(Deep sequencing)。
什么是Sanger法测序(一代测序)Sanger法测序利用一种DNA聚合酶来延伸结合在待定序列模板上的引物。
直到掺入一种链终止核苷酸为止。
每一次序列测定由一套四个单独的反应构成,每个反应含有所有四种脱氧核苷酸三磷酸(dNTP),并混入限量的一种不同的双脱氧核苷三磷酸(ddNTP)。

生物信息学常用名词解释(一)在生物信息中会出现很多的特殊名词,从这次内容开始,我们将逐渐推送一些生物信息相关的一些名词解释。
生物信息学(bioinformatics):综合计算机科学、信息技术和数学的理论和方法来研究生物信息的交叉学科。
包括生物学数据的研究、存档、显示、处理和模拟,基因遗传和物理图谱的处理,核苷酸和氨基酸序列分析,新基因的发现和蛋白质结构的预测等。
基因组(genome):是指一个物种的单倍体的染色体数目,又称染色体组。
它包含了该物种自身的所有基因。
基因(gene):是遗传信息的物理和功能单位,包含产生一条多肽链或功能RNA所必需的全部核苷酸序列。
基因组学(genomics):是指对所有基因进行基因组作图(包括遗传图谱、物理图谱、转录图谱)、核酸序列测定、基因定位和基因功能分析的科学。
基因组学包括结构基因组学(structural genomics)、功能基因组学(functional genomics)、比较基因组学(Comparative genomics)。
蛋白质组学(proteomics):阐明生物体各种生物基因组在细胞中表达的全部蛋白质的表达模式及功能模式的学科。
包括鉴定蛋白质的表达、存在方式(修饰形式)、结构、功能和相互作用等。
高通量测序:高通量测序技术(High-throughputsequencing,HTS)是对传统Sanger测序(称为一代测序技术)革命性的改变, 一次对几十万到几百万条核酸分子进行序列测定, 因此在有些文献中称其为下一代测序技术(next generation sequencing,NGS )足见其划时代的改变, 同时高通量测序使得对一个物种的转录组和基因组进行细致全貌的分析成为可能, 所以又被称为深度测序(Deep sequencing)。
下一代测序:英文名为Next Generation Sequencing,简称为NGS。
也叫做二代测序或者高通量测序。

高通量测序(NGSHTS)关键名词解释转自:基迪奥生物微信公众号什么是Read?高通量测序平台产生的序列标签就称为reads。
什么是soft-clipped reads?当基因组发生某一段的缺失,或转录组的剪接,在测序过程中,横跨缺失位点及剪接位点的reads回帖到基因组时,一条reads被切成两段,匹配到不同的区域,这样的reads叫做soft-clipped reads,这些reads对于鉴定染色体结构变异及外源序列整合具有重要作用。
什么是Contig?拼接软件基于reads之间的overlap区,拼接获得的序列称为Contig(重叠群)。
什么是Scaffold?基因组de novo测序,通过reads拼接获得Contigs后,往往还需要构建454 Paired-end库或Illumina Mate-pair库,以获得一定大小片段(如3Kb、6Kb、10Kb、20Kb)两端的序列。
基于这些序列,可以确定一些Contig之间的顺序关系,这些先后顺序已知的Contigs 组成Scaffold。
什么是Contig N50?Reads拼接后会获得一些不同长度的Contigs。
将所有的Contig 长度相加,能获得一个Contig总长度。
然后将所有的Contigs按照从长到短进行排序,如获得Contig 1,Contig 2,Contig 3...………Contig 25。
将Contig按照这个顺序依次相加,当相加的长度达到Contig总长度的一半时,最后一个加上的Contig长度即为Contig N50。
举例:Contig 1+Contig 2+ Contig 3+Contig 4=Contig总长度*1/2时,Contig 4的长度即为Contig N50。
Contig N50可以作为基因组拼接的结果好坏的一个判断标准。
什么是Scaffold N50?Scaffold N50与Contig N50的定义类似。

名词解释一、生物学名称解释1. 什么是高通量测序技术?高通量测序技术(High-throughput sequencing,HTS)是对传统Sanger测序(称为一代测序技术)革命性的改变, 一次对几十万到几百万条核酸分子进行序列测定, 因此在有些文献中称其为下一代测序技术(next generation sequencing,NGS )足见其划时代的改变, 同时高通量测序使得对一个物种的转录组和基因组进行细致全貌的分析成为可能, 所以又被称为深度测序(Deep sequencing)。
2. 什么是Sanger法测序(一代测序)?Sanger法测序利用一种DNA聚合酶来延伸结合在待定序列模板上的引物。
直到掺入一种链终止核苷酸为止。
每一次序列测定由一套四个单独的反应构成,每个反应含有所有四种脱氧核苷酸三磷酸(dNTP),并混入限量的一种不同的双脱氧核苷三磷酸(ddNTP)。
由于ddNTP缺乏延伸所需要的3-OH基团,使延长的寡聚核苷酸选择性地在G、A、T或C处终止。
终止点由反应中相应的双脱氧而定。
每一种dNTPs和ddNTPs的相对浓度可以调整,使反应得到一组长几百至几千碱基的链终止产物。
它们具有共同的起始点,但终止在不同的的核苷酸上,可通过高分辨率变性凝胶电泳分离大小不同的片段,凝胶处理后可用X-光胶片放射自显影或非同位素标记进行检测。
3. 什么是SNP、SNV(单核苷酸位点变异)?单核苷酸多态性(single nucleotide polymorphism,SNP)和单核苷酸位点变异(single nucleotide variants, SNV)。
个体间基因组DNA序列同一位置单个核苷酸变异(替代、插入或缺失)所引起的多态性。
不同物种、个体基因组DNA序列同一位置上的单个核苷酸存在差别的现象。
有这种差别的基因座、DNA序列等可作为基因组作图的标志。
人基因组上平均约每1000个核苷酸即可能出现1个单核苷酸多态性的变化,其中有些单核苷酸多态性可能与疾病有关,但可能大多数与疾病无关。

什么是高通量测序?高通量测序技术(High-throughput sequencing,HTS)是对传统Sanger测序(称为一代测序技术)革命性的改变, 一次对几十万到几百万条核酸分子进行序列测定, 因此在有些文献中称其为下一代测序技术(next generation sequencing,NGS )足见其划时代的改变, 同时高通量测序使得对一个物种的转录组和基因组进行细致全貌的分析成为可能, 所以又被称为深度测序(Deep sequencing)。
什么是Sanger法测序(一代测序)Sanger法测序利用一种DNA聚合酶来延伸结合在待定序列模板上的引物。
直到掺入一种链终止核苷酸为止。
每一次序列测定由一套四个单独的反应构成,每个反应含有所有四种脱氧核苷酸三磷酸(dNTP),并混入限量的一种不同的双脱氧核苷三磷酸(ddNTP)。
由于ddNTP缺乏延伸所需要的3-OH基团,使延长的寡聚核苷酸选择性地在G、A、T或C处终止。
终止点由反应中相应的双脱氧而定。
每一种dNTPs和ddNTPs的相对浓度可以调整,使反应得到一组长几百至几千碱基的链终止产物。
它们具有共同的起始点,但终止在不同的的核苷酸上,可通过高分辨率变性凝胶电泳分离大小不同的片段,凝胶处理后可用X-光胶片放射自显影或非同位素标记进行检测。
什么是基因组重测序(Genome Re-sequencing)全基因组重测序是对基因组序列已知的个体进行基因组测序,并在个体或群体水平上进行差异性分析的方法。
随着基因组测序成本的不断降低,人类疾病的致病突变研究由外显子区域扩大到全基因组范围。
通过构建不同长度的插入片段文库和短序列、双末端测序相结合的策略进行高通量测序,实现在全基因组水平上检测疾病关联的常见、低频、甚至是罕见的突变位点,以及结构变异等,具有重大的科研和产业价值。
什么是de novo测序de novo测序也称为从头测序:其不需要任何现有的序列资料就可以对某个物种进行测序,利用生物信息学分析手段对序列进行拼接,组装,从而获得该物种的基因组图谱。
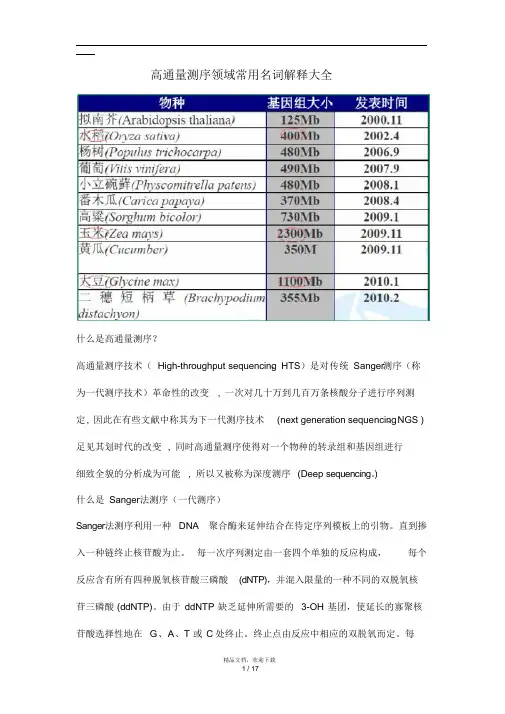
高通量测序领域常用名词解释大全什么是高通量测序?高通量测序技术(High-throughput sequencin,g HTS)是对传统Sanger测序(称为一代测序技术)革命性的改变, 一次对几十万到几百万条核酸分子进行序列测定, 因此在有些文献中称其为下一代测序技术(next generation sequencin,g NGS )足见其划时代的改变, 同时高通量测序使得对一个物种的转录组和基因组进行细致全貌的分析成为可能, 所以又被称为深度测序(Deep sequencing。
)什么是Sanger法测序(一代测序)Sanger法测序利用一种DNA 聚合酶来延伸结合在待定序列模板上的引物。
直到掺入一种链终止核苷酸为止。
每一次序列测定由一套四个单独的反应构成,每个反应含有所有四种脱氧核苷酸三磷酸(dNTP),并混入限量的一种不同的双脱氧核苷三磷酸(ddNTP)。
由于ddNTP 缺乏延伸所需要的3-OH 基团,使延长的寡聚核苷酸选择性地在G、A、T 或C 处终止。
终止点由反应中相应的双脱氧而定。
每一种dNTPs 和ddNTPs 的相对浓度可以调整,使反应得到一组长几百至几千碱基的链终止产物。
它们具有共同的起始点,但终止在不同的的核苷酸上,可通过高分辨率变性凝胶电泳分离大小不同的片段,凝胶处理后可用X-光胶片放射自显影或非同位素标记进行检测。
什么是基因组重测序(Genome Re-sequencin)g全基因组重测序是对基因组序列已知的个体进行基因组测序,并在个体或群体水平上进行差异性分析的方法。
随着基因组测序成本的不断降低,人类疾病的致病突变研究由外显子区域扩大到全基因组范围。
通过构建不同长度的插入片段文库和短序列、双末端测序相结合的策略进行高通量测序,实现在全基因组水平上检测疾病关联的常见、低频、甚至是罕见的突变位点,以及结构变异等,具有重大的科研和产业价值。
什么是de novo 测序de novo 测序也称为从头测序:其不需要任何现有的序列资料就可以对某个物种进行测序,利用生物信息学分析手段对序列进行拼接,组装,从而获得该物种的基因组图谱。

高通量名词解释高通量技术在生物学研究中的应用。
引言。
高通量技术是一种在生物学研究中广泛应用的技术,它可以大大提高实验的效率和数据的产出。
这些技术包括高通量测序、高通量筛选、高通量成像等,可以帮助科学家们更深入地了解生物学过程。
本文将重点介绍高通量技术在生物学研究中的应用,并探讨其在基因组学、蛋白质组学、代谢组学等领域的重要作用。
高通量测序技术。
高通量测序技术是通过对DNA或RNA进行大规模的测序,来获取生物体内基因组、转录组的信息。
这项技术的发展使得科学家们能够更快速、更精确地解读生物体的遗传信息。
通过高通量测序技术,科学家们可以对基因组进行全面的分析,从而揭示基因与疾病之间的关系,研究基因的表达调控机制,甚至进行基因编辑等研究。
此外,高通量测序技术还可以应用于微生物组学研究,帮助科学家们了解微生物在人体内的分布和功能。
高通量筛选技术。
高通量筛选技术是一种用于快速筛选大量样本的技术,可以帮助科学家们在短时间内找到感兴趣的生物分子。
在药物研发领域,高通量筛选技术可以用于快速筛选潜在的药物靶点,加速新药的研发过程。
在生物学研究中,高通量筛选技术可以帮助科学家们发现新的蛋白质相互作用,探索细胞信号通路等。
高通量成像技术。
高通量成像技术是一种用于快速获取大量细胞或组织的图像信息的技术,可以帮助科学家们了解生物体内的结构和功能。
在细胞生物学研究中,高通量成像技术可以用于观察细胞的形态、运动和代谢活动,帮助科学家们研究细胞的生理功能和病理机制。
在神经科学研究中,高通量成像技术可以用于观察大脑的神经元活动,揭示神经网络的组织和功能。
高通量技术在基因组学研究中的应用。
基因组学是研究生物体基因组结构和功能的学科,高通量技术在基因组学研究中发挥着重要作用。
通过高通量测序技术,科学家们可以对不同生物体的基因组进行全面的比较分析,揭示基因组的进化历史和功能差异。
同时,高通量筛选技术可以帮助科学家们快速筛选出与特定生物学过程相关的基因,加速基因功能的研究。

高通量测序常用名词汇总一代测序技术:即传统的 Sanger 测序法, Sanger 法是根据核苷酸在待定序列模板上的引物点开始,随机在某一个特定的碱基处终止,并且在每个碱基后面进行荧光标记,产生以 A、T、 C、 G 结束的四组不同长度的一系列核苷酸,每一次序列测定由一套四个单独的反应构成,每个反应含有所有四种脱氧核苷酸三磷酸(dNTP) ,并混入限量的一种不同的双脱氧核苷三磷酸 (ddNTP) 。
由于 ddNTP 缺乏延伸所需要的 3-OH 基团,使延长的寡聚核苷酸选择性地在 G、A、T 或 C 处终止,使反应得到一组长几百至几千碱基的链终止产物。
它们具有共同的起始点,但终止在不同的的核苷酸上,可通过高分辨率变性凝胶电泳分离大小不同的片段,通过检测得到 DNA 碱基序列。
二代测序技术: next generation sequencing( NGS )又称为高通量测序技术,与传统测序相比,二代测序技术可以一次对几十万到几百万条核酸分子同时进行序列测定,从而使得对一个物种的转录组和基因组进行细致全貌的分析成为可能,所以又被称为深度测序(Deep sequencing )。
NGS 主要的平台有 Roche ( 454 & 454 +), Illumina ( HiSeq 2000/2500、GA IIx 、 MiSeq ), ABI SOLiD 等。
基因:Gene ,是遗传的物质基础,是DNA或RNA分子上具有遗传信息的特定核苷酸序列。
基因通过复制把遗传信息传递给下一代,使后代出现与亲代相似的性状。
DNA :Deoxyribonucleic acid,脱氧核糖核酸,一个脱氧核苷酸分子由三部分组成:含氮碱基、脱氧核糖、磷酸。
脱氧核糖核酸通过3',5'- 磷酸二酯键按一定的顺序彼此相连构成长链,即 DNA 链,DNA 链上特定的核苷酸序列包含有生物的遗传信息,是绝大部分生物遗传信息的载体。

—1—高通量测序技术分类及简介什么是高通量测序?高通量测序(high-throughput sequencing)并不是指一般意义上通量高的测序,而是特指二代测序(next generation sequencing ,NGS)。
NGS 也翻译成下一代测序、新一代测序、平行测序。
NGS 一次反应能同时对数百亿个核酸分子进行测序(cluster 密度高达数M/mm2),虽然测序长度(读长)比一代测序短,但是模板分子数(=平行进行的测序反应数)的增加幅度惊人,所以测序通量比一代测序提高了数千万倍,测序成本的降低速度超越摩尔定律。
由于数据量大规模提高,NGS 使得对一个物种进行基因组分析和转录组分析成为现实;由于成本大规模降低,NGS 使得临床和消费者基因检测应用变成了现实。
一代测序与二代测序的要点比较如下:什么是denovo 测序?denovo测序也叫从头测序,指一个物种第一次开展全基因组测序,其NGS数据的生物信息学数据分析由于没有现成的基因组参考序列(reference sequence)可用,算法比较特殊,难度也比较大。
通常会组合运用多种测序方式,比如NGS,转录组测序(提供RNA 剪接与可变转录本等信息),三代测序(长读长)等技术,数据相互参照,以取得高质量的组装图,因此成本也比较高。
denovo测序的化学反应与标准NGS测序一样;但是其生物信息学数据分析算法不同,全基因组序列组装过程中不使用基因组参考序列,运算耗时较长。
什么是重测序(re-sequencing)?随着NGS技术的发展,基因组测序所需成本和时间较传统技术大幅降低,越来越多的物种获得了全基因组序列。
有了基因组参考序列后,对于同一物种其他个体的测序,其生物信息学数据分析就变得相对简单了,此种测序称为重测序。
这种测序的化学反应部分与标准的NGS一样,但是生物信息学数据分析比denovo测序简单,依赖reference sequence进行全基因组组装,运算简单,速度快。

高通量测序基础知识汇总一代测序技术:即传统的Sanger测序法,Sanger法是根据核苷酸在待定序列模板上的引物点开始,随机在某一个特定的碱基处终止,并且在每个碱基后面进行荧光标记,产生以A、T、C、G结束的四组不同长度的一系列核苷酸,每一次序列测定由一套四个单独的反应构成,每个反应含有所有四种脱氧核苷酸三磷酸(dNTP),并混入限量的一种不同的双脱氧核苷三磷酸(ddNTP)。
由于ddNTP缺乏延伸所需要的3-OH基团,使延长的寡聚核苷酸选择性地在G、A、T或C处终止,使反应得到一组长几百至几千碱基的链终止产物。
它们具有共同的起始点,但终止在不同的的核苷酸上,可通过高分辨率变性凝胶电泳分离大小不同的片段,通过检测得到DNA碱基序列。
二代测序技术:next generation sequencing(NGS)又称为高通量测序技术,与传统测序相比,二代测序技术可以一次对几十万到几百万条核酸分子同时进行序列测定,从而使得对一个物种的转录组和基因组进行细致全貌的分析成为可能,所以又被称为深度测序(Deep sequencing)。
NGS主要的平台有Roche(454 & 454+),Illumina(HiSeq 2000/2500、GA IIx、MiSeq),ABI SOLiD等。
基因:Gene,是遗传的物质基础,是DNA或RNA分子上具有遗传信息的特定核苷酸序列。
基因通过复制把遗传信息传递给下一代,使后代出现与亲代相似的性状。
DNA:Deoxyribonucleic acid,脱氧核糖核酸,一个脱氧核苷酸分子由三部分组成:含氮碱基、脱氧核糖、磷酸。
脱氧核糖核酸通过3',5'-磷酸二酯键按一定的顺序彼此相连构成长链,即DNA链,DNA链上特定的核苷酸序列包含有生物的遗传信息,是绝大部分生物遗传信息的载体。
RNA:Ribonucleic Acid,,核糖核酸,一个核糖核苷酸分子由碱基,核糖和磷酸构成。
高通量测序流程和原理
高通量测序(High-throughput sequencing)是一种快速、高效的DNA测序技术,也被称为第二代测序技术。
它的出现极大地推动了基因组学和生物信息学的发展,为基因组变异、表达调控、蛋白质组学等研究领域提供了强大的支持。
高通量测序的流程可以简单概括为DNA提取、文库构建、测序仪测序和数据分析四个步骤。
首先是DNA提取,从样本中提取出所需的DNA,可以是基因组DNA、表达物的cDNA等。
接下来是文库构建,将提取的DNA片段连接到测序引物上,形成文库。
然后是测序仪测序,将文库中的DNA片段进行高通量测序,得到大量的原始测序数据。
最后是数据分析,对原始数据进行质控、比对、组装和功能注释等一系列分析,最终得到所需的生物信息学结果。
高通量测序的原理主要基于测序引物的引导下,通过不断地合成和检测新的核苷酸碱基,从而逐渐构建起整个DNA片段的序列。
常见的高通量测序技术包括Illumina测序、Ion Torrent测序、PacBio测序等,它们各自采用不同的原理和方法,但都能实现高通量的DNA测序。
在实际应用中,高通量测序技术被广泛应用于基因组测序、转录组测序、表观基因组测序等领域。
它不仅在科学研究中发挥着重要作用,还在临床诊断、生物工程、农业育种等领域有着广阔的应用前景。
总之,高通量测序技术以其快速、高效、准确的特点,成为现代生物学研究中不可或缺的重要工具,为我们深入了解生命的奥秘提供了有力支持。
随着技术的不断进步和应用的不断拓展,相信高通量测序技术将为生命科学领域带来更多的惊喜和突破。
生物信息学中的高通量测序技术随着科技的迅速发展,生物学领域越来越重视高通量测序技术的应用。
高通量测序技术是一种快速测序大量生物分子的方法,尤其是DNA和RNA分子。
这项技术的应用范围非常广泛,包括了基因组学、转录组学、蛋白质组学等领域。
今天,我们将会深入探讨生物信息学中高通量测序技术的应用和发展。
什么是高通量测序技术?高通量测序技术是一种快速并且自动的测序方法,可以用于同时测序多个DNA或RNA分子。
这是一项革命性的技术,而在实际应用中,高通量测序技术可以大幅度降低测序成本,提高测序速度,并且提高数据准确度。
迄今为止,高通量测序技术已经成为生物学领域研究重要的工具。
高通量测序技术的种类1. Sanger测序Sanger测序是传统的测序方法,也被称作链终止法测序。
这种方法利用DNA聚合酶能在特定的条件下,将有标签的哺乳动物链结束核苷酸(ddNTPs)加入到DNA单链中,从而得到不同长度的DNA片段。
然后,通过分离特定长度的DNA片段并进行酶水解,就可以得到原始的DNA序列。
Sanger测序的优点是准确性高,适用于较短的DNA片段测序。
但是这种方法非常费时、费钱,并且不能进行大规模的DNA测序。
2. PyrosequencingPyrosequencing方法是一种基于酵素活性的RNA扩增技术。
首先,将DNA片段与引物和未标记的核苷酸混合,然后在特定条件下引发DNA聚合。
接下来,将酶质子释放到反应中,进一步触发酶反应,从而释放出类似于火花的光。
通过检测这些光的表现形式,便可以得到DNA序列。
Pyrosequencing方法具有较高的分辨率和专业的测序精度,并且可以进行高速并行测序,可以快速获得大量的核苷酸序列。
3. Illumina测序Illumina测序是一种高效的测序方法,可以同时测序上至数百万个不同的片段。
该方法被广泛应用于测序人类基因组、病原体和肿瘤学等方面。
Illumina测序优点是样品处理时间极短,不需要大量的火花合成,测序成本非常低,通常可以得到准确的测序结果。
高通量测序常见名词解释什么是高通量测序?高通量测序技术(High-throughput sequencing,HTS)是对传统Sanger测序(称为一代测序技术)革命性的改变,一次对几十万到几百万条核酸分子进行序列测定, 因此在有些文献中称其为下一代测序技术(next generation sequencing,NGS )足见其划时代的改变, 同时高通量测序使得对一个物种的转录组和基因组进行细致全貌的分析成为可能, 所以又被称为深度测序(Deep sequencing)。
什么是Sanger法测序(一代测序)Sanger法测序利用一种DNA聚合酶来延伸结合在待定序列模板上的引物。
直到掺入一种链终止核苷酸为止。
每一次序列测定由一套四个单独的反应构成,每个反应含有所有四种脱氧核苷酸三磷酸(dNTP),并混入限量的一种不同的双脱氧核苷三磷酸(ddNTP)。
由于ddNTP缺乏延伸所需要的3-OH基团,使延长的寡聚核苷酸选择性地在G、A、T或C处终止。
终止点由反应中相应的双脱氧而定。
每一种dNTPs和ddNTPs的相对浓度可以调整,使反应得到一组长几百至几千碱基的链终止产物。
它们具有共同的起始点,但终止在不同的的核苷酸上,可通过高分辨率变性凝胶电泳分离大小不同的片段,凝胶处理后可用X-光胶片放射自显影或非同位素标记进行检测。
什么是基因组重测序(Genome Re-sequencing)全基因组重测序是对基因组序列已知的个体进行基因组测序,并在个体或群体水平上进行差异性分析的方法。
随着基因组测序成本的不断降低,人类疾病的致病突变研究由外显子区域扩大到全基因组范围。
通过构建不同长度的插入片段文库和短序列、双末端测序相结合的策略进行高通量测序,实现在全基因组水平上检测疾病关联的常见、低频、甚至是罕见的突变位点,以及结构变异等,具有重大的科研和产业价值。
什么是de novo测序de novo测序也称为从头测序:其不需要任何现有的序列资料就可以对某个物种进行测序,利用生物信息学分析手段对序列进行拼接,组装,从而获得该物种的基因组图谱。
高通量测序基础知识汇总一代测序技术:即传统的Sanger测序法,Sanger法是根据核苷酸在待定序列模板上的引物点开始,随机在某一个特定的碱基处终止,并且在每个碱基后面进行荧光标记,产生以A、T、C、G结束的四组不同长度的一系列核苷酸,每一次序列测定由一套四个单独的反应构成,每个反应含有所有四种脱氧核苷酸三磷酸(dNTP),并混入限量的一种不同的双脱氧核苷三磷酸(ddNTP)。
由于ddNTP缺乏延伸所需要的3-OH 基团,使延长的寡聚核苷酸选择性地在G、A、T或C处终止,使反应得到一组长几百至几千碱基的链终止产物。
它们具有共同的起始点,但终止在不同的的核苷酸上,可通过高分辨率变性凝胶电泳分离大小不同的片段,通过检测得到DNA碱基序列。
二代测序技术:next generation sequencing(NGS)又称为高通量测序技术,与传统测序相比,二代测序技术可以一次对几十万到几百万条核酸分子同时进行序列测定,从而使得对一个物种的转录组和基因组进行细致全貌的分析成为可能,所以又被称为深度测序(Deep sequencing)。
NGS主要的平台有Roche(454 & 454+),Illumina(HiSeq 2000/2500、GA IIx、MiSeq),ABI SOLiD等。
基因:Gene,是遗传的物质基础,是DNA或RNA分子上具有遗传信息的特定核苷酸序列。
基因通过复制把遗传信息传递给下一代,使后代出现与亲代相似的性状。
DNA:Deoxyribonucleic acid,脱氧核糖核酸,一个脱氧核苷酸分子由三部分组成:含氮碱基、脱氧核糖、磷酸。
脱氧核糖核酸通过3',5'-磷酸二酯键按一定的顺序彼此相连构成长链,即DNA链,DNA链上特定的核苷酸序列包含有生物的遗传信息,是绝大部分生物遗传信息的载体。
RNA:Ribonucleic Acid,,核糖核酸,一个核糖核苷酸分子由碱基,核糖和磷酸构成。
核糖核苷酸经磷酯键缩合而成长链状分子称之为RNA链。
RNA是存在于生物细胞以及部分病毒、类病毒中的遗传信息载体。
不同种类的RNA链长不同,行使各式各样的生物功能,如参与蛋白质生物合成的RNA有信使RNA、转移RNA和核糖体RNA等。
16S rDNA:"S"是沉降系数,是反映生物大分子在离心场中向下沉降速度的一个指标,值越高,说明分子越大。
rDNA(ribosome DNA)指的是原核生物基因组中编码核糖体RNA (rRNA)分子对应的DNA序列,16S rDNA 是原核生物编码核糖体小亚基16S rRNA的基因。
细菌rRNA(核糖体RNA)按沉降系数分为3种,分别为5S、16S和23S rRNA。
16S rDNA是细菌染色体上编码16S rRNA相对应的DNA序列,存在于所有细菌染色体基因中。
16S rRNA 普遍存在于原核生物中。
16S rRNA 分子,其大小约1540bp,既含有高度保守的序列区域,又有中度保守和高度变化的序列区域,其可变区序列因细菌不同而异,恒定区序列基本保守,所以可利用恒定区序列设计引物,将16S rDNA片段扩增出来,通过高通量测序利用可变区序列的差异来对不同菌属、菌种的细菌进行分类鉴定。
cDNA:complementary DNA,互补脱氧核糖核酸,与RNA链互补的单链DNA,以RNA 为模板,在反转录酶的作用下所合成的DNA。
Small RNA:生物体内一类高度保守的重要的功能分子,其大小在18-30nt,包括microRNA、siRNA、snRNA、snoRNA和piRNA(piwi-interacting RNA)等,它的主要功能是诱导基因沉默,调控细胞生长、发育、基因转录和翻译等生物学过程。
以miRNA 为例介绍它们的功能:miRNA与RNA诱导沉默复合体(RNA induced silencing complex,RISC)结合,并将此复合体与其互补的mRNA序列结合,根据靶序列与miRNA的互补程度,从而导致靶序列降解或干扰靶序列蛋白质的翻译过程。
SD区域:Segment duplication,串联重复是由序列相近的一些 DNA 片段串联组成。
串联重复在人类基因多样性的灵长类基因中发挥重要作用。
Genotype and phenotype:基因型与表型,基因型是指某一生物个体全部基因组合的总称;表型,又称性状,是基因型和环境共同作用的结果。
基因组:Genome,单倍体细胞核、细胞器(线粒体、叶绿体)或病毒粒子所含的全部DNA 分子或RNA分子。
全基因组de novo测序:又称从头测序,它不依赖于任何现有的序列资料,而直接对某个物种的基因组进行测序,然后利用生物信息学分析手段对序列进行拼接、组装,从而获得该物种的基因组序列图谱。
全基因组重测序:对已有参考序列(Reference Sequence)物种的不同个体进行基因组测序,并以此为基础进行个体或群体水平的遗传差异性分析。
全基因组重测序能够发现大量的单核苷酸多态性位点(SNP)、拷贝数变异(Copy Number Variation,CNV)、插入缺失(InDel,Insertion/Deletion)、结构变异(Structure Variation,SV)等变异类型,以准确快速的方法将单个参考基因组信息上升为群体遗传特征。
转录组:Transcriptome,是指特定生长阶段某组织或细胞内所有转录产物的集合;狭义上指所有mRNA的集合。
转录组测序:对某组织在某一功能状态下所能转录出来的所有RNA进行测序,获得特定状态下的该物种的几乎所有转录本序列信息。
通常转录组测序是指对mRNA进行测序获得相关序列的过程。
其根据所研究物种是否有参考基因组序列分为转录组de novo测序(无参考基因组序列)和转录组重测序(有参考基因组序列)。
外显子组:Exome,人类基因组全部外显子区域的集合称为外显子组,是基因中重要的编码蛋白的部分,并涵盖了与个体表型相关的大部分的功能性变异。
外显子组测序:是指利用序列捕获技术将全基因组外显子区域DNA捕捉并富集后进行高通量测序的基因组分析方法。
外显子测序相对于基因组重测序成本较低,对研究已知基因的SNP、InDel 等具有较大的优势。
目标区域测序:应用相关试剂盒对基因组上感兴趣的目标区域进行捕获富集后进行大规模测序,一般需要根据目标区域专门定制捕获芯片。
宏基因组:Metagenome,指特定生活环境中全部微小生物遗传物质的总和。
它包含了可培养的和未可培养的微生物的基因。
目前主要指环境样品中的细菌和真菌的基因组总和。
宏基因组16S rRNA测序:可以对特定环境下的细菌和古细菌群体的微生物种类和风度进行有效的鉴定。
对不同地点、不同条件下的多个样本16S rRNA的PCR产物平行测序,可以比较不同样本间的微生物组成及成分差异,进而阐明物种丰度、种群结果等生态学信息。
表观遗传学:Epigenetics,是指在基因组DNA序列没有改变的情况下,基因的表达调控和性状发生了可遗传的变化。
表观遗传的现象很多,已知的有DNA甲基化(DNA methylation),基因组印记(genomic impriting),母体效应(maternal effects),基因沉默(gene silencing),核仁显性,休眠转座子激活和RNA编辑(RNA editing)等。
全基因组甲基化测序:DNA 甲基化是指在 DNA 甲基化转移酶的作用下,在基因组 CpG 二核苷酸的胞嘧啶5'碳位共价键结合一个甲基基团。
DNA 甲基化已经成为表观遗传学和表观基因组学的重要研究内容。
甲基化是基因表达的主要调控方式之一,研究染色体DNA甲基化情况是了解基因调控的重要手段。
对已经有参考基因组的物种的基因组DNA用标准亚硫酸氢盐(Bisulfite)处理后,未甲基化的胞嘧啶C会脱氨基形成尿嘧啶U,经PCR扩增,U替换为胸腺嘧啶T,而发生甲基化的胞嘧啶C保持不变。
将处理组与参考基因组序列进行比对,可发现甲基化位点并对甲基化情况进行定量分析的方法叫做全基因组甲基化测序。
ChIp-Seq:Chromatin Immunoprecipitation sequencing,即染色质免疫共沉淀-测序技术,即通过染色质免疫共沉淀技术特异性地富集目的蛋白结合的DNA片段。
对富集得到的DNA片段进行纯化与文库构建,然后进行高通量测序,从而得到全基因组范围内可以与目的蛋白相互作用的DNA片段的方法叫做ChIP-Seq。
数字表达谱:Digital Gene Expression Profile,利用新一代高通量测序技术和高性能计算分析技术,能够全面、经济、快速地检测某一物种特定组织在特定状态下的基因表达情况,即运用特定的酶对mRNA距polyA tail 21-25nt的位置进行酶切,所获得的带polyA尾的序列(Tag)通过高通量测序,该tag被测得的次数即是对应基因的表达值。
数字基因表达谱已被广泛应用于基础科学研究、医学研究和药物研发等领域。
特点是经济,但获得的数据量有限。
若想获得转录本的更多信息的话,一般都采用转录组测序的方法来测序。
SBS:sequencing by synthesis,边合成边测序反应,是指在DNA聚合酶的作用下延伸碱基所进行的测序。
Run:指高通量测序平台单次上机测序反应。
Lane:也叫channel,单泳道,每条泳道包含2列(column),每列分布有多个小区(tile),如图1。
不同的测序平台Flow Cell中所含的Lane不一样,如HiSeq 2000是2个flow cell,每个flow cell中含有8个lane;HiSeq 2500是包含2个mini flow cell(快速运行模式)和2个high output flow cell,两个模式不能同时运行,其中每个mini flow cell包含2个lane,每个high output flow cell中包含8个lane;Miseq系统的flow cell仅含有1个lane。
Tile:小区,每条Lane中有2列tile,合计120个小区。
每个小区上分布数目繁多的簇结合位点,如图1。
Cluster:簇,在Illumina测序平台中会采用桥式PCR方式生产DNA簇,每个DNA簇才能产生亮度达到CCD可以分辨的荧光点。
Index:标签,在Illumina平台的多重测序(Multiplexed Sequencing)过程中会使用Index 来区分样品,并在常规测序完成后,针对Index部分额外进行7个循环的测序,通过Index 的识别,可以在1条Lane中区分12种不同的样品。