荧光原位杂交检测的原理
- 格式:pdf
- 大小:9.15 MB
- 文档页数:40

荧光原位杂交(Fluorescence In Situ Hybridization, 简称FISH)由于其直观, 快速, 敏感性高和方便灵活越来越得到广泛应用, 尤其是在血液学领域中. 因为白血病标本比较容易取得和制备, 不同类型的白血病又往往有其特异的染色体异常, FISH在白血病诊断, 治疗监测, 预后估计和微小残留病检测等诸方面都正成为不可缺少的重要手段.FISH的基本原理很简单, 就是标记了荧光的单链DNA(探针)和与其互补的DNA(玻片上的标本)退火杂交, 通过观察荧光信号在染色体上的位置来反映相应基因的情况.FISH探针按标记方法可分为直接标记和间接标记: 用生物素(biotin)或地高辛(digoxingenin)标记称为间接标记, 杂交后需要通过免疫荧光抗体检测方能看到荧光信号, 因而步骤较多, 操作麻烦, 其优点是在信号较弱或较小时可经抗原抗体反应扩大; 直接用荧光素标记DNA的方法称为直接标记. 由于直接标记的探针杂交后可马上观察到荧光信号, 省去了烦琐的免疫荧光反应, 不再需要购买荧光抗体, 也由于近年来荧光互的亮度和抗淬灭性的不断改进和提高, 直接标记的荧光探针越来越成为首选, 采用多种不同颜色的荧光, 方便在同一标本上同时检测多钟异常. 其荧光强度和信号大小都易于在普通荧光显微镜下观察, 操作过程中也不需要严格避光, 使FISH过程变得简便而易于操作. FISH并不能取代传统的白血病MCI诊断, 但它却能使MIC分型更为准确和深入. , MIC即细胞形态学(M), 免疫学和细胞遗传学(C), 三者结合对白血病进行分型诊断, 对不同类型的白血病采用不同治疗方案手段. 随着人们对白血病的不断认识, 仅进行MIC分型不够全面, 还要加上对白血病的分子(M)诊断, 成为MICM分型. FISH就是连接细胞遗传学和分子生物学的桥梁.FISH流程仪器设备1、医用微波炉;2、水浴锅;3、OLYMPUS BX51荧光显微镜;4、OLYMPUS DP11数字显微照相机。
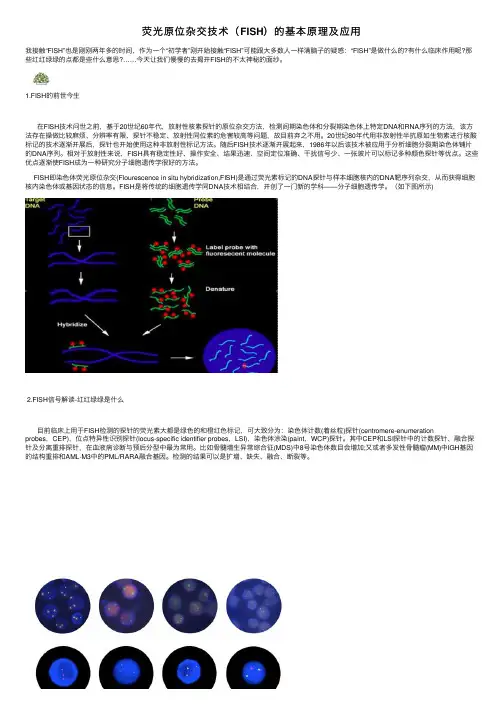
荧光原位杂交技术(FISH)的基本原理及应⽤我接触“FISH”也是刚刚两年多的时间,作为⼀个“初学者”刚开始接触“FISH”可能跟⼤多数⼈⼀样满脑⼦的疑惑:“FISH”是做什么的?有什么临床作⽤呢?那些红红绿绿的点都是些什么意思?……今天让我们慢慢的去揭开FISH的不太神秘的⾯纱。
1.FISH的前世今⽣在FISH技术问世之前,基于20世纪60年代,放射性核素探针的原位杂交⽅法,检测间期染⾊体和分裂期染⾊体上特定DNA和RNA序列的⽅法,该⽅法存在操做⽐较⿇烦、分辨率有限、探针不稳定、放射性同位素的危害较⾼等问题,故⽬前弃之不⽤。
20世纪80年代⽤⾮放射性半抗原如⽣物素进⾏核酸标记的技术逐渐开展后,探针也开始使⽤这种⾮放射性标记⽅法。
随后FISH技术逐渐开展起来,1986年以后该技术被应⽤于分析细胞分裂期染⾊体铺⽚的DNA序列。
相对于放射性来说,FISH具有稳定性好、操作安全、结果迅速、空间定位准确、⼲扰信号少、⼀张玻⽚可以标记多种颜⾊探针等优点。
这些优点逐渐使FISH成为⼀种研究分⼦细胞遗传学很好的⽅法。
FISH即染⾊体荧光原位杂交(Flourescence in situ hybridization,FISH)是通过荧光素标记的DNA探针与样本细胞核内的DNA靶序列杂交,从⽽获得细胞核内染⾊体或基因状态的信息。
FISH是将传统的细胞遗传学同DNA技术相结合,开创了⼀门新的学科——分⼦细胞遗传学。
(如下图所⽰)2.FISH信号解读-红红绿绿是什么⽬前临床上⽤于FISH检测的探针的荧光素⼤都是绿⾊的和橙红⾊标记,可⼤致分为:染⾊体计数(着丝粒)探针(centromere-enumerationprobes,CEP),位点特异性识别探针(locus-specific identifier probes,LSI),染⾊体涂染(paint,WCP)探针。
其中CEP和LSI探针中的计数探针、融合探针及分离重排探针,在⾎液病诊断与预后分型中最为常⽤。

荧光原位杂交技术原理及操作步骤1974年Evans首次将染色体显带技术和染色体原位杂交联合应用,提高了定位的准确性。
20世纪70年代后期人们开始探讨荧光标记的原位杂交,即FISH技术。
1981年Harper成功地将单拷贝的DNA序列定位到G显带标本上,标志着染色体定位技术取得了重要进展。
20世纪90年代,随着人类基因组计划的进行,由于绘制高分辨人类基因组图谱的需要,FISH技术得到了迅速的发展和广泛应用。
1.原理 FISH(fluorescence in situ hybridization)技术是一种重要的非放射性原位杂交技术。
它的基本原理是:如果被检测的染色体或DNA纤维切片上的靶DNA与所用的核酸探针是同源互补的,二者经变性-退火-复性,即可形成靶DNA与核酸探针的杂交体。
将核酸探针的某一种核苷酸标记上报告分子如生物素.地高辛,可利用该报告分子与荧光素标记的特异亲和素之间的免疫化学反应,经荧光检测体系在镜下对待测DNA进行定性.定量或相对定位分析。
2.实验流程 FISH样本的制备→探针的制备→探针标记→杂交→染色体显带→荧光显微镜检测→结果分析。
3.特点原位杂交的探针按标记分子类型分为放射性标记和非放射性标记。
用同位素标记的放射性探针优势在于对制备样品的要求不高,可以通过延长曝光时间加强信号强度,故较灵敏。
缺点是探针不稳定.自显影时间长.放射线的散射使得空间分辨率不高.及同位素操作较繁琐等。
采用荧光标记系统则可克服这些不足,这就是FISH技术。
FISH技术作为非放射性检测体系,具有以下优点:1.荧光试剂和探针经济.安全;2.探针稳定,一次标记后可在两年内使用;3.实验周期短.能迅速得到结果.特异性好.定位准确;4.FISH可定位长度在1kb的DNA序列,其灵敏度与放射性探针相当;5.多色FISH通过在同一个核中显示不同的颜色可同时检测多种序列;6.既可以在玻片上显示中期染色体数量或结构的变化,也可以在悬液中显示间期染色体DNA的结构。

微生物荧光原位杂交实验技术背景微生物荧光原位杂交实验技术是在原位杂交技术的基础上发展而来的。
原位杂交技术最早应用于染色体分析,后来逐渐应用于微生物检测领域。
随着荧光标记技术的不断发展,人们开始利用荧光标记探针进行原位杂交,从而提高了检测的灵敏度和特异性。
原理微生物荧光原位杂交实验技术的原理是利用特定的荧光标记探针与细胞中的微生物进行杂交,从而将微生物定性和定量地检测出来。
该技术的基本原理是碱基互补配对原则,即探针的序列与待测微生物的序列互补,从而形成稳定的杂交双链。
利用荧光检测仪器检测荧光信号,从而实现对微生物的定量和定位分析。
实验方法样品的制备:将待测样品进行处理,使微生物细胞分离并保持活性。
探针的制备:将特定的DNA或RNA片段进行标记,形成荧光探针。
杂交反应:将样品和探针在一定条件下进行杂交反应,形成杂交双链。
洗涤和干燥:去除未结合的探针和杂质,保持杂交信号的特异性。
荧光检测:利用荧光检测仪器检测样品的荧光信号,并对数据进行处理和分析。
实验结果通过微生物荧光原位杂交实验技术,我们可以得到样品的定性和定量数据。
实验的成功率较高,特异性较强,能够清晰地检测出目标微生物的存在和数量。
该技术的灵敏度较高,可以检测出低拷贝数的微生物基因,为研究提供了有力的工具。
实验讨论微生物荧光原位杂交实验技术具有许多优势,如高特异性、高灵敏度和能够保持细胞结构的完整性等。
然而,该技术也存在一些不足之处,如探针制备过程较为繁琐、杂交反应条件要求较高以及荧光检测仪器价格昂贵等。
荧光探针的稳定性也可能影响实验结果的可靠性。
因此,在应用该技术时需要注意这些因素,并选择合适的探针和实验条件,以保证实验结果的准确性和可靠性。
结论微生物荧光原位杂交实验技术在研究领域具有广泛的应用前景。
除了在微生物检测方面的应用,该技术还可以应用于其他领域,如基因表达分析、细胞凋亡研究等。
虽然该技术存在一些不足之处,但随着技术的不断发展和优化,相信未来会有更多的应用前景等待着我们去探索和发现。


荧光原位杂交技术及其应用作者:钱文丹陈波利来源:《乡村科技》 2018年第25期1 荧光原位杂交技术原理荧光原位杂交技术(Fluorescence in situ hybridization,FISH)是一种重要的非放射性原位杂交技术。
其基本原理是如果被检测的染色体或DNA纤维切片上的靶DNA与所用的核酸探针是同源互补的,二者经变性—退火—复性,即可形成靶DNA与核酸探针的杂交体。
将核酸探针的某一种核苷酸标记上报告分子如生物素、地高辛,可利用该报告分子与荧光素标记的特异亲和素之间的免疫化学反应,经荧光检测体系在镜下对待测DNA进行定性、定量或相对定位分析[1]。
与其他杂交技术进行综合比较发现,荧光原位杂交技术(FISH)具有一些优势:循环周期短,稳定性高,非常安全;分辨率高,为3~20 Mb;探针能较长时间保存;多色标记,简单直观;在荧光显微镜下在同一切片上同时观察几种DNA探针的定位,直接得到其相对序列和位置,从而大大加速生物基因组和功能基因组定位的研究。
2 荧光原位杂交技术要点FISH 选用的标本可以是分裂期细胞染色体,也可以是间期细胞。
生物素、地高辛、Dinitrophenyl(DNP)、AminoacetylFluorine(AAF)等均可用于探针标记。
近年来,大片段的DNA探针(100~400 kb)已被研制出来,由于控针较长,故可将荧光物质直接标记在核苷酸上,使杂交过程进一步简化,而且杂交信号更强。
荧光原位杂交可以通过CCD(电荷耦合器件)相机系统或激光共聚焦扫描成像系统将摄取的信号存储在计算机中,经过软件特殊处理后显示在屏幕上。
使用数码成像相机系统或CCD相机系统,灰度图像被拍摄几次并存储在计算机中,接下来通过一些人造色,软件系统接收获得的示例图像,经过软件的综合处理,最后以多色图像显示出来。
此外,在G-带状染色体被75 酒精或甲醇褪色后,FISH可以更清楚地识别易位。
FISH 技术和RFLP(Restrict Fragment Lenth Polymorphysim)结合,可以更准确地描述染色体长短臂的结构变化以及染色体梳子或复制品的性质。

荧光原位杂交技术原理及操作步骤1974年Evans首次将染色体显带技术和染色体原位杂交联合应用,提高了定位的准确性。
20世纪70年代后期人们开始探讨荧光标记的原位杂交,即FISH技术。
1981年Harper成功地将单拷贝的DNA序列定位到G显带标本上,标志着染色体定位技术取得了重要进展。
20世纪90年代,随着人类基因组计划的进行,由于绘制高分辨人类基因组图谱的需要,FISH技术得到了迅速的发展和广泛应用。
1.原理 FISH(fluorescence in situ hybridization)技术是一种重要的非放射性原位杂交技术。
它的基本原理是:如果被检测的染色体或DNA纤维切片上的靶DNA与所用的核酸探针是同源互补的,二者经变性-退火-复性,即可形成靶DNA与核酸探针的杂交体。
将核酸探针的某一种核苷酸标记上报告分子如生物素.地高辛,可利用该报告分子与荧光素标记的特异亲和素之间的免疫化学反应,经荧光检测体系在镜下对待测DNA进行定性.定量或相对定位分析。
2.实验流程 FISH样本的制备→探针的制备→探针标记→杂交→染色体显带→荧光显微镜检测→结果分析。
3.特点原位杂交的探针按标记分子类型分为放射性标记和非放射性标记。
用同位素标记的放射性探针优势在于对制备样品的要求不高,可以通过延长曝光时间加强信号强度,故较灵敏。
缺点是探针不稳定.自显影时间长.放射线的散射使得空间分辨率不高.及同位素操作较繁琐等。
采用荧光标记系统则可克服这些不足,这就是FISH技术。
FISH技术作为非放射性检测体系,具有以下优点:1.荧光试剂和探针经济.安全;2.探针稳定,一次标记后可在两年内使用;3.实验周期短.能迅速得到结果.特异性好.定位准确;4.FISH可定位长度在1kb的DNA序列,其灵敏度与放射性探针相当;5.多色FISH通过在同一个核中显示不同的颜色可同时检测多种序列;6.既可以在玻片上显示中期染色体数量或结构的变化,也可以在悬液中显示间期染色体DNA的结构。

荧光原位杂交技术(fish)的基本原理和应用理论说明1. 引言1.1 概述荧光原位杂交技术(Fluorescence in situ Hybridization,简称FISH)是一种广泛应用于生物学研究的重要技术。
它通过在细胞或组织水平上定位和检测特定DNA或RNA序列的分布情况,可以提供关于基因组结构、功能和表达的有价值信息。
该技术最早于20世纪80年代被开发出来,并且经过不断改进与扩展,如今已成为分子生物学研究中不可或缺的工具之一。
1.2 文章结构本文将首先介绍荧光原位杂交技术的基本原理,包括DNA探针的选择与设计、杂交反应条件的优化以及检测与可视化方法。
然后,我们将深入探讨荧光原位杂交技术在生物医学研究领域、植物遗传研究领域和动物进化研究领域的应用实例。
接下来,我们将评述荧光原位杂交技术的优势与局限性,包括其高灵敏度、高分辨率等优势以及对样本处理要求高、无法确定基因功能等局限性。
最后,我们将给出结论并展望荧光原位杂交技术的未来发展方向。
1.3 目的本文的目的是系统地介绍荧光原位杂交技术的基本原理和应用领域,以帮助读者深入了解这一重要技术。
通过阅读本文,读者将能够全面了解荧光原位杂交技术在生物学研究中的作用和意义,并对该技术的优势与局限性有所了解。
此外,本文也将探讨该技术未来可能的发展方向,为读者提供展望与思考。
2. 荧光原位杂交技术基本原理:2.1 DNA探针的选择与设计:荧光原位杂交技术(FISH)是一种利用DNA或RNA分子作为探针,通过特异性互补配对识别和定位目标序列的方法。
在进行FISH实验时,首先需要选择合适的DNA探针。
DNA探针通常由由人工合成的寡聚核苷酸(oligonucleotide)或从天然来源提取得到的全长DNA片段构建而成。
选择DNA探针时,需要考虑以下因素:首先是目标序列的特异性,即该序列在待检测样品中是否具有较高的丰度,并且只存在于感兴趣的目标区域中。
其次是探针长度和两个主要互补区域之间核苷酸序列的碱基组成比例。

细胞荧光原位杂交检查
细胞荧光原位杂交(Fluorescence In Situ Hybridization, FISH)是一种用于检测细胞内基因组序列的分子生物学技术。
它通过将特定的DNA或RNA探针与目标序列杂交,并用荧光素标记探针,然后在荧光显微镜下观察杂交信号,从而确定目标序列的位置和数量。
FISH检查可用于研究细胞中基因组序列的改变,如染色体异常、基因扩增、基因缺失等。
它也可用于检测某些基因在特定组织或细胞类型中的表达情况。
下面是FISH检查的一般步骤:
1. 准备探针:根据需要,设计并制备特异性探针,通常为DNA或RNA探针。
2. 制备细胞样品:从组织或细胞培养物中制备样品,一般需要用胰蛋白酶消化、固定等步骤处理细胞。
3. 杂交:将探针与样品中的目标序列进行杂交,一般需要在一定温度和离子强度下进行。
4. 洗涤:去除未结合的探针,减少背景干扰。
5. 荧光素标记:用特定的荧光素标记探针,以便在荧光显微镜下观察。
6. 观察:用荧光显微镜观察杂交信号,并记录目标序列的数量和位置。
FISH检查的优点包括高特异性、高灵敏度、能够准确定位目标序列的位置。
然而,它也存在一些局限性,如需要昂贵的设备和试剂、需要训练有素的技术人员等。

荧光原位杂交实验(FISH)荧光原位杂交(Fluorescence in situ hybridization FISH)是一门新兴的分子细胞遗传学技术,是20世纪80年代末期在原有的放射性原位杂交技术的基础上发展起来的一种非放射性原位杂交技术。
目前这项技术已经广泛应用于动植物基因组结构研究、染色体精细结构变异分析、病毒感染分析、人类产前诊断、肿瘤遗传学和基因组进化研究待许多领域。
1实验方法原理:荧光原位杂交(Fluorescence in situ hybridization FISH)是一门新兴的分子细胞遗传学技术,是20世纪80年代末期在原有的放射性原位杂交技术的基础上发展起来的一种非放射性原位杂交技术。
目前这项技术已经广泛应用于动植物基因组结构研究、染色体精细结构变异分析、病毒感染分析、人类产前诊断、肿瘤遗传学和基因组进化研究待许多领域。
FISH 的基本原理是用已知的标记单链核酸为探针,按照碱基互补的原则,与待检材料中未知的单链核酸进行异性结合,形成可被检测的杂交双链核酸。
由于DNA分子在染色体上是沿着染色体纵轴呈线性排列,因而可以探针直接与染色体进行杂交从而将特定的基因在染色体上定位。
与传统的放射性标记原位杂交相比,荧光原位杂交具有快速、检测信号强、杂交特异性高和可以多重染色等特点,因此在分子细胞遗传学领域受到普遍关注。
杂交所用的探针大致可以分类三类:1)染色体特异重复序列探针,例如α卫星、卫星III类的探针,其杂交靶位常大于1Mb,不含散在重复序列,与靶位结合紧密,杂交信号强,易于检测;2)全染色体或染色体区域特异性探针,其由一条染色体或染色体上某一区段上极端不同的核苷酸片段所组成,可由克隆到噬菌体和质粒中的染色体特异大片段获得;3)特异性位置探针,由一个或几个克隆序列组成。
探针的荧光素标记可以采用直接和间接标记的方法。
间接标记是采用生物素标记DNA探针,杂交之后用藕联有荧光素亲和素或者链霉亲和素进行检测,同时还可以利用亲和素-生物素-荧光素复合物,将荧光信号进行放大,从而可以检测500bp的片段。

DNA荧光原位杂交(FISH)简要综述DNA荧光原位杂交(FISH)简要综述DNA荧光原位杂交(FISH)技术是70年代末80年代初开始发展起来的一种重要的非放射性原位杂交技术,它的基本原理是将DNA探针用特殊修饰的核苷酸分子标记(如biotin-dUTP或digoxigenin-dUTP),然后将标记的探针直接原位杂交到染色体或DNA纤维切片上,再用与荧光素分子偶联的单克隆抗体与探针分子特性结合来检测DNA序列在染色体或DNA纤维上的定位。
利用FISH进行DNA序列的定位具有实验周期短、灵敏度高、分辨率高、直观可见等优点。
总的说来,FISH技术的发展沿着两条路线前进:一方面是采用不同的探针,从而衍生出许多FISH新技术,如Muticolor-FISH、CGH、GISH、CISS、BAC-FISH、Chromosome Painting、Rreverse Chromosome Painting等等;另一方面则是努力提高FISH技术的分辨率,将靶目标从中期染色体发展到DNA纤维,使其分辨率由1Mb 发展到1kb,这更进一步拓展了FISH技术的应用领域,成为分子细胞遗传学的一项代表技术。
A.不同探针的应用:1.以基因组为探针的GISH技术可以定位外源DNA片段在染色体上的位置、大小、插入点等。
Durnam(1985)首先将GISH应用于体细胞杂种的异源染色体检测。
本实验室采用GISH方法在小麦中成功定位了许多外源染色体、染色体片段以及染色体结构变异。
2.以不同的荧光素标记探针的Muticolor-FISH,可以同时定位不同探针序列的分布。
1990年Nederlof等创建了多色荧光原位杂交技术。
他们用生物素、AAF(氨基乙酰荧光素)和CP三种半抗原对不同探针作单、双、三标记,再用这三种半抗原相应的分别标记了异硫氰酸荧光素(FITC,绿色)、氨甲香豆素乙酸(AMCA,蓝色)和碱性蕊香红(TRITC,红色)的抗体来检测荧光,该技术最多可同时观察7个靶染色体。
组织荧光原位杂交
实验
组织荧光原位杂交实验是一种检测特定基因在细胞内表达情况的技术,可以在不破坏细胞形态的情况下进行。
它是利用DNA核酸合成技术,将一个DNA序列克隆到一个载体中,然后将该载体转染到细胞中,使之与目的基因杂交,并通过荧光技术检测杂交体的存在及其强度,从而间接检测目的基因的表达情况。
实验步骤如下:
1.准备探针:准备荧光标记的探针,一般采用双螺旋DNA或cDNA作为探针,将它们与特定荧光染料混合,得到荧光探针。
2.准备细胞:采用细胞培养和支持剂,将细胞培养到最佳密度,以便可以在实验中检测到荧光信号。
3.细胞转染:将探针转染到细胞中,以进行原位杂交实验。
4.荧光检测:采用荧光显微镜技术,检测转染后细胞的荧光信号强度,从而检测出目的基因的表达情况。