分析realtimePCR数据
- 格式:doc
- 大小:406.00 KB
- 文档页数:10


荧光定量pcr步骤荧光定量PCR(real-timePCR)一种高通量的核酸定量分析技术,用于检测和定量检测基因表达以及实验条件下的细菌基因或病毒基因含量。
荧光定量PCR是基于反转录-聚合酶链反应(RT-PCR)和实时PCR技术,结合这两种技术,可以非常快速地检测和定量基因表达。
本文将介绍荧光定量PCR的步骤。
第一步:样品的准备与检测1.1品的准备:首先,细菌或病毒样品根据实验要求进行灭菌或病毒灭活。
1.2测:根据需要,采用适当的抗体检测样品中是否有病毒和细菌,将病毒和细菌样品中的RNA或DNA分离出来,将分离出来的核酸用于下一步检测。
第二步:荧光定量PCR反应2.1品添加:将分离出来的核酸和所需的实验试剂(如反转录酶、DNA聚合酶、定量PCR探针、模板DNA,以及相关配套试剂)混合,反应体系得到。
2.2动PCR反应:将反应体系定温热处理,使反转录酶向模板DNA 中的特定序列引物亲和,以实现反转录。
2.3入PCR探针:将定量PCR探针加入反应液中,以实现基因表达荧光定量PCR。
2.4复PCR循环:每次循环引入一定量的反应物,以实现基因表达荧光定量PCR,并在每次循环时观察荧光信号,从而实现基因表达定量。
第三步:数据分析3.1据分析:对荧光信号数据进行定量分析,实现基因表达定量,并将结果画在实验曲线上,以观察基因表达的变化情况。
3.2验结果:在实验曲线上,横坐标为PCR循环次数,纵坐标为基因表达量,可以观察实验结果,以确定基因表达量的情况。
荧光定量PCR步骤是用于检测和定量检测基因表达以及实验条件下的细菌基因或病毒基因含量的有效技术,它包括样品的准备和检测、荧光定量PCR反应、数据分析三个步骤,可以快速准确地定量检测基因表达情况,为实验中的细菌和病毒基因分析领域提供有效的参考依据。


实时荧光定量PCR的数据分析方法
作者:易健明, 屈武斌, 张成岗, YI Jian-Ming, QU Wu-Bin, ZHANG Cheng-Gang
作者单位:易健明,张成岗,YI Jian-Ming,ZHANG Cheng-Gang(军事医学科学院放射与辐射医学研究所,蛋白质组学国家重点实验室,全军军事认知与心理卫生研究中心,北京100850;安徽医科大学研究生院,安徽合肥230032)
, 屈武斌,QU Wu-Bin(军事医学科学院放射与辐射医学研究所,蛋白质组学国家重点实验室,全军军事认知
与心理卫生研究中心,北京100850)
刊名:
生物技术通讯
英文刊名:Letters in Biotechnology
年,卷(期):2015,26(1)
引用本文格式:易健明.屈武斌.张成岗.YI Jian-Ming.QU Wu-Bin.ZHANG Cheng-Gang实时荧光定量PCR的数据分析方法[期刊论文]-生物技术通讯 2015(1)。




一般来讲,进行real-time qPCR MasterMix都是2×的浓缩液,只需要加入模板和引物就可以。
由于real-time qPCR灵敏度高,所以每个样品至少要做3个平行孔,以防在后面的数据分析中,由于Ct相差较多或者SD太大,无法进行统计分析。
通常来讲,反应体系的引物终浓度为100-400mM;模板如果是总RNA一般是10ng-500,如果cDNA,通常情况下是1ul或者1ul的10倍稀释液,要根据目的基因的表达丰度进行调整。
当然这些都是经验值,在操作过程中,还需要根据所用MasterMix,模板和引物的不同进行优化,达到一个最佳反应体系。
在反应体系配置过程中,有下面几点需要注意:1. MasterMix不要反复冻融,如果经常使用,最好溶解后放在4度。
2. 更多的配制Mix进行,减少加样误差。
最好能在冰上操作。
一般来讲,进行real-time qPCR MasterMix都是2×的浓缩液,只需要加入模板和引物就可以。
由于real-time qPCR灵敏度高,所以每个样品至少要做3个平行孔,以防在后面的数据分析中,由于Ct相差较多或者SD太大,无法进行统计分析。
通常来讲,反应体系的引物终浓度为100-400mM;模板如果是总RNA一般是10ng-500,如果cDNA,通常情况下是1ul或者1ul的10倍稀释液,要根据目的基因的表达丰度进行调整。
当然这些都是经验值,在操作过程中,还需要根据所用MasterMix,模板和引物的不同进行优化,达到一个最佳反应体系。
在反应体系配置过程中,有下面几点需要注意:1. MasterMix不要反复冻融,如果经常使用,最好溶解后放在4度。
3. 每管或每孔都要换新枪头!不要连续用同一个枪头加样!4. 所有成分加完后,离心去除气泡。
5. 每个样品至少3个平行孔。
参比或者校正染料(reference dye,passive dye)常用的是ROXTM(现在已经是ABI的注册商标了!)或者其他染料,只要不影响检测PCR产物的荧光值就可以。

real time PCR 数据分析real-timepcr数据分析无论使用哪种类型的实时PCR,正确的数据分析对于获得有效的实验结果都非常重要。
这里我们介绍实时PCR数据分析的知识。
在讨论基本分析过程之前,先介绍如何设计一个好的实验。
如果你是自己设计的引物和探针,那有助于下一步的工作。
但是在有些情况下,人们使用出版文献上的序列会更方便。
记住,即便是出版物提供的序列也不能保证会得到优化的实验结果。
而且排版错误的可能性也需要考虑在内。
所以进入实验室之前使用blast对全部序列进行核实确保他们是正确的。
下订单前先检察引物和探针的序列和tm值是实验设计的基本要求。
标准曲线是判断实验质量的重要手段。
以已知模板、PCR产物、合成寡核苷酸或转录RNA为标准曲线,可以确定PCR的效率、灵敏度、动态范围等参数。
建立标准曲线时使用od260的模板样本。
模板的总量由DNA分子的数量来描述。
将质量转化为DNA含量的公式如下:(质量(克)*阿伏伽德罗常数)每个碱基的平均质量*模板的长度。
例如,合成70-mer单链DNA时,样品质量为0.8*10-11gm。
代入公式:(0.8*10?-11*6.023*10?23molecules/mole)330gm/mole/base*70base。
如果使用双链模板,基底的平均质量为660克/摩尔/基底。
标准曲线使用的模板含量从1*10?7开始连续稀释7次每次稀释10倍,最终得到10个模板拷贝。
这样的浓度有助于得到最高的δrn和最低的ct。
用excel画曲线时以模板数量的对数值为x,ct(cyclethreshold)值为y轴。
标准曲线的计算公式如下:y=mx+b。
y是CT,M是斜率,x=log10templateamount,b=y-截距。
用斜率计算出实验效率efficiency【10?(-1/斜率)】-1。
实验效率告诉我们pcr反应的执行情况。
鉴定系数r?2是实际结果和理论值相符程度,表示稀释和移液的准确性。

实时荧光定量PCR仪用于流感病毒检测的效果分析
实时荧光定量PCR(Real-time quantitative PCR, qPCR)技术是一种高灵敏度和高特异性的检测技术,在流感病毒的检测中得到了广泛的应用。
本文将对实时荧光定量PCR仪在流感病毒检测中的效果进行分析。
实时荧光定量PCR仪能够实现对流感病毒的准确定量。
PCR反应中的引物和探针是专门设计用于选择性地扩增和检测流感病毒的基因序列,通过荧光信号的变化可以实时监测PCR扩增的过程。
而且,实时荧光定量PCR仪具有高灵敏度,可以检测到非常低浓度的病毒核酸,对样本中的流感病毒进行精确测量。
实时荧光定量PCR仪在流感病毒的快速检测和定量方面具有优势。
实时荧光定量PCR仪的数据分析和结果判读也相对简便。
实时荧光定量PCR仪可以实时监测PCR反应的荧光强度,并将反应强度转化为相对或绝对的目标基因拷贝数,通过阈值周期数(Ct value)作为结果判读的依据。
阈值周期数的大小与样本中目标基因的存在量成反比,因此可以通过比较不同样本中的Ct值来判断流感病毒的感染程度和扩增量。
这种结果的呈现和数据分析相对简单明了,适用于实验人员的操作和结果解读。

Real-time PCR 原理介绍实时荧光定量PCR技术于1996年由美国Applied Biosystems公司推出,由于该技术不仅实现了PCR从定性到定量的飞跃,而且与常规PCR相比,它具有特异性更强、有效解决PCR污染问题、自动化程度高等特点,目前已得到广泛应用。
本文试就其定量原理、方法及参照问题作一介绍。
一.实时荧光定量PCR原理所谓实时荧光定量PCR技术,是指在PCR反应体系中加入荧光基团,利用荧光信号积累实时监测整个PCR进程,最后通过标准曲线对未知模板进行定量分析的方法。
1.Ct 值的定义在荧光定量PCR技术中,有一个很重要的概念 —— Ct值。
C代表Cycle,t代表threshold,Ct值的含义是:每个反应管内的荧光信号到达设定的域值时所经历的循环数(如图1所示)。
图1. Ct值的确定2.荧光域值(threshold)的设定PCR反应的前15个循环的荧光信号作为荧光本底信号,荧光域值的缺省设置是3-15个循环的荧光信号的标准偏差的10倍,即:threshold = 10 ´ SDcycle 6-153.Ct值与起始模板的关系研究表明,每个模板的Ct值与该模板的起始拷贝数的对数存在线性关系〔1〕,起始拷贝数越多,Ct值越小。
利用已知起始拷贝数的标准品可作出标准曲线,其中横坐标代表起始拷贝数的对数,纵坐标代Ct值(如图2所示)。
因此,只要获得未知样品的Ct值,即可从标准曲线上计算出该样品的起始拷贝数。
图2. 荧光定量标准曲线4.荧光化学荧光定量PCR所使用的荧光化学可分为两种:荧光探针和荧光染料〔2〕。
现将其原理简述如下:1)TaqMan荧光探针:PCR扩增时在加入一对引物的同时加入一个特异性的荧光探针,该探针为一寡核苷酸,两端分别标记一个报告荧光基团和一个淬灭荧光基团。
探针完整时,报告基团发射的荧光信号被淬灭基团吸收;PCR扩增时,Taq酶的5’-3’外切酶活性将探针酶切降解,使报告荧光基团和淬灭荧光基团分离,从而荧光监测系统可接收到荧光信号,即每扩增一条DNA链,就有一个荧光分子形成,实现了荧光信号的累积与PCR产物形成完全同步。

荧光定量PCR实验及数据分析一、概述荧光定量PCR(Quantitative Realtime PCR,简称qPCR)是一种结合了PCR技术的高灵敏度和荧光探针技术的实时定量特性的分子生物学分析方法。
该方法通过实时监测PCR反应过程中荧光信号的变化,对模板DNA或RNA的初始浓度进行定量分析。
荧光定量PCR技术在基因表达研究、病原体检测、基因突变分析以及药物疗效评估等领域具有广泛的应用价值。
在荧光定量PCR实验中,通常使用特异性引物和荧光探针来识别并扩增目标序列。
荧光探针的设计是关键步骤之一,它必须能够与目标序列特异性结合并在PCR过程中产生可检测的荧光信号。
实验过程中还需严格控制反应条件,包括温度、时间、引物和探针的浓度等,以确保实验的准确性和可重复性。
数据分析是荧光定量PCR实验不可或缺的一部分。
通过对实验数据的收集、整理和分析,可以获取目标序列的初始浓度信息,进而对实验结果进行解读和评估。
数据分析方法包括相对定量和绝对定量两种,前者通过比较不同样本间目标序列的相对表达量来评估差异,后者则通过标准曲线法或质粒拷贝数法等方法来确定目标序列的绝对浓度。
荧光定量PCR技术是一种高效、灵敏且特异的分子生物学分析方法,对于研究基因表达、病原体检测等领域具有重要意义。
通过不断优化实验操作和数据分析方法,可以进一步提高荧光定量PCR实验的准确性和可靠性,为科学研究和临床实践提供有力支持。
1. 荧光定量PCR技术的概述荧光定量PCR技术,是一种基于DNA聚合酶链式反应的分子生物学技术,它通过引入荧光标记,实时监测PCR过程中目标DNA片段的扩增情况,从而实现对特定基因拷贝数的精确量化。
该技术结合了PCR的高效扩增能力与荧光信号的灵敏检测,使得微量DNA分子的检测成为可能,并在遗传学、分子生物学、医学诊断等领域中发挥着重要作用。
荧光定量PCR技术主要依赖于特异性引物和探针的设计,使得PCR扩增过程具有高度的特异性。

由于Real-time qPCR 的众多优点,现在已是生命科学领域的一项常规技术。
越来越多的研究文章中涉及RT-PCR 的实验,也基本上被real-time qPCR 所代替。
由于real-time aPCR 输出的数据不同于常规的PCR 电泳检测,不少没有做过real-time qPCR 的研究者往往感到高深莫测,不知从何入手;甚至一些做过次实验的研究者也会对数据处理分析感到迷惑,不知所措。
本文就从real-time qPCR 的发展史说起,包括real-time qPCR 的原理,实验设计,实际操作,数据分析,常见问题解答五个方面,手把手教你从各个方面了解real-time qPCR,彻底的从菜鸟到高手!一、Real-time qPCR 发展史Real-time qPCR 就是在PCR 扩增过程中,通过荧光信号,对PCR 进程进行实时检测。
由于在PCR 扩增的指数时期,模板的Ct 值和该模板的起始拷贝数存在线性关系,所以成为定量的依据。
由于常规的PCR 的缺点,real-time qPCR 由于其操作简便,灵敏度高,重复性好等优点发展非常迅速。
现在已经涉及到生命科学研究的各个领域,比如基因的差异表达分析,SNP 检测,等位基因的检测,药物开辟,临床诊断,转基因研究等。
在Real-time qPCR 技术的发展过程中,定量PCR 仪的发展起了至关重要的作用。
1995 年,美国PE 公司(已经并入Invitrogen 公司)成功研制了Taqman 技术,1996 年推出了首台荧光定量PCR 检测系统,通过检测每一个循环的荧光强度,通过Ct 值进行数据分析。
从而荧光定量PCR 获得广泛应用。
现在的定量PCR 仪有ABI7000、7300、7500,7700、7900HT、StepOnePlusTM、StepOneTM、PRISM@StepOneTM 系列;BIO-RAD 的CFX96、iCycler iQ5@、MyiQ@、MJ Research Chromo4TM Opticon 系列;Stratagene MxTM 系列;Roche LightCycler@ 系列;Eppendorf Masercycler@;Corbett Rotor-GeneTM;Cepheid SmartCycler@和BIOER 的LineGene 系列。

首先我们得来说说染料法SYBR Green的原理,SYBR Green能够结合在双链DNA上面的荧光染料,只有结合了双链DNA,才会发荧光,而游离的不会发光。
图1 SYBR Green原理
但是,染料没有选择特异性,就是只要有双链DNA,就会染上,并发荧光。
如果使用的引物产生了非特异性扩增,那么荧光强度就不是目的条带真实的强度,因此会产生严重偏差。
融解曲线我们如何判断本次qPCR扩增的都是目的片段
呢?这就需要用到融解曲线。
在qPCR循环反应结束之后,系统会进行测定融解曲线,通常的方式就是从70度加热到90度,然后每隔一定时间(1s或者少于1s)测定系统荧光强度,随着温度的升高,dsDNA都解开双链,SYBR Green 都游离之后不发荧光,荧光逐渐下降。
那么画出来一个个荧光强度和温度的曲线:
图2 融解曲线
然后我们运用医学中学到的高等数学C的求导,把荧光强度对温度求导,得出下图
图3 融解曲线的求导
为什么中间的峰那么高?dR/dT 越大,表明荧光值变化最快。
随着温度上升,达到图中Tm点时,大部分扩增出来的双链DNA解开,荧光值下降非常快。
如果qPCR产物非常特异那么,融解曲线在80-90之间会形成一个单峰(温度和qPCR产物长度以及GC含量相关)。
但是除了单峰还会出现什么样的情况呢?请看下面两个示意图
图4 前置杂峰
图5 后置杂峰。

荧光定量PCR(Quantitative Real-Time PCR,简称qPCR)是一种分子生物学技术,用于精确测定样本中特定核酸序列的数量。
其基本原理基于PCR(聚合酶链式反应)技术和实时荧光检测,能够在PCR扩增过程中连续监测荧光信号的变化,从而实现对起始模板量的定量分析。
荧光定量PCR原理简述:1.PCR扩增:qPCR采用传统的PCR方法,包括变性(DNA双链解开成单链)、退火(引物与靶序列配对)和延伸(DNA聚合酶合成新链)这三个基本步骤,反复进行使得目标序列指数级扩增。
2.荧光标记与检测:SYBR Green法:SYBR Green是一种非特异性的双链DNA结合染料,在游离状态下几乎不发出荧光,但一旦与双链DNA结合后,荧光强度显著增强。
因此,随着PCR过程中的产物增加,荧光信号也相应增加,荧光强度与PCR产物的数量成正比。
TaqMan探针法:此方法更为特异,使用一种特殊的寡核苷酸探针,其两端分别标记了荧光报告基团和淬灭基团。
在PCR反应中,当探针与靶序列配对时,位于中间的探针被Taq 酶水解,导致荧光报告基团与淬灭基团分离,从而产生荧光信号。
只有当特定的扩增产物生成时才会释放荧光。
荧光定量PCR实验步骤概览:1.样品制备:RNA提取:从组织、细胞或其他生物样本中提取总RNA,常用TRIZOL或类似试剂进行裂解、离心分相和乙醇沉淀来纯化RNA。
cDNA合成:对于mRNA的定量,需要先将RNA逆转录为cDNA。
2.设计与合成引物:针对目标基因设计一对特异性的PCR引物,用于扩增目的片段。
3.PCR反应体系构建:将纯化的cDNA或DNA模板、特异性引物、Taq聚合酶、缓冲液、dNTPs和其他必要成分如SYBR Green染料或TaqMan探针等加入至PCR管中,配置成最终的PCR反应体系。
4.实时荧光PCR扩增与检测:在荧光定量PCR仪上进行PCR反应,仪器在每次循环的适当阶段收集荧光信号,并记录下来。

用2-△△Ct法分析real-time PCR数据-----联合应用LightCycler Data Analysis软件和MS ExcelBy netmee,netmee163.引用请注明作者。
1、打开存取的数据文件,点击2、依次点击,,这是适合SYBR green为染料的选项。
点击step1:Baseline下的“change graph settings”小图标。
取消弹出的Customize Graph选项卡中的Logarithmis选项,点击“OK”按钮,退出选项卡。
3、点击下的“change graph settings”小图标,取消弹出的Customize Graph选项卡中的Logarithmis选项,点击“OK”按钮,退出选项卡。
4、在选项卡中拖动红色标记线,选取个条曲线都为直线上升部位。
5、可以在选项卡中观察是否需选取的是曲线直线上升部分。
观察绿线部分与S型曲线交叉的部分是否为直线。
6、如果确为直线,则左侧的“Crossing Point”值为所需要的Ct值。
7、依次选取下图菜单:将数据导出为文本文件。
8、打开所保存的文本文件,如图选取9、将数据粘贴入新建的excel文件,“Crossing Point”列即是Ct值,删除“Standard”和“Calculated Concentration”列。
10、将目的基因,本例中为“iNOS”的Ct值按标本对应剪切入beta-actin值右侧一列。
分别标记两列数据为“beta-actin”和“iNOS”。
11、设置所有Ct值的单元格格式12、数字选项卡,分类选择为数值,点击确定。
13、将E列(目的基因Ct值右侧一列)输入公式“=D4-C4”,求出目的基因与同管beta-actin Ct值之差,即△Ct。
14、向下拉复制公式,将△Ct列数值计算出。
15、在△Ct 列右侧一列插入公式“=POWER(2,(0-E4))”,此即目的基因相对beta-actin的相对表达量,即2-△Ct(2的-△Ct次方)。
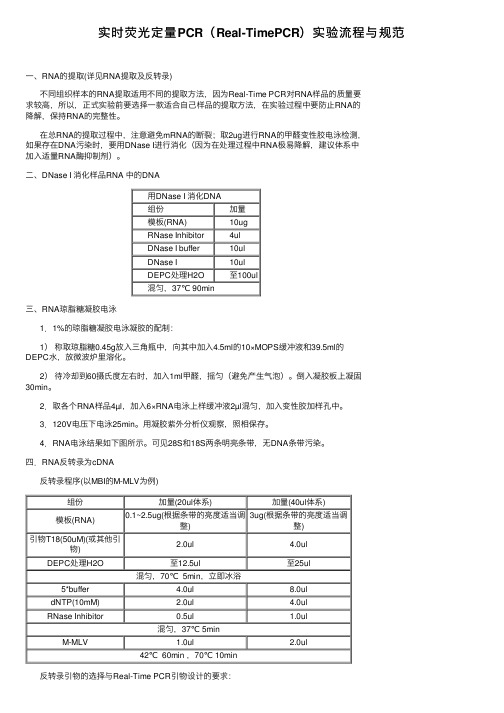
实时荧光定量PCR(Real-TimePCR)实验流程与规范⼀、RNA的提取(详见RNA提取及反转录)不同组织样本的RNA提取适⽤不同的提取⽅法,因为Real-Time PCR对RNA样品的质量要求较⾼,所以,正式实验前要选择⼀款适合⾃⼰样品的提取⽅法,在实验过程中要防⽌RNA的降解,保持RNA的完整性。
在总RNA的提取过程中,注意避免mRNA的断裂;取2ug进⾏RNA的甲醛变性胶电泳检测,如果存在DNA污染时,要⽤DNase I进⾏消化(因为在处理过程中RNA极易降解,建议体系中加⼊适量RNA酶抑制剂)。
⼆、DNase I 消化样品RNA 中的DNA⽤DNase I 消化DNA组份加量模板(RNA)10ugRNase Inhibitor 4ulDNase I buffer10ulDNase I 10ulDEPC处理H2O ⾄100ul混匀,37℃ 90min三、RNA琼脂糖凝胶电泳1.1%的琼脂糖凝胶电泳凝胶的配制:1)称取琼脂糖0.45g放⼊三⾓瓶中,向其中加⼊4.5ml的10×MOPS缓冲液和39.5ml的DEPC⽔,放微波炉⾥溶化。
2)待冷却到60摄⽒度左右时,加⼊1ml甲醛,摇匀(避免产⽣⽓泡)。
倒⼊凝胶板上凝固30min。
2.取各个RNA样品4µl,加⼊6×RNA电泳上样缓冲液2µl混匀,加⼊变性胶加样孔中。
3.120V电压下电泳25min。
⽤凝胶紫外分析仪观察,照相保存。
4.RNA电泳结果如下图所⽰。
可见28S和18S两条明亮条带,⽆DNA条带污染。
四.RNA反转录为cDNA反转录程序(以MBI的M-MLV为例)组份加量(20ul体系)加量(40ul体系)模板(RNA)0.1~2.5ug(根据条带的亮度适当调整)3ug(根据条带的亮度适当调整)引物T18(50uM)(或其他引物)2.0ul 4.0ulDEPC处理H2O⾄12.5ul⾄25ul混匀,70℃ 5min,⽴即冰浴5*buffer 4.0ul8.0ul dNTP(10mM) 2.0ul 4.0ul RNase Inhibitor0.5ul 1.0ul混匀,37℃ 5minM-MLV 1.0ul 2.0ul42℃ 60min ,70℃ 10min反转录引物的选择与Real-Time PCR引物设计的要求:1)随机六聚体引物:当特定mRNA由于含有使反转录酶终⽌的序列⽽难以拷贝其全长序列时,可采⽤随机六聚体引物这⼀不特异的引物来拷贝全长mRNA。

分析real-time-PCR数据
————————————————————————————————作者:————————————————————————————————日期:
ﻩ
用2-△△Ct法分析real-time PCR数据-----联合应用LightCycler Data Analysis软件和MS Excel
By netmee,
引用请注明作者。
1、打开存取的数据文件,点击
2、依次点击,,这是适合SYBRgreen为染料的选项。
点击step1:Baseline下的“change graph settings”小图标。
取消弹出的Customize
Graph选项卡中的Logarithmis选项,点击“OK”按钮,退出选项卡。
3、点击下的“change graph settings”小图标
,取消弹出的CustomizeGraph选项卡中的Logarithm
is选项,点击“OK”按钮,退出选项卡。
4、在选项卡中拖动红色标记线,选取个条曲线都为直线上升部位。
5、可以在选项卡中观察是否需选取的是曲线直线上升部分。
观察绿线部分与S型曲线交叉的部分是否为直线。
6、如果确为直线,则左侧的“CrossingPoint”值为所需要的Ct值。
7、依次选取下图菜单:将数据导出为文本文件。
8、打开所保存的文本文件,如图选取
9、将数据粘贴入新建的excel文件,“CrossingPoint”列即是Ct值,删除“Standa rd”和“Calculated Concentration”列。
10、将目的基因,本例中为“iNOS”的Ct值按标本对应剪切入beta-actin值右侧一列。
分别标记两列数据为“beta-actin”和“iNOS”。
11、设置所有Ct值的单元格格式
12、数字选项卡,分类选择为数值,点击确定。
13、将E列(目的基因Ct值右侧一列)输入公式“=D4-C4”,求出目的基因与同管beta-actin Ct值之差,即△Ct。
14、向下拉复制公式,将△Ct列数值计算出。
15、在△Ct列右侧一列插入公式“=POWER(2,(0-E4))”,此即目的基因相对beta-actin 的相对表达量,即2-△Ct(2的-△Ct次方)。
16、在2-△Ct列右侧插入公式“=F4/$F$4”,此列即“2-△△Ct”列,复制公式填满所有标本对应的2-△△Ct空格。
17、至此,2-△△Ct法计算的各标本目的基因的相对表达量完成,2-△△Ct列的数据可以用统计软件进行分析。