
汉恒生物基于Gibson Assembly的无缝克隆快速设计教程
软件:Genome Compiler
设计举例:把LacZ克隆到pEGFP-N3中,得pLacZ-EGFP
难点:LacZ需要和EGFP融合表达,所以克隆位点的选择尤其关键!
注:Snapgene也可以设计,容后开篇介绍。
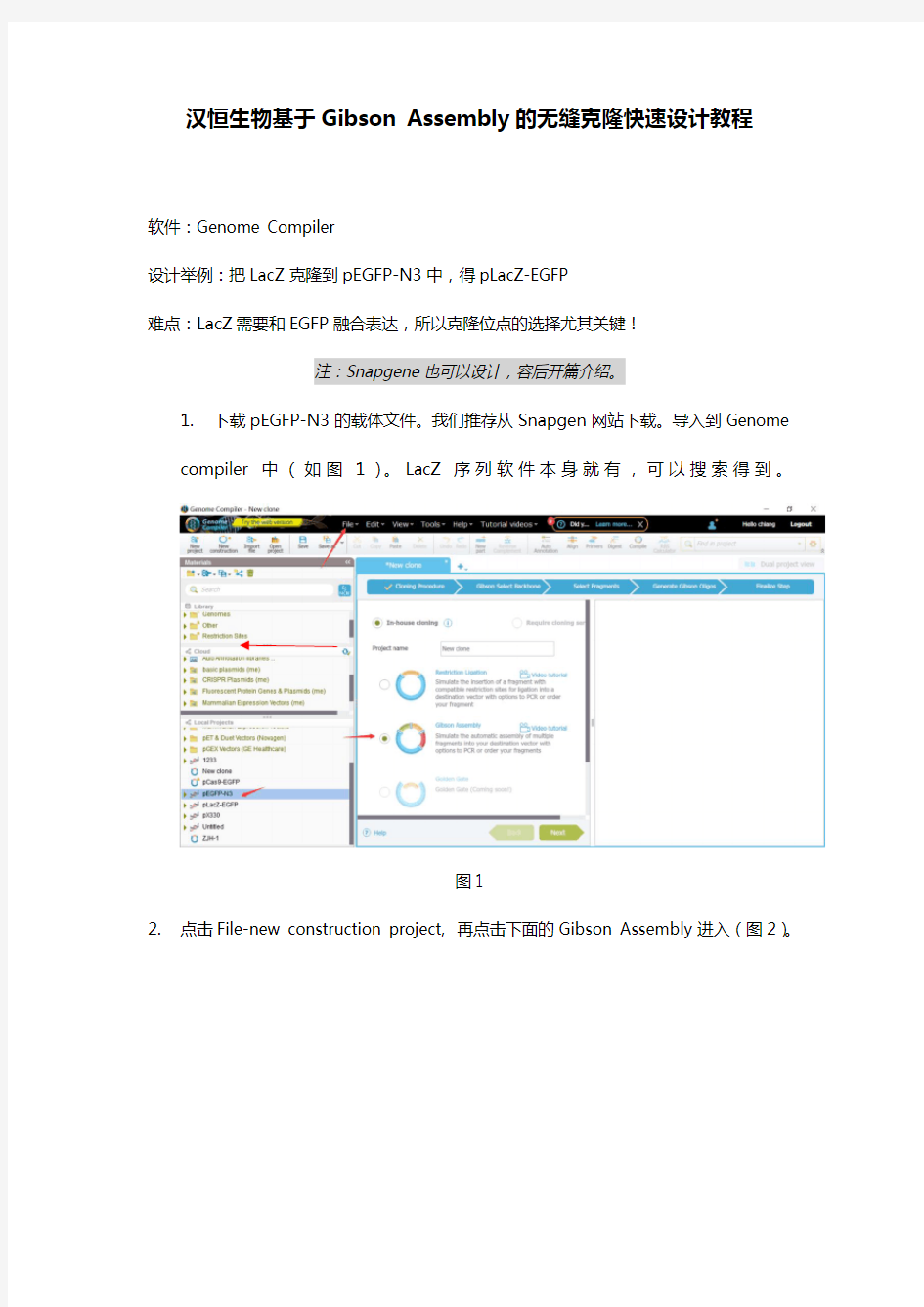
1.下载pEGFP-N3的载体文件。我们推荐从Snapgen网站下载。导入到
Genomecompiler中(如图1)。LacZ序列软件本身就有,可以搜索得到。
图1
2.点击File-new construction project, 再点击下面的Gibson Assembly进入(图2)。
图2
3.把BackbonepEGFP-N3拖入,载体信息会完全展示出来,包括序列、图谱和各种注
释(图3-4)。点击sequence和circular可以在质粒的序列和图谱之间切换,方便查看。图4所示的是我们选中对多克隆位点MCS的序列展示。
图3
图4
4.图4之间有一些选项:快速克隆首先需要对载体进行线性化处理。通常有2种方法:
双酶切线性化和PCR线性化。本次教程我们来讲双酶切线性化。
5.本例是做融合蛋白的克隆构建,所以必须考虑读码框的问题。简而言之,就是要融合表
达的两个蛋白之间间隔的碱基数必需是3的倍数,比如9bp,15bp,21bp这样子,而且不能含有终止密码子Stop Codon-TAA,TAG和TGA。
6.鉴于此,我们选择双酶切的时候一定要足够小心,确保满足第6条的标准。比如在本
例中我们选择NheI和BamHI(如图5),BamHI识别序列为GGATCC6碱基限制性内切酶,编码两个氨基酸G和S,因此,可以作为读码框的一部分。而BamHI识别位点GGATCC距离下游GFP的举例为15bp,刚好包含5个氨基酸GSIAT(如图15)。如果不能完美对框,比如距离为14bp,则需要在引物合成的时候补上1个(非终止密码子)碱基,如果是13bp,则需要补2个(非终止密码子)碱基等。
图5
7.然后我们选中NheI,会自动出来下面的选项,酶切位点的处理方式,包括不处理Non
processed(不处理),regenerate(再生)和Eliminate(去除)。我们选择regenerate(再生),这样保证构建的克隆酶切位点可以恢复(图6)。同样的,下游的酶切位点也同样处理。然后点击下一步。
图6
8.这一步是选择insert(插入片段)。我们选择”Addfragment“(图7)。把LacZ文件
拖进去(图8).中间可以对插入片段重命名,位置也可以自由定制(本例中选择的时候注意不要带Stop Codon)。本例LacZ序列不带stopcondon,我们选择selectall 即可。如果是做多片段组装,方法同上。需要对齐读码框的请一定格外小心。点击下一步。
图7
图8
9.这一步就进入引物设计设计阶段。默认重叠序列15-25bp,TM值在50度(图9)。
点击runassembly。引物序列就设计好了(图10).但我们不着急,继续下一步。
图9
图10
10.下一步就把克隆虚拟组装好了(图11)。但我们一定要check一下读码框是否和我们
预想的一样正确。我们选中LacZ-EGFP序列。然后选中Annotationlayers,点击ORF
旁边的小齿轮进入设置(图12,13)。因为这个融合读码框很大(超多600个氨基酸),我们范围也选定到大于600aa。点击确认(图14)。
图11
图12
图13
图14
11.图14图谱中会出现一个环形箭头,代表一个ORF。我们切换到序列,找到LacZ和EGFP
的融合出看看细节(图15)。箭头分别指示了3个ORF—LacZ,EGFP和LacZ-EGFP 融合,发现融合以后两者的读码框严格保留,而中间就多了GSIAT 5个氨基酸。说明和我们预想的一致,设计ok。点击完成即可。
图15
12.完了保存。引物会在图谱里面显示出来。切换到线性图谱模式,选中引物,右键选择
“copyprimercontent“到文件里就是引物序列。直接合成即可,但请check引物方向和正义链反义链(图16)。
图16
13.然后引物合成回来以后按照说明书做克隆构建即可。
图解blast验证引物教程 1、进入网页:https://www.doczj.com/doc/ec2572381.html,/BLAST/ 2、点击Search for short, nearly exact matches 3、在search栏中输入引物系列: 注:文献报道ABCG2的引物为5’-CTGAGATCCTGAGCCTTTGG-3’; 5’-TGCCCATCACAACATCATCT-3’ (1)输入方法可先输入上游引物,进行blast程序,同样方法在进行下游引物的blast程序。这种方法叫繁琐,而且在结果分析特异性时要看能与上游引物的匹配的系列,还要看与下游引物匹配的系列——之后看两者的交叉。 (2)简便的做法是同时输入上下游引物:有以下两种方法。输入上下游引物系列都从5’——3’。 A、输入上游引物空格输入下游引物
B、输入上游引物回车输入下游引物 4、在options for advanced blasting中: select from 栏通过菜单选择Homo sapiens【ORGN】Expect后面的数字改为10 5、在format中: select from 栏通过菜单选择Homo sapiens【ORGN】Expect后面的数字填上0 10
6、点击网页中最下面的“BLAST!” 7、出现新的网页,点击Format! 果。
(1)图形格式: 图中①代表这些序列与上游引物匹配、并与下游引物互补的得分值都位于40~50分 图中②代表这些序列与上游引物匹配的得分值位于40~50分,而与下游引物不互补 图中③代表这些序列与下游引物互补的得分值小于40分,而与上游引物不匹配 通过点击相应的bar可以得到匹配情况的详细信息。 (2)结果信息概要: 从左到右分别为: A、数据库系列的身份证:点击之后可以获得该序列的信息 B、系列的简单描述 C、高比值片段对(high-scoring segment pairs, HSP)的字符得分。按照得分的高低由大到小排列。得分的计算公式=匹配的碱基×2+0.1。举例:如果有20个碱基匹配,则其得分为40.1。 D、E值:代表被比对的两个序列不相关的可能性。【The E value decreases exponentially as the Score (S) that is assigned to a match between two sequences increases】。E值最低的最有意义,也就是说序列的相似性最大。设定的E值是我们限定的上限,E值太高的就不显示了 E、最后一栏有的有UEG的字样,其中: U代表:Unigene数据库 E代表:GEO profiles数据库 G代表:Gene数据库
Primer 5.0搜索引物: 1.Primer Length我常设置在18-30bp,短了特异性不好,长了没有必要。当然有特殊要求的除外,如加个酶切位点什么的。 2.PCR Product size最好是100-500bp之间,小于100bp的PCR产物琼脂糖凝胶电泳出来,条带很模糊,不好看。至于上限倒也不必要求苛刻。 3.Search parameters还是选Manual吧,Search stringency应选High,GC含量一般是40-60%。其它参数默认就可以了。 4.搜索出来的引物,按Rating排序,逐个送Oligo软件里评估。当然,搜索出的引物,其扩增产物很短,你可以不选择它,或是引物3端≥2个A或T,或引物内部连续的G或C太多,或引物3端≥2个G或C,这样的引物应作为次选,没得选了就选它。对于这样的引物,如果其它各项指标还可以,我喜欢在引物末端去掉一个不满意的或加上一个碱基,看看引物的评估参数有没有变好点。 Oligo 6.0评估引物: 1.在analyze里,Duplex Formation不管是上游引物、下游引物还是上下游引物之间,The most stable 3’-Dimer绝对值应小于4.5kcal/mol, The most stable Dimer overall绝对值一般应小于多少kcal/mol跟PCR退火温度有关,我几次实验感觉在PCR退火温度在65°的时候,The most stable Dimer ove rall 6.7kcal/mol没有问题。 2.Hairpin Formation根据黄金法则 3.False priming sites: Primer的priming efficiency应该是错配地方的4倍左右,更多当然更好。 4.在PCR栏,个人感觉其所显示的optimal annealing temperature数值值得参考。在PCR摸索条件的时候,退火温度为其数值加减2的范围就可以了。 5.Internal stability很重要:我们希望引物的内部稳定性是中间高、两边低的弧形,最起码保证3端不要过于稳定。下图1引物3端过于稳定,很容易导致不适当扩增。△G参照黄金法则,这其实很好理解:把一滴水放到大海里,这滴水就会不停的扩散分布,扩散的越厉害越稳定,所以△G绝对值越大结构越稳定。 最后说一句,敢于尝试就会成功。 第二贴 --科室工作很多,小医生了,没有办法,所以肯怕不能满足很多战友的要求(qq聊或帮助设计),在此表示抱歉。就楼上的问题我试着回答一下,不一定正确,供参考吧。 --1、两个评价系统不一样,个人感觉oligo评价引物好点,primer出来的引物,我一般按效率排序,再结合退火温度和引物长度,选择引物到oligo测试。这是初步的选择,其实引物到了oligo里,退火温度也不一样。 --2、3端的二聚体应该避免,这个要看你的退火温度决定,一个50°的退火温度肯定和65°对二聚体的影响不一样了,一般来讲尽量控制在-4.5kcal/mol以下(个人观点,很多东西真得还是需要自己摸索)。 --3、个人感觉3端有A无A影响不大,3端有T的没有经验。有T是不是一定不行,个人感觉不见得。软件是评估,法则也不是没有例外,不是1+1=2那么确定。 --4、错配和二聚体谁轻谁重,个人觉得“到致命的程度”谁都重要,我也说不好。我设计的时候,尽量两个都不得罪。 --5、GC含量并非不重要,它直接影响引物各端稳定性,3端来两个G或C,稳定性就上去了,粘在模板上很牢。所以我设计的时候,尽量避免这样的情况出现。 谈一下我学这个引物设计的过程吧:
引物设计和Primer-BLAST的应用 Lv Peng 2015.11.18
CONTENT 1.PCR-引物设计目的 2.引物设计原则 3.设计引物软件 4.在线设计工具 5.probeBase 简介
1.1PCR(Polymerase Chain Reaction) 聚合酶链式反应 1971 Khorana 提出设想 1985 Kary Mullis 发明了PCR 1986年5月 Mullis在冷 泉港实验室 做专题报告 冷泉港实验室(The Cold Spring Harbor Laboratory,缩写CSHL),又译为科尔德斯普林实验室。
几不同的PCR技术 1.扩增已知序列两侧DNA的PCR:反向PCR(Inverse PCR,IPCR)、锚定PCR(anchored PCR)、RACE(Rapid Amplification of cDNA Ends)、连接介导的PCR(ligation-mediated PCR,LM-PCR); 2.检测有限量稀有靶序列,即一对引物扩增产物不足以以通过凝胶电泳观察到的时:巢式PCR(nested PCR); 3.快速、灵敏、特异而准确定量的PCR:实时荧光定量PCR (real-time quantitative PCR,RQ-PCR)。
特性 优化 碱基组成 (G+C )含量应在40%-60%,4种碱基要分布均匀;长度 一般为18-27个核苷酸长度。上下游引物长度差别不能大于3bp ;重复和自身互补序列 不能有大于3bp 的反向重复序列或自身互补序列存在;上下游引物互补性一个引物的3’末端序列不能结合到另一个引物的任何位点上; 解链温度(Tm ) 两个引物的Tm 值相差不能大于5℃,扩增产物与引物的Tm 值相差不能大于10℃3’末端 引物3’末端碱基尽量为G 或C ,不能使3’末端有NNGC 或NNCG 序列引物序列不要有局部的GC rich 或AT rich (特别是3’端),避开T/C 或A/G 的连续结构 1.2引物设计原则 引物特性及优化设计
mi引物设计原则 1. 引物的长度一般为15-30 bp,常用的是18-27 bp,但不应大于38,因为过长会导致其延伸温度大于74℃,不适于Taq DNA聚合酶进行反应。 2. 引物序列在模板内应当没有相似性较高,尤其是3’端相似性较高的序列,否则容易导致错配。引物3’端出现3个以上的连续碱基,如GGG或CCC,也会使错误引发机率增加。 3. 引物3’端的末位碱基对Taq酶的DNA合成效率有较大的影响。不同的末位碱基在错配位置导致不同的扩增效率,末位碱基为A的错配效率明显高于其他3个碱基,因此应当避免在引物的3’端使用碱基A。另外,引物二聚体或发夹结构也可能导致PCR反应失败。5’端序列对PCR影响不太大,因此常用来引进修饰位点或标记物。 4. 引物序列的GC含量一般为40-60%,过高或过低都不利于引发反应。上下游引物的GC含量不能相差太大。 5. 引物所对应模板位置序列的Tm值在72℃左右可使复性条件最佳。Tm值的计算有多种方法,如按公式Tm=4(G+C)+2(A+T),在Oligo软件中使用的是最邻近法(the nearest neighbor method)。 6. ΔG值是指DNA双链形成所需的自由能,该值反映了双链结构内部碱基对的相对稳定性。应当选用3’端ΔG值较低(绝对值不超过9),而5’端和中间ΔG 值相对较高的引物。引物的3’端的ΔG值过高,容易在错配位点形成双链结构并引发DNA聚合反应。 7. 引物二聚体及发夹结构的能值过高(超过4.5kcal/mol)易导致产生引物二聚体带,并且降低引物有效浓度而使PCR反应不能正常进行。 8. 对引物的修饰一般是在5’端增加酶切位点,应根据下一步实验中要插入PCR 产物的载体的相应序列而确定。 引物序列应该都是写成5-3方向的, Tm之间的差异最好控制在1度之内, 另外我觉得扩增长度大一些比较好,500bp左右。 要设计引物首先要找到DNA序列的保守区。同时应预测将要扩增的片段单链是否形成二级结构。如这个区域单链能形成二级结构,就要避开它。如这一段不能
引物设计的11条黄金法则
PCR引物设计的11条黄金法则 1.引物最好在模板cDNA的保守区内设计。DNA序列的保守区是通过物种间相似序列的比较确定的。在NCBI上搜索不同物种的同一基因,通过序列分析软件(比如DNAman)比对(Alignment),各基因相同的序列就是该基因的保守区。 2.引物长度一般在15~30碱基之间。 引物长度(primerlength)常用的是18-27bp,但不应大于38,因为过长会导致其延伸温度大于74℃,不适于TaqDNA聚合酶进行反应。 3.引物GC含量在40%~60%之间,Tm值最好接近72℃。 GC含量(composition)过高或过低都不利于引发反应。上下游引物的GC含量不能相差太大。另外,上下游引物的Tm值(meltingtemperature)是寡核苷酸的解链温度,即在一定盐浓度条件下,50%寡核苷酸双链解链的温度。有效启动温度,一般高于Tm值
5~10℃。若按公式Tm=4(G+C)+2(A+T)估计引物的Tm值,则有效引物的Tm为55~80℃,其Tm值最好接近72℃以使复性条件最佳。 4.引物3′端要避开密码子的第3位。 如扩增编码区域,引物3′端不要终止于密码子的第3位,因密码子的第3位易发生简并,会影响扩增的特异性与效率。 5.引物3′端不能选择A,最好选择T。 引物3′端错配时,不同碱基引发效率存在着很大的差异,当末位的碱基为A时,即使在错配的情况下,也能有引发链的合成,而当末位链为T 时,错配的引发效率大大降低,G、C错配的引发效率介于A、T之间,所以3′端最好选择T。 6.碱基要随机分布。 引物序列在模板内应当没有相似性较高,尤其是3’端相似性较高的序列,否则容易导致错误引发(Falsepriming)。降低引物与模板相似性的一种方法是,引物中四种碱基的分布最好是随机的,不要有聚嘌呤或聚嘧啶的存在。尤其3′端
核酸环介导等温扩增技术(LAMP)引物设计与实例Time:2009-12-07 PM 14:52 Author:bioer Hits: 1459 times 烟头整理 LAMP的特点 LAMP与以往的核酸扩增方法相比具有如下优点: (1)操作简单 LAMP核酸扩增是在等温条件下进行,对于中小医院只需要水浴锅即可,产物检测用肉眼观察或浊度仪检测沉淀浊度即可判断。对于RNA的扩增只需要在反应体系中加入逆转录酶就可同步进行(RT-LAMP),不需要特殊的试剂及仪器。 (2)快速高效 因为不需要预先的双链DNA热变性,避免了温度循环而造成的时间损失。核酸扩增在l h内均可完成,添加环状引物后时间可以节省1/2,多数情况在20-30 rain均可检测到扩增产物。且产物可以扩增至109倍,达0.5 mg/mL。应用专门的浊度仪可以达到实时定量检测。 (3)高特异性 由于是针对靶序列6个区域设计的4种特异性引物。6个区域中任何区域与引物不匹配均不能进行核酸扩增。故其特异性极高。 (4)高灵敏度 对于病毒扩增模板可达几个拷贝,比PCR高出数量级的差异。 缺点: 由于LAMP扩增是链置换合成,靶序列长度最好在300 bp以内。>500 bp则较难扩增。故不能进行长链DNA的扩增。由于灵敏度高。极易受到污染而产生假阳性结果。故要特别注意严谨操作,以及在产物的回收鉴定、克隆、单链分离方面均逊色于传统的PCR 方法。 引物设计实例 LAMP引物设计的在线网站(http://primerexplorer.jp/e/),只要导入靶基因就能自动生成成组引物。 以某一微生物的鞭毛基因为例讲解一下LAMP引物设计的过程: 首先单击浏览按钮选择靶基因序列文件,靶序列默认的是小于22 kbp。支持三个类型的文件,普通文本格式(仅含序列), FASTA格式和GenBank 格式文件。
1.引物最好在模板cDNA的保守区内设计。 DNA序列的保守区是通过物种间相似序列的比较确定的。在NCBI上搜索不同物种的同一基因,通过序列分析软件(比如DNAman)比对(Alignment),各基因相同的序列就是该基因的保守区。 2.引物长度一般在15~30碱基之间。 引物长度(primer length)常用的是18-27 bp,但不应大于38,因为过长会导致其延伸温度大于74℃,不适于Taq DNA 聚合酶进行反应。 3.引物GC含量在40%~60%之间,Tm值最好接近72℃。 GC含量(composition)过高或过低都不利于引发反应。上下游引物的GC含量不能相差太大。另外,上下游引物的Tm值(melting temperature)是寡核苷酸的解链温度,即在一定盐浓度条件下,50%寡核苷酸双链解链的温度。有效启动温度,一般高于Tm值5~10℃。若按公式Tm= 4(G+C)+2(A+T)估计引物的Tm值,则有效引物的Tm为55~80℃,其Tm 值最好接近72℃以使复性条件最佳。 4.引物3′端要避开密码子的第3位。 如扩增编码区域,引物3′端不要终止于密码子的第3位,因密码子的第3位易发生简并,会影响扩增的特异性与效率。 5.引物3′端不能选择A,最好选择T。 引物3′端错配时,不同碱基引发效率存在着很大的差异,当末位的碱基为A时,即使在错配的情况下,也能有引发链的合成,而当末位链为T时,错配的引发效率大大降低,G、C 错配的引发效率介于A、T之间,所以3′端最好选择T。 6. 碱基要随机分布。 引物序列在模板内应当没有相似性较高,尤其是3’端相似性较高的序列,否则容易导致错误引发(False priming)。降低引物与模板相似性的一种方法是,引物中四种碱基的分布最好是随机的,不要有聚嘌呤或聚嘧啶的存在。尤其3′端不应超过3个连续的G或C,因这样会使引物在GC富集序列区错误引发。 7. 引物自身及引物之间不应存在互补序列。 引物自身不应存在互补序列,否则引物自身会折叠成发夹结构(Hairpin)使引物本身复性。这种二级结构会因空间位阻而影响引物与模板的复性结合。引物自身不能有连续4个碱基的互补。 两引物之间也不应具有互补性,尤其应避免3′ 端的互补重叠以防止引物二聚体(Dimer与Cross dimer)的形成。引物之间不能有连续4个碱基的互补。 引物二聚体及发夹结构如果不可避免的话,应尽量使其△G值不要过高(应小于4.5kcal/mol)。否则易导致产生引物二聚体带,并且降低引物有效浓度而使PCR 反应不能正常进行。 8. 引物5′ 端和中间△G值应该相对较高,而3′ 端△G值较低。 △G值是指DNA 双链形成所需的自由能,它反映了双链结构内部碱基对的相对稳定性,△G 值越大,则双链越稳定。应当选用5′ 端和中间△G值相对较高,而3′ 端△G值较低(绝对值不超过9)的引物。引物3′ 端的△G 值过高,容易在错配位点形成双链结构并引发DNA 聚合反应。(不同位置的△G值可以用Oligo 6软件进行分析) 9.引物的5′端可以修饰,而3′端不可修饰。 引物的5′ 端决定着PCR产物的长度,它对扩增特异性影响不大。因此,可以被修饰而不影响扩增的特异性。引物5′ 端修饰包括:加酶切位点;标记生物素、荧光、地高辛、Eu3+等;引入蛋白质结合DNA序列;引入点突变、插入突变、缺失突变序列;引入启动子序列等。引物的延伸是从3′ 端开始的,不能进行任何修饰。3′ 端也不能有形成任何二级结构可能。 10. 扩增产物的单链不能形成二级结构。
引物设计step by step 1、在NCBI上搜索到目的基因,找到该基因的mRNA,在CDS选项中,找到编码区所在位置,在下面的origin中,Copy该编码序列作为软件查询序列的候选对象。 2、用Primer Premier5搜索引物 ①打开Primer Premier5,点击File-New-DNA sequence, 出现输入序列窗口,Copy目的序列在输入框内(选择As),此窗口内,序列也可以直接翻译成蛋白。点击Primer,进入引物窗口。 ②此窗口可以链接到“引物搜索”、“引物编辑”以及“搜索结果”选项,点击Search按钮,进入引物搜索框,选择“PCR primers”,“Pairs”,设定搜索区域和引物长度和产物长度。在Search Parameters里面,可以设定相应参数。一般若无特殊需要,参数选择默认即可,但产物长度可以适当变化,因为100~200bp的产物电泳跑得较散,所以可以选择300~500bp. ③点击OK,软件即开始自动搜索引物,搜索完成后,会自动跳出结果窗口,搜索结果默认按照评分(Rating)排序,点击其中任一个搜索结果,可以在“引物窗口”中,显示出该引物的综合情况,包括上游引物和下游引物的序列和位置,引物的各种信息等。 ④对于引物的序列,可以简单查看一下,避免出现下列情况:3’不要出现连续的3个碱基相连的情况,比如GGG或CCC,否则容易引起错配。此窗口中需要着重查看的包括:Tm 应该在55~70度之间,GC%应该在45%~55%间,上游引物和下游引物的Tm值最好不要相差太多,大概在2度以下较好。该窗口的最下面列出了两条引物的二级结构信息,包括,发卡,二聚体,引物间交叉二聚体和错误引发位置。若按钮显示为红色,表示存在该二级结构,点击该红色按钮,即可看到相应二级结构位置图示。最理想的引物,应该都不存在这些二级结构,即这几个按钮都显示为“None”为好。但有时很难找到各个条件都满足的引物,所以要求可以适当放宽,比如引物存在错配的话,可以就具体情况考察该错配的效率如何,是否会明显影响产物。对于引物具体详细的评价需要借助于Oligo来完成,Oligo自身虽然带有引物搜索功能,但其搜索出的引物质量感觉不如Primer5. ⑤在Primer5窗口中,若觉得某一对引物合适,可以在搜索结果窗口中,点击该引物,然后在菜单栏,选择File-Print-Current pair,使用PDF虚拟打印机,即可转换为Pdf文档,里面有该引物的详细信息。 3、用Oligo验证评估引物 ①在Oligo软件界面,File菜单下,选择Open,定位到目的cDNA序列(在primer中,该序列已经被保存为Seq文件),会跳出来两个窗口,分别为Internal Stability(Delta G)窗口和Tm窗口。在Tm窗口中,点击最左下角的按钮,会出来引物定位对话框,输入候选的上游引物序列位置(Primer5已经给出)即可,而引物长度可以通过点击Change-Current oligo length来改变。定位后,点击Tm窗口的Upper按钮,确定上游引物,同样方法定位下游引物位置,点击Lower按钮,确定下游引物。引物确定后,即可以充分利用Analyze 菜单中各种强大的引物分析功能了。
1-2890(引物1) #1: Product of length 640 (rating: 171) Contains region of the molecule from 1 to 640 Tm: 72.1 C TaOpt: 48.8 C GC: 32.3 Sense Primer: CCTGGTTAATCCAAATCAC Similarity: 100.0% Length: 19 Tm: 44.1 C GC: 42.1 dH: -142.0 kcal/mol dS: -372.4 cal/mol dG: -29.2 kcal/mol Antisense Primer: GACAGGCCCTAATTAAGTT Similarity: 100.0% Length: 20 Tm: 45.0 C GC: 42.1 dH: -158.0 kcal/mol dS: -418.4 cal/mol dG: -31.5 kcal/mol Tm Difference: 0.9 GC Difference: 0 #1: Product of length 540 (rating: 171) Contains region of the molecule from 1 to 540 Tm: 72.2 C TaOpt: 49.4 C GC: 33.1 Sense Primer: CCTGGTTAATCCAAATCACT Similarity: 100.0% Length: 20 Tm: 45.8 C GC: 40.0 dH: -149.8 kcal/mol dS: -393.2 cal/mol dG: -30.8 kcal/mol Antisense Primer: ATAAGATTTGAGGTCAGCCA Similarity: 100.0% Length: 20 Tm: 46.4 C GC: 40.0 dH: -147.7 kcal/mol dS: -386.3 cal/mol dG: -30.7 kcal/mol Tm Difference: 0.6 GC Difference: 0.0 1-2890(引物2) #1: Product of length 603 (rating: 171) Contains region of the molecule from 514 to 1116 Tm: 73.9 C TaOpt: 50.3 C GC: 37.0 Sense Primer: TTGAAGATGGCTGACCT Similarity: 100.0% Length: 18 Tm: 42 C GC: 47.1 dH: -129.6 kcal/mol dS: -335.0 cal/mol dG: -27.9 kcal/mol Antisense Primer: GGAGGCCCTTTAACTTAA
PCR引物设计原则 引物(Primer)是人工合成的两段寡核苷酸序列。 1、引物的长度一般为15-30bp,常用的是18-27bp,但不应大于38,因为过长会导致其延伸温度大于74℃,不适于Taq DNA聚合酶进行反应。 2、G十C含量:应在40%-60%之间,PCR扩增中的复性温度一般是较低Tm 值引物的Tm值减去5-10度。引物长度小于20时,其Tm恒等于4(G十C)十2(A十T)。 3、碱基分布的随机性:应避免连续出现4个以上的单一碱基。尤其是不应在其3’端出现超过3个的连续G或C,否则会使引物在G十C富集序列区错误引发. 4、引物自身:不能含有自身互补序列,否则会形成发夹样二级结构. 5、引物之间:两个引物之间不应有多于4个的互补或同源碱基,不然会形成引物二聚体,尤应避免3’端的互补重叠。引物3’端最好选T,错配的几率与A 相比大大的降低了。G、C之间错配的概率小于A、T. 6、引物的5’端可以修饰,而3’端不能进行修饰。5’端的修饰包括:加酶切位点,标记生物素,荧光,地高辛、Eu3+等,引入蛋白质结合的DNA序列,引入点突变,插入突变、缺失突变序列、引入启动子序列。因为引物的延伸是从3’端开始的,因而3’端不能进行任何修饰,另外3’端也不能有形成任何二
级结构的可能。 如何设计引物 不同的核苷酸序列表达的氨基酸氨基酸序列是相同的,所以氨基酸序列才是真正保守的。 引物最好在模板cDNA的保守区域内设计(DNA的保守区是通过物种间相似序列的比较确定的,在NCBI上搜索不同物种的同一基因,通过序列分析软件比对(Alignment),各基因相同的序列就是该基因的保守区)。 PCR引物设计 PCR反应中有两条引物,即5′端引物和3′引物。设计引物时以一条DNA单链为基准(常以信息链为基准),5′端引物与位于待扩增片段5′端上的一小段DNA序列相同;3′端引物与位于待扩增片段3′端的一小段DNA序列互补。 引物设计软件 Primer Premier5.0 (自动搜索)* vOligo6 (引物评价) vVector NTI Suit vDNAsis vOmiga vDNAstar vPrimer3 (在线服务)
本文叙述了一种用于甲基化分析的探针法定量PCR的引物和探针设计方法,目前用于甲基化检测的引物探针设计工具非常多,都有使用成功的案例,经过初步多方尝试,本文中叙述的为本人认为较为靠谱的方法。Oligo7的优势在于专业,参数详尽且可自由设置,模块化设计,学会后使用便利。专业的活就是要专业的用专业的工具干。
首先是进行序列转换,有较多的在线工具和联机软件都可实现,这里使用https://www.doczj.com/doc/ec2572381.html,/methprimer/,较为简单直观。
直接将目标序列放入如上图的编辑框中,此也可直接用于相关引物的设计,不过本人没使用过,因为不能设计探针。submit后就有转化后的序列信息,如下图: 以上详细标记了CpG位置和非CpG位置的C,可直接复制到Word标注使用,下面就可以使用Oligo7利用上边的序列设计引物和探针了,如果是设计非甲基化引物探针,则使用原始序列。
关于引物和探针的一些主要参数,主要参考invtrogen的建议: Primer设计的基本原则: a)引物长度一般在18-35mer。 b)G-C含量控制在40-60%左右。 c)避免近3’端有酶切位点或发夹结构。 d)如果可能避免在3’端最后5个碱基有2个以上的G或C。 e)如果可能避免在3’端最后1个碱基为A。 f)避免连续相同碱基的出现,特别是要避免GGGG或更多G出现。 g)退火温度Tm控制在58-60C左右。 h)如果是设计点突变引物,突变点应尽可能在引物的中间。 T aqMan 探针设计的基本原则: a)T aqMan 探针位置尽可能靠近扩增引物(扩增产物50-150bp),但不能与引物重叠。 b)长度一般为18-40mer 。 c)G-C含量控制在40-80%左右。 d)避免连续相同碱基的出现,特别是要避免GGGG或更多G出现。 e)在引物的5’端避免使用G。 f)选用比较多的碱基C。 g)退火温度Tm控制在68-70℃左右。 另:目标变异碱基最好在3’末端或3’末端-1位置,保证扩增特异性,对于甲基化,则最好是C。
DNA测序结果分析比对(实例) 关键词:dna测序结果2013-08-22 11:59来源:互联网点击次数:14423 从测序公司得到的一份DNA测序结果通常包含.seq格式的测序结果序列文本和.ab1格式的测序图两个文件,下面是一份测序结果的实例: CYP3A4-E1-1-1(E1B).ab1 CYP3A4-E1-1-1(E1B).seq .seq文件可以用系统自带的记事本程序打开,.ab1文件需要用专门的软件打开。软件名称:Chromas 软件Chromas下载 .seq文件打开后如下图: .ab1文件打开后如下图: 通常一份测序结果图由红、黑、绿和蓝色测序峰组成,代表不同的碱基序列。测序图的两端(下图原图的后半段被剪切掉了)大约50个碱
基的测序图部分通常杂质的干扰较大,无法判读,这是正常现象。这也提醒我们在做引物设计时,要避免将所研究的位点离PCR序列的两端太近(通常要大于50个碱基距离),以免测序后难以分析比对。 我的课题是研究基因多态性的,因此下面要介绍的内容也主要以判读测序图中的等位基因突变位点为主。 实际上,要在一份测序图中找到真正确实的等位基因多态位点并不是一件容易的事情。一般认为等位基因位点假如在测序图上出现像套叠的两个峰,就是杂合子位点。实际比对后才知道,情况并非那么简单,下面测序图中标出的两个套峰均不是杂合子位点,如图并说明如下:
说明: 第一组套峰,两峰的轴线并不在同一位置,左侧的T峰是干扰峰;第二组套峰,虽两峰轴线位置相同,但两峰的位置太靠近了,不是杂合子峰,蓝色的C峰是干扰峰通常的杂合子峰由一高一略低的两个轴线相同的峰组成,此处的序列被机器误判为“C”,实际的序列应为“A”,通常一个高大碱基峰的前面 1~2个位点很容易产生一个相同碱基的干扰峰,峰的高度大约是高大碱基峰的1/2,离得越近受干扰越大。 一个摸索出来的规律是:主峰通常在干扰峰的右侧,干扰峰并不一定比主峰低。最关键的一点是一定要拿疑似为杂合子峰的测序图位点与测序结果的文本序列和基因库中的比对结果相比较;一个位点的多个样本相比较;你得出的该位点的突变率与权威文献或数据库中的突变率相比较。 通常,对于一个疑似突变位点来说,即使是国际上权威组织大样本的测序结果中都没有报道的话,那么单纯通过测序结果就判定它是突变点,是并不严谨的,因一份 PCR产物中各个碱基的实际含量并不相同,很难避免不产生误差的。对于一个未知突变位点的发现,通常还需要用到更精确的酶切技术。 (责任编辑:大汉昆仑王)
引物设计原则及酶切位点选择和设计 [整理]:最初的时候,由于害怕设计酶切位点最后且不开,所以经常采用最通用的方法,用T载体克隆解决问题,但后来发现她也有问题,就是浓度提不上去,你需要体大量的载体来酶切,所以感到还是直接扩增好一点。但这就需要你仔细设计引物。连入质粒中的重要目的就是进行酶切和连接,当然首先就是在想要合成或者是进行PCR扩增出靶基因的时候在核酸的两端接入酶切位点,酶切位点是与你的质粒的特点相关的,可以在质粒的图谱说明书上找取相应的位点,进行设计。 (一)设计引物前应做的准备工作: 准备载体图谱,大致准备把片断插在那个部分 对片断进行酶切分析,确定一下那些酶切位点不能用 准备一本所买公司的酶的商品目录,便于查酶的各种数据及两种酶是否可以配用 (二)设计引物所要考虑的问题 两个位点应是载体上的,,所连接片断上没有这两个位点,且距离不能太近,往往导致两个酶都切不好。因此,紧挨在一起,只能切一个,除非恰好是与上面两个酶在一起的酶切位点。我看promega的说明书上说,最好隔四个。还有一种情况是:不能有碱基的交叉,比如AGATCTTAAG,这样的位点比较难切。 两个酶切点最好不要是同尾酶(切下来的残基不要互补),否则效果相当于单酶切。 最好使用酶切效率高的。 最好使用双酶切有共同buffer的酶。 最好使用较常用的酶(如hind3,bamh1,ecor1等),最好使用自己实验室有的酶,这样可以省钱。 Tm的计算,关于Tm的问题,很多的战友都有疑惑。其实园子里有很多的解释了。 Tm叫溶解温度(melting temperature, Tm),即是DNA双链溶解所需的温度。大家可以理解,这个温度是由互补的DNA区域决定的,而不互补的区域对DNA的溶解是没有作用的。因此,对于引物的Tm,只有和模板互补的区域对Tm才有贡献。计算Tm时,只计算互补的区域(除非你的酶切位点也与模板互补)。不少战友设计的引物都Tm过低,是因为他们误把保护碱基和酶切位点都计算到Tm里了,最后的结果是导致了PCR反应的诸多困难。所以,设计引物的时候,先不管5'端的修饰序列,把互补区的Tm控制在55度以上(我喜欢控制在58以上,具体根据PCR的具体情况,对于困难的PCR,需要适当提高Tm),再加上酶切位点和保护碱基,这样的引物通常都是可用的,即使有小的问题,也可以挽回。Tm温度高的引物就比较容易克服3‘发卡、二聚体及3'非特异结合等问题。简单的计算公式可以用2+4的公式。若你计算的Tm值达到了快90 ,不包括酶切位点。引物公司给你发的单子是包括酶切位点的。自己可以再估计一下。如你设计了带酶切位点的引物,总长分别为29、33个碱基,去掉酶切位点和保护碱基,分别为17、21个碱基。引物公司给的单子是70多度,实际用的只有50度,用55度扩的结果也差不多。 其它关于Tm值的计算,有用PP5.0进行评价的,需要考虑的参数包括:base number、GC%、Tm、hairpin、dimer、false priming、cross dimer。退一般退火温度为Tm-5度,退火温度的计算可以不把加入的酶切位点及保护碱基考虑进去,如上所言,PCR几个循环后,引物外侧的序列已经参入了扩增片断中,所以你可以在预变性后多加几步,温度比你Tm值低些(这样可能会增加非特异性),Tm值是你包括酶切位点及保护碱基的Primer计算出来的。1.一般在5'端加保护碱基,如果你扩增后把目的条带做胶回收转入T-VECTOR或者其它的载体的话,酶切时可以不需加保护碱基2.有人的经验加入酶切位点的引物可以和未加入时使用相同的退火温度,结果也还是令人满意。
Oligo 设计教程 在正式进行引物设计前,我们首先面临的一个任务就是向Oligo 程序导入模板序列,根据不同的实验情况,导入模板有三种方法:1,直接用键盘输入: a,点击file菜单中的New Sequence 浮动命令,或直接点击工具栏中的New Sequence命令,进入序列展示窗口; b,此时即可键入DNA序列; c,如果需要的话,Oligo提供碱基回放功能,在边键入时边读出碱基,防止输入错误。点击Edit菜单中的“Readback on”即可。 2,利用复制和粘贴:当我们序列已经作为TXT文件存在或其它oligo不能直接open的文件格式,如word文件.html格式,这个功能就显得很有用了。在相应文件中复制序列后在序列展示窗口粘贴,oligo会自动去除非碱基字符。当序列输入或粘贴完成后,点击Accept/Discard菜单中的Accept浮动命令,即可进入引物设计模式。 3,如果序列已经保存为Seq格式或者FASTA,GenBank格式时,oligo就可以直接打开序列文件。 点击File菜单中的“Open”浮动命令,找到所需文件,打开即可。
进入引物设计模式后,oligo一般会弹出三个窗口,分别是6-碱基频率窗口,碱基退火温度窗口以及序列内部碱基稳定性窗口,其中的退火温度窗口是我们引物设计的主窗口,其它的两个窗口则在设计过程中起辅助作用,比如6-碱基频率窗口可以使我们很直观地看到所设计引物在相应物种基因组中的出现频率,如果我们的模板是基因组DNA或混合DNA时,该信息就显得有用了,而内部稳定性窗口则可以显示引物的5’端稳定性是否稍高于3’端等。 一,普通引物对的搜索: 以Mouse 4E(cDNA序列)为例。我们的目的是以Mouse 4E (2361 bp)为模板,设计一对引物来扩增出600-800bp长的PCR产物。 1,点击“Search“菜单中的”For Primers and Probes“命令,进入引物搜索对话框; 2,由于我们要设计的是一对PCR引物,因此正、负链的复选框都要选上,同时选上Compatible pairs。 在Oligo默认的状态下,对此引物对的要求有:a,无二聚体;b,3’端高度特异,GC含量有限定,d,去除错误引发引物等。3,剩下的工作是确定上、下游引物的位置及PCR产物的长度以及引物设计参数。 ①单击:“search Ranges”按钮,弹出“Search Ranges”对
引物设计的原理与方法 This model paper was revised by the Standardization Office on December 10, 2020
PCR引物设计的原理及方法 阎振鑫S111666(四川大学生命科学学院细胞生物学成都 610014) 摘要:自20世纪后期发展了PCR技术以来,PCR已经改变了整个生物学研究的进程。而PCR反应的第一步就是设计引物,引物设计的好坏直接关系到PCR的成败。PCR引物设计有许多的原则必须要遵循:引物与引物之间避免形成稳定的二聚体或发夹结构,引物与模板的序列要紧密互补。引物不能在模板的非目的位点引发DNA聚合反应等。另外,引物的设计方法也越来越多,出现了许多专门的设计软件和网站,如:PrimerPremier5.0等。 关键词:PCR 引物原理方法 NCBI PrimerPremier5.0 PCR primer design principle and method YanZhenxin (sichuan Univercity, Life science college cell biology chengdu 610014 ) Abstract: When PCR technology was find, PCR has changed all of the program in research of biology. The design of primer is the frist step of PCR. It is relation to the fate of PCR. There are some principals must be obey: dipolymer and hairpin structure must be avoid between different primers. The DNA polymerization reaction should not be triggered at the wrong site. Therefore, there are more and more methods of design primer, include the professional softwares and professional web site. Key word: PCR primer principle NCBI PrimerPremier5.0 聚合酶链式反应(Polymerase chain reaction。PCR)是20世纪后期发展起来的 一种体外扩增特异DNA片断的技术。具有快速、简便及高度敏感等优点,能极大地缩短目的基因扩增时间[1]。因此,其一直是生物学者们致力于构建cDNA文库、基因克隆以及表达调控研究的必要前提和基础[2]。PCR的第一步就是引物设计。引物设计的好坏,直接影响了PCR的结果,因此这一步很关键。成功的PCR反应既要高效,又要特异性扩增产物,因此对引物设计提出了较高的要求。引物设计需要注意的地方很多,在大多数情况下,我们都是在知道已知模板序列时进行PCR扩增的。在某些情况比如构建文库的时候也会在不知道模板序列的情况下进行设计。这个时候随机核苷酸序列
mi引物设计原则 1、引物的长度一般为15-30 bp,常用的就是18-27 bp,但不应大于38,因为过长会导致其延伸温度大于74℃,不适于Taq DNA聚合酶进行反应。 2、引物序列在模板内应当没有相似性较高,尤其就是3’端相似性较高的序列,否则容易导致错配。引物3’端出现3个以上的连续碱基,如GGG或CCC,也会使错误引发机率增加。 3、引物3’端的末位碱基对Taq酶的DNA合成效率有较大的影响。不同的末位碱基在错配位置导致不同的扩增效率,末位碱基为A的错配效率明显高于其她3个碱基,因此应当避免在引物的3’端使用碱基A。另外,引物二聚体或发夹结构也可能导致PCR反应失败。5’端序列对PCR影响不太大,因此常用来引进修饰位点或标记物。 4、引物序列的GC含量一般为40-60%,过高或过低都不利于引发反应。上下游引物的GC含量不能相差太大。 5、引物所对应模板位置序列的Tm值在72℃左右可使复性条件最佳。Tm值的计算有多种方法,如按公式Tm=4(G+C)+2(A+T),在Oligo软件中使用的就是最邻近法(the nearest neighbor method)。 6、ΔG值就是指DNA双链形成所需的自由能,该值反映了双链结构内部碱基对的相对稳定性。应当选用3’端ΔG值较低(绝对值不超过9),而5’端与中间ΔG值相对较高的引物。引物的3’端的ΔG值过高,容易在错配位点形成双链结构并引发DNA聚合反应。 7、引物二聚体及发夹结构的能值过高(超过4、5kcal/mol)易导致产生引物二聚体带,并且降低引物有效浓度而使PCR反应不能正常进行。 8、对引物的修饰一般就是在5’端增加酶切位点,应根据下一步实验中要插入PCR产物的载体的相应序列而确定。 引物序列应该都就是写成5-3方向的, Tm之间的差异最好控制在1度之内, 另外我觉得扩增长度大一些比较好,500bp左右。 要设计引物首先要找到DNA序列的保守区。同时应预测将要扩增的片段单链就是否形成二级结构。如这个区域单链能形成二级结构,就要避开它。如这一段不
图解blast 验证引物教程 ——以文献报道的人类的ABCG2的引物为例 1、 进入网页:https://www.doczj.com/doc/ec2572381.html,/BLAST/ 2、 点击Basic BLAST 中的nucleotide blast 选项 3、 完成2操作后就进入了Basic Local Alignment Search Tool 界面 (1)在Enter Query Sequence 栏中输入引物序列: 注:文献报道ABCG2的引物为5’-CTGAGATCCTGAGCCTTTGG-3’; 5’-TGCCCATCACAACATCATCT-3’ 简便的做法是同时输入上下游引物。输入上下游引物系列都从5’— 3’。 输入上游引物后,加上≥20个字母n ,再输入下游引物,如下图: 生 物 秀
(2)在Choose Search Set 栏中: Database 根据预操作基因的种属定了,本引物可选Human genomic + transcript 或Others (nr etc.)。本人倾向于选后者,觉得此库信息更多。如下图: (3)在Program Selection 中:选择Somewhat similar sequences (blastn)项,如下图: (4)在此界面最下面:如下图 生物秀-专心做生物 w w w .b b i o o .c o m
Show results in a new window 项是显示界面的形式,可选可不选,在此我们选上了。关键要点击Algorithm parameters 参数设置,进入参数设置界面。 4. 参数设置: (1)在General Parameters 中:Expect thresshold 期望阈值须改为1000,大于1000也可以;在Word size 的下拉框将数字改为7。如下图: (2)Scoring Parameters 无须修改 (3)Filters and Masking 中,一般来说也没有必要改 5.点击最下面一栏的BLAST 按钮,如图: 6.点击BLAST 按钮后,跳转出现如下界面: 7. 等待若干秒之后,自动跳转出现显示BLAST 结果的网页。该网页用三种形式来显示blast 的结果。 生物秀-专心做生物 w w w .b b i o o .c o m