NEB保护碱基-各种酶切位点保护碱基
- 格式:doc
- 大小:65.50 KB
- 文档页数:4
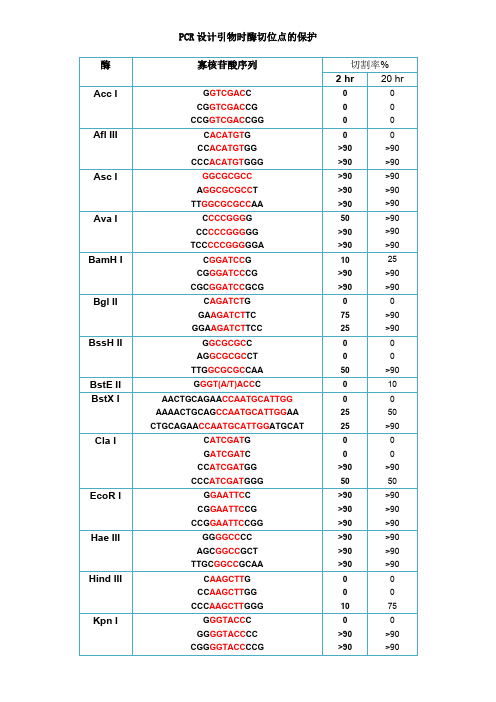
PCR设计引物时酶切位点的保护
注释:
1.如果要加在序列的5‘端,就在酶切位点识别碱基序列(红色)的5’端加上相应的碱基(黑色),相同如果要在3‘端加保护碱基,就在酶切位点识别碱基序列(红色)的3’端加上相应的碱基(黑色)。
2.切割率:正确识别并酶切的效率
3.加保护碱基时最好选用切割率高时加的相应碱基。
一般情况下,普通的内切酶只加入两个保护碱基,其内切反应就可以正常进行;而有一类,仅仅只加入两个保护碱基,其内切反应就不能正常进行,这是因为内切酶不能正常结合DNA段上。
如NdeI就属这类,需要加入至少6个保护碱基,常用的HindIII也要三个。
添加什么保护碱基,如果严格点,是根据两条引物的Tm值和各引物的碱基分布及GC含量。
如果某条引物Tm值偏小,GC%较低,添加时多加G或C,反之亦反。
当在引物5’端添加酶切位点时要考虑:
a)该目的序列内部不得含有相同的酶切位点,这样的错误会给将来的克隆造成麻烦。
b)如果打算PCR 后直接酶切,不要忘了在酶切位点的外侧再加上保护碱基,不同的酶对于保护碱基的要求是不同的。
如果不设计保护碱基,则多半要用TA 克隆的方式连接到质粒上,这时要注意Taq 酶的选择,若想在目的序列上附加上并不存在的序列,如限制位点和启动子序列,可以加入到引物5'端而不影响特异性。
当计算引物Tm 值时并不包括这些序列,但是应该对其进行互补性和内部二级结构的检测。

PCR设计引物时酶切位点的保护碱基引物设计是PCR实验的关键步骤之一,引物的好坏会直接影响到PCR反应的成功与否。
而在引物设计过程中,酶切位点的保护碱基是需要考虑的重要因素之一在PCR实验中,引物的作用是指定PCR反应的放大区域,并提供启动位点供聚合酶结合。
一般情况下,引物至少需要包含一段特定的DNA序列,以便与目标序列互补配对。
在引物设计过程中,选择合适的酶切位点是十分必要的。
酶切位点是指位于特定DNA序列上的限制酶可以识别并切割的区域。
酶切位点的选择通常需要考虑如下几个方面:1.切割效果:选择切割效果好的酶切位点可以提高PCR反应的特异性和灵敏度。
经典的选择是选择一种具有4-6个碱基的酶切位点,并且该位点在引物中间的位置。
这可以有效防止酶切位点的保护碱基对PCR反应的影响。
2.特异性:引物需要选择适合的酶切位点,以确保只有目标序列被放大,而不包括其他与之相关的非特异性序列。
因此,在选择酶切位点时应尽量避免与其他非特异性序列存在相似性。
3.引物长度:引物长度的选择也与酶切位点相关。
如果引物长度过短,可能会导致酶切位点过于靠近PCR反应产物的端点,从而使切割效果不佳。
因此,在引物设计时,应选择适当的引物长度,以保证酶切位点的保护碱基不会对PCR反应产物的生成产生不利影响。
酶切位点的保护碱基是指在特定的DNA序列上,通过选择相应的碱基来避免受到酶切的影响。
常见的保护碱基有甲基化碱基、磷酸化碱基以及接上阻断扩增的非互补碱基等。
1.甲基化碱基:将酶切位点中的一些碱基进行甲基化处理,可以有效地阻止特定酶的切割作用。
甲基化碱基可以通过DNA甲基转移酶进行甲基化修饰。
2.磷酸化碱基:磷酸化碱基是在引物设计过程中添加磷酸基团的方法,通过给酶切位点添加一个磷酸基团来阻断酶的切割作用。
3.非互补碱基:为了阻断酶切位点的切割作用,可以在酶切位点的周围引入一个与其不互补的碱基序列。
这样可以阻断酶的结合和切割。
总的来说,选择合适的酶切位点和保护碱基对PCR实验的成功至关重要。

克隆PCR产物的方法之一,是在PCR产物两端设计一定的限制酶切位点,经酶切后克隆至用相同酶切的载体中。
但实验证明,大多数限制酶对裸露的酶切位点不能切断。
必须在酶切位点旁边加上一个至几个保护碱基,才能使所定的限制酶对其识别位点进行有效切断。
有研究者使用了15种限制酶,分别比较了各种限制酶在其酶切位点旁边分别加0、1、2、3个保护碱基后的切断情况。
结果显示,基本上所有限制酶,在其酶切位点旁边加上3个以上的保护碱基后,可以对其酶切位点进行有效切断。
一般来讲,在酶切位点前加入两个GC碱基,因为如果保护碱基为A T的话,保护碱基在PCR 产物的末端,A T之间只有两个氢键,结合力差,容易在末端产生单链,这样的话限制性内切酶就无法作用。
其实加保护碱基的多少,是具体情况具体讨论,比如HindIII、BamHI等就得有三个保护碱基,少了一个就无法切动。
注释:
1.如果要加在序列的5‘端,就在酶切位点识别碱基序列(红色)的5’端加上相应的碱基(黑色),相同如果要在3‘端加保护碱基,就在酶切位点识别碱基序列(红色)的3’端加上相应的碱基(黑色)。
2.切割率:正确识别并酶切的效率
3. 加保护碱基时最好选用切割率高时加的相应碱基。
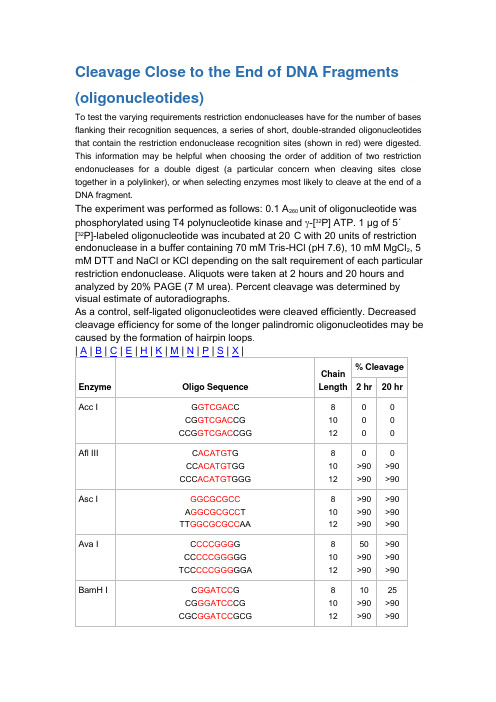
Cleavage Close to the End of DNA Fragments (oligonucleotides)To test the varying requirements restriction endonucleases have for the number of bases flanking their recognition sequences, a series of short, double-stranded oligonucleotides that contain the restriction endonuclease recognition sites (shown in red) were digested. This information may be helpful when choosing the order of addition of two restriction endonucleases for a double digest (a particular concern when cleaving sites close together in a polylinker), or when selecting enzymes most likely to cleave at the end of a DNA fragment.The experiment was performed as follows: 0.1 A260 unit of oligonucleotide was phosphorylated using T4 polynucleotide kinase and -[32P] ATP. 1 µg of 5´[32P]-labeled oligonucleotide was incubated at 20°C with 20 units of restriction endonuclease in a buffer containing 70 mM Tris-HCl (pH 7.6), 10 mM MgCl2, 5 mM DTT and NaCl or KCl depending on the salt requirement of each particular restriction endonuclease. Aliquots were taken at 2 hours and 20 hours and analyzed by 20% PAGE (7 M urea). Percent cleavage was determined by visual estimate of autoradiographs.As a control, self-ligated oligonucleotides were cleaved efficiently. Decreased cleavage efficiency for some of the longer palindromic oligonucleotides may be caused by the formation of hairpin loops.Cleavage Close to the End of DNA Fragments (linearized vector)Linearized vectors were incubated with the indicated enzymes (10 units/µg) for 60 minutes at the recommended incubation temperature and NEBuffer for each enzyme. Following ligation and transformation, cleavage efficiencies were determined by dividing the number of transformants from the digestion reaction by the number obtained from religation of the linearized DNA (typically 100-500 colonies) and subtracting from 100%. "Base Pairs from End" refers to the number of double-stranded base pairs between the recognition site and the terminus of the fragment; this number does not include the single- stranded overhang from the initial cut. Since it has not been demonstrated whether these single-stranded nucleotides contribute to cleavage efficiency, 4 bases should be added to the indicated numbers when designing PCR primers. Average efficiencies were rounded to the nearest whole number; experimental variation was typically within 10%. The numbers in parentheses refer to the number of independent trials for each enzyme tested (from Moreira, R. and Noren, C. (1995), Biotechniques, 19, 56-59).Note: As a general rule, enzymes not listed below require 6 bases pairs on either side of their recognition site to cleave efficiently.| A | B | E | H | K | M | N | P | S | X |。

酶寡核苷酸序列切割率%2 hr20 hrNot I TT GCGGCCGC AAATTT GCGGCCGC TTTAAAATAT GCGGCCGC TATAAAATAAGAAT GCGGCCGC TAAACTATAAGGAAAAAA GCGGCCGC AAAAGGAAAA10102525101090>90Nsi I TGC ATGCAT GCACCA ATGCAT TGGTTCTGCAGTT10>90>90>90Pac I TTAATTAAG TTAATTAA CCC TTAATTAA GG 025>90Pme I GTTTAAACG GTTTAAAC CGG GTTTAAAC CCAGCTTT GTTTAAAC GGCGCGCCGG752550>90Pst I G CTGCAG CTGCA CTGCAG TGCAAA CTGCAG AACCAATGCATTGGAAAA CTGCAG CCAATGCATTGGAACTGCAG AACCAATGCATTGGATGCAT10>90>9010>90>90Pvu I C CGATCG GAT CGATCG ATTCG CGATCG CGA102510Sac I C GAGCTC G1010Sac II G CCGCGG CTCC CCGCGG GGA50>90Sal I GTCGAC GTCAAAAGGCCATAGCGGCCGC GC GTCGAC GTCTTGGCCATAGCGGCCGCGGACGC GTCGAC GTCGGCCATAGCGGCCGCGGAA10105075Sca I G AGTACT CAAA AGTACT TTT 10752575C CCCGGG G CC CCCGGG GG TCC CCCGGG GGA10>901050>90Spe I G ACTAGT CGG ACTAGT CCCGG ACTAGT CCGCTAG ACTAGT CTAG 1010>90>905050Sph I G GCATGC CCAT GCATGC ATGACAT GCATGC ATGT102550Stu I A AGGCCT TGA AGGCCT TCAAA AGGCCT TTT >90>90>90>90>90>90Xba I C TCTAGA GGC TCTAGA GCTGC TCTAGA GCACTAG TCTAGA CTAG>907575>90>90>90Xho I C CTCGAG GCC CTCGAG GGCCG CTCGAG CGG10102575Xma I C CCCGGG GCC CCCGGG GGCCC CCCGGG GGGTCCC CCCGGG GGGA2550>9075>90>90酶寡核苷酸序列切割率%2 hr20 hrAcc I G GTCGAC CCG GTCGAC CGCCG GTCGAC CGG 0Afl III C ACATGT GCC ACATGT GGCCC ACATGT GGG>90>90>90>90Asc I GGCGCGCCA GGCGCGCC TTT GGCGCGCC AA >90>90>90>90>90>90CC CCCGGG GG TCC CCCGGG GGA >90>90>90>90BamH I C GGATCC GCG GGATCC CGCGC GGATCC GCG10>90>9025>90>90Bgl II C AGATCT GGA AGATCT TCGGA AGATCT TCC7525>90>90BssH II G GCGCGC CAG GCGCGC CTTTG GCGCGC CAA50>90BstE II G GGT(A/T)ACC C010BstX I AACTGCAGAA CCAATGCATTGGAAAACTGCAG CCAATGCATTGG AACTGCAGAA CCAATGCATTGG ATGCAT252550>90Cla I C ATCGAT GG ATCGAT CCC ATCGAT GGCCC ATCGAT GGG>9050>9050EcoR I G GAATTC CCG GAATTC CGCCG GAATTC CGG >90>90>90>90>90>90Hae III GG GGCC CCAGC GGCC GCTTTGC GGCC GCAA >90>90>90>90>90>90Hind III C AAGCTT GCC AAGCTT GGCCC AAGCTT GGG1075Kpn I G GGTACC CGG GGTACC CCCGG GGTACC CCG>90>90>90>90Mlu I G ACGCGT CCG ACGCGT CG2550Nco I C CCATGG GCATG CCATGG CATG5075Nde I C CATATG GCC CATATG GGCGC CATATG GCGGGGTTT CATATG AAACCCGGAATTC CATATG GAATTCCGGGAATTC CATATG GAATTCCC7575>90>90Nhe I G GCTAGC CCG GCTAGC CGCTA GCTAGC TAG10102550。

常用酶切位点序列和保护碱基引言在分子生物学和遗传工程领域,酶切位点序列和保护碱基是非常重要的概念。
酶切位点序列指的是DNA或RNA上特定的核苷酸序列,这些序列可以被特定的酶识别并切割。
保护碱基则是指在实验过程中采取措施来保护DNA或RNA上特定的核苷酸,使其不被酶切割。
本文将对常用的酶切位点序列和保护碱基进行详细介绍,包括其定义、常见的酶切位点序列、如何选择合适的保护碱基等内容。
酶切位点序列定义酶切位点序列是指DNA或RNA分子上具有一定规律性、可以被特定的限制性内切酶识别并结合从而发挥催化作用的核苷酸序列。
这些限制性内切酶通常能够识别4-8个核苷酸,并在识别到相应的位点后将DNA或RNA分子切割成片段。
常见的酶切位点序列1.EcoRI: 5’-GAATTC-3’,3’-CTTAAG-5’2.HindIII: 5’-AAGCTT-3’,3’-TTCGAA-5’3.BamHI: 5’-GGATCC-3’,3’-CCTAGG-5’4.XhoI: 5’-CTCGAG-3’,3’-GAGCTC-5’5.NotI: 5’-GCGGCCGC-3’,3’-CGCCGGCG-5’这些酶切位点序列是常用的限制性内切酶的识别序列,它们在分子生物学实验中被广泛应用。
通过将DNA或RNA与特定的限制性内切酶一起反应,可以实现DNA或RNA的特定部位切割。
保护碱基定义保护碱基是指在实验过程中采取措施来保护DNA或RNA上特定的核苷酸,使其不被酶切割。
这种保护通常通过对特定的碱基进行修饰或使用化学试剂来实现。
如何选择合适的保护碱基选择合适的保护碱基需要考虑以下几个因素: 1. 酶切位点序列:首先要了解所使用的限制性内切酶的酶切位点序列,以确定需要保护的碱基。
2. 保护方法:根据实验需求和实验条件选择合适的保护方法。
常见的保护方法包括使用化学修饰剂修饰碱基、使用特殊的核苷酸引物或引入特定的修饰基团等。
3. 保护效果:选择的保护碱基应能够有效地阻止限制性内切酶与目标位点结合并发挥催化作用。
酶切保护碱基设计---fromNEBCleavage Close to the End of DNA Fragments (linearized vector)Linearized vectors were incubated with the indicated enzymes (10 units/μg) for 60 minutes at the recommended incubation temperature and NEBuffer for each enzyme. Following ligation and transformation, cleavage efficiencies were determined by dividing the number of transformants from the digestion reaction by the number obtained from religation of the linearized DNA (typically 100-500 colonies) and subtracting from 100%. "Base Pairs from End" refers to the number of double-stranded base pairs between the recognition site and the terminus of the fragment; this number does not include the single-stranded overhang from the initial cut. Since it has not been demonstrated whether these single-stranded nucleotides contribute to cleavage efficiency, 4 bases should be added to the indicated numbers when designing PCR primers. Average efficiencies were rounded to the nearest whole number; experimental variation was typically within 10%. The numbers in parentheses refer to the number of independent trials for each enzyme tested (from Moreira, R. and Noren, C. (1995), Biotechniques, 19, 56-59).Note: As a general rule, enzymes not listed below require 6 bases pairs on either side of their recognition site to cleave efficiently.| A | B | E | H | K | M | N | P | S | X |Enzyme Base pairsfrom End%CleavageEfficiency Vector Initial CutAat II32188 (2)100 (2)95 (2)LITMUS 29LITMUS 28LITMUS 29Nco INco IPinA IAcc65 I21 99 (2)75 (3)LITMUS 29pNEB193Spe ISac IAfl II113 (2)LITMUS 29Stu IAge I11 100 (1)100 (2)LITMUS 29LITMUS 29Xba IAat IIApa I2100 (1)LITMUS 38Spe I Asc I197 (2)pNEB193BamH I Avr II1100 (2)LITMUS 29Sac I BamH I197 (2)LITMUS 29Hind III BglII3100 (2)LITMUS 29Nsi I BsiW I2100 (2)LITMUS 29BssH II BspE I2100 (1)LITMUS 39BsrG IBsrG I21 99 (2)88 (2)LITMUS 39LITMUS 38Sph IBspE IBssH II2100 (2)LITMUS 29BsiW I Eag I2100 (2)LITMUS 39Nhe IEcoR I111 100 (1)88 (1)100 (1)LITMUS 29LITMUS 29LITMUS 39Xho IPst INhe IEcoR V1100 (2)LITMUS 29Pst IHind III321 90 (2)91 (2)0 (2)LITMUS 29LITMUS 28LITMUS 29Nco INco IBamH IKas I21 97 (1)93 (1)LITMUS 38LITMUS 38NgoM IVHind IIIKpn I221 100 (2)100 (2)99 (2)LITMUS 29LITMUS 29pNEB193Spe ISac ISac IMlu I299 (2)LITMUS 39Eag I Mun I2100 (1)LITMUS 39NgoM IV Nco I2100 (1)LITMUS 28Hind III NgoM IV2100 (1)LITMUS 39Mun INhe I12 100 (1)82 (1)LITMUS 39LITMUS 39Eag INot I741 100 (2)100 (1)98 (2)Bluescript SK-Bluescript SK-Bluescript SK-Spe IKsp IXba INsi I332 100 (2)77 (4)95 (2)LITMUS 29LITMUS 29LITMUS 28BssH IIBgl IIBssH IIPac I176 (3)pNEB193BamH I Pme I194 (2)pNEB193Pst I Pst I3298 (1)50 (5)LITMUS 29LITMUS 39Hind IIISac I199 (2)LITMUS 29Avr II Sal I321 89 (2)23 (2)61 (3)LITMUS 39LITMUS 39LITMUS 38Spe ISph ISph ISpe I22 100 (2)100 (2)LITMUS 29LITMUS 29Acc65 IKpn ISph I221 99 (1)97 (1)92 (2)LITMUS 39LITMUS 39LITMUS 38Sal ISal IXba I11 99 (2)94 (1)LITMUS 29LITMUS 29Age IPinA IXho I197 (2)LITMUS 29EcoR IXma I22 98 (1)92 (1)pNEB193pNEB193Asc IBssH IINew England Biolabs Technical Literature - Updated 03/05/2004Cleavage Close to the End of DNA Fragments (oligonucleotides)To test the varying requirements restriction endonucleases have for the number of bases flanking their recognition sequences, a series of short, double-stranded oligonucleotides that contain the restriction endonuclease recognition sites (shown in red) were digested. This information may be helpful when choosing the order of addition of two restriction endonucleases for a double digest (a particular concern when cleaving sites close together in a polylinker), or when selecting enzymes most likely to cleave at the end of a DNA fragment.The experiment was performed as follows: 0.1 A260 unit of oligonucleotide was phosphorylated using T4 polynucleotide kinas e and γ-[32P] ATP. 1 μg of 5′ [32P]-labeled oligonucleotide was incubated at 20°C with 20 units of restriction endonuclease in a buffer containing 70 mM Tris-HCl (pH 7.6), 10 mM MgCl2, 5 mM DTT and NaCl or KCl depending on the salt requirement of each particular restriction endonuclease. Aliquots were taken at 2 hours and 20 hours and analyzed by 20% PAGE (7 M urea). Percent cleavage was determined by visual estimate of autoradiographs.As a control, self-ligated oligonucleotides were cleaved efficiently. Decreased cleavage efficiency for some of the longer palindromic oligonucleotides may be caused by the formation of hairpin loops.| A | B | C | E | H | K | M | N | P | S | X |% Cleavage Enzyme Oligo SequenceChain Length2 hr 20 hr Acc IG GTCGAC C CG GTCGAC CG CCG GTCGAC CGG8 10 12 0 0 0 0 0 0 Afl IIIC ACATGT G CC ACATGT GG CCC ACATGT GGG8 10 12 0 >90 >900 >90>90Asc IGGCGCGCC A GGCGCGCC T TT GGCGCGCC AA8 10 12 >90 >90 >90 >90 >90 >90 Ava IC CCCGGG G CC CCCGGG GG TCC CCCGGG GGA8 10 12 50 >90 >90 >90 >90>90 BamH IC GGATCC G CG GGATCC CG CGC GGATCC GCG8 10 12 10 >90 >90 25 >90>90 Bgl IIC AGATCT G GA AGATCT TC GGA AGATCT TCC8 10 12 0 75 25 0 >90 >90 BssH IIG GCGCGC C AG GCGCGC CT TTG GCGCGC CAA8 10 12 0 0 50 0 0 >90 BstE II G GGT(A/T)ACC C 9 0 10 BstX IAACTGCAGAA CCAATGCATTGG AAAACTGCAG CCAATGCATTGG AA CTGCAGAA CCAATGCATTGG ATGCAT22 24 27 0 25 25 0 50 >90 Cla IC ATCGAT G G ATCGAT C CC ATCGAT GG CCC ATCGAT GGG8 8 10 12 0 0 >90 500 0 >9050EcoR IG GAATTC C CG GAATTC CG CCG GAATTC CGG8 10 12 >90 >90 >90>90 >90 >90Hae IIIGG GGCC CC AGC GGCC GCT8 10>90 >90 >90 >90TTGC GGCC GCAA12>90>90Hind III C AAGCTT GCC AAGCTT GGCCC AAGCTT GGG810121075Kpn I G GGTACC CGG GGTACC CCCGG GGTACC CCG81012>90>90>90>90Mlu I G ACGCGT CCG ACGCGT CG8102550Nco I C CCATGG GCATG CCATGG CATG8145075Nde I C CATATG GCC CATATG GGCGC CATATG GCGGGGTTT CATATG AAACCC GGAATTC CATATG GAATTCC GGGAATTC CATATG GAATTCCC 8121820227575>90>90Nhe I G GCTAGC CCG GCTAGC CGCTA GCTAGC TAG8101210102550Not I TT GCGGCCGC AAATTT GCGGCCGC TTTAAAATAT GCGGCCGC TATAAAATAAGAAT GCGGCCGC TAAACTAT AAGGAAAAAA GCGGCCGC AAAAGGAAAA 12 16202428101025101090>90Nsi I TGC ATGCAT GCACCA ATGCAT TGGTTCTGCAGTT 12 2210>90>90>90Pac I TTAATTAAG TTAATTAA CCC TTAATTAA GG8101225>90Pme I GTTTAAACG GTTTAAAC CGG GTTTAAAC CCAGCTTT GTTTAAAC GGCGCGCCGG 81012247550>90Pst I G CTGCAG CTGCA CTGCAG TGCAAA CTGCAG AACCAATGCATTGG AAAA CTGCAG CCAATGCATTGGAA CTGCAG AACCAATGCATTGGATGCAT 81422242610>90>9010>90>90Pvu I C CGATCG GAT CGATCG ATTCG CGATCG CGA81012102510Sac I C GAGCTC G81010Sac II G CCGCGG CTCC CCGCGG GGA81250>90Sal I GTCGAC GTCAAAAGGCCATAGCGGCCGC GC GTCGAC GTCTTGGCCATAGCGGCCGCGG ACGC GTCGAC GTCGGCCATAGCGGCCGCGGAA 28 303210105075Sca I G AGTACT CAAA AGTACT TTT81210752575Sma I CCCGGGC CCCGGG GCC CCCGGG GGTCC CCCGGG GGA68101210>90101050>90Spe I G ACTAGT C GG ACTAGT CC CGG ACTAGT CCG CTAG ACTAGT CTAG 81012141010>90>905050Sph I G GCATGC C CAT GCATGC ATG ACAT GCATGC ATGT 81214102550Stu I A AGGCCT TGA AGGCCT TC AAA AGGCCT TTT 81012>90>90>90>90>90>90Xba I C TCTAGA G GC TCTAGA GC TGC TCTAGA GCA CTAG TCTAGA CTAG 8101214>907575>90>90>90Xho I C CTCGAG G CC CTCGAG GG CCG CTCGAG CGG 8101210102575Xma I C CCCGGG G CC CCCGGG GG CCC CCCGGG GGG TCCC CCCGGG GGGA 81012142550>9075>90>90。
酶切位点保护碱基-PCR引物设计用于限制性内切酶酶切反应本文给出了分子克隆中常用限制性内切酶的保护碱基序列,如AccI,AflIII,AscI,AvaI,BamHI,BglII,BssHII,BstEII,BstXI,ClaI,EcoRI,HaeIII,HindIII,K pnI,MluI,NcoI,NdeI,NheI,NotI,NsiI,PacI,PmeI,PstI,PvuI,SacI,S acII,SalI,ScaI,SmaI,SpeI,SphI,StuI,XbaI,XhoI,XmaI,为什么要添加保护碱基?在分子克隆实验中,有时我们会在待扩增的目的基因片段两端加上特定的酶切位点,用于后续的酶切和连接反应。
由于直接暴露在末端的酶切位点不容易直接被限制性核酸内切酶切开,因此在设计PCR引物时,人为的在酶切位点序列的5‘端外侧添加额外的碱基序列,即保护碱基,用来提高将来酶切时的活性。
其次,在分子克隆实验中选择载体的酶切位点时,相临的两个酶切位点往往不能同时使用,因为一个位点切割后留下的碱基过少以至于影响旁边的酶切位点切割。
该如何添加保护碱基?添加保护碱基时,最关心的应该是保护碱基的数目,而不是种类。
什么样的酶切位点,添加几个保护碱基,是有数据可以参考的。
添加什么保护碱基,如果严格点,是根据两条引物的Tm值和各引物的碱基分布及GC含量。
如果某条引物Tm值偏小,GC%较低,添加时多加G或C,反之亦反。
为了解不同内切酶对识别位点以外最少保护碱基数目的要求,NEB采用了一系列含识别序列的短双链寡核苷酸作为酶切底物进行实验。
实验结果对于确定双酶切顺序将会有帮助(比如在多接头上切割位点很接近时),或者当切割位点靠近DNA末端时也很有用。
在本表中没有列出的酶,则通常需在识别位点两端至少加上6个保护碱基,以确保酶切反应的进行。
实验方法:用γ-[32P]ATP在T4多聚核苷酸激酶的作用下标记0.1A260单位的寡核苷酸。
酶切位点保护碱基-PCR引物设计用于限制性内切酶发布: 2010-05-24 20:19| 来源:生物吧| 编辑:刘浩| 查看: 161 次本文给出了分子克隆中常用限制性内切酶的保护碱基序列,如AccI,AflIII,AscI,AvaI,BamHI,BglII,BssHII,BstEII,BstXI,ClaI,EcoRI,HaeIII,HindIII,KpnI,MluI,NcoI,NdeI,NheI,NotI,NsiI,PacI,PmeI,PstI,PvuI,SacI,SacII,SalI,ScaI,SmaI,SpeI,SphI,StuI,XbaI,XhoI,XmaI,为什么要添加保护碱基?在分子克隆实验中,有时我们会在待扩增的目的基因片段两端加上特定的酶切位点,用于后续的酶切和连接反应。
由于直接暴露在末端的酶切位点不容易直接被限制性核酸内切酶切开,因此在设计PCR引物时,人为的在酶切位点序列的5‘端外侧添加额外的碱基序列,即保护碱基,用来提高将来酶切时的活性。
其次,在分子克隆实验中选择载体的酶切位点时,相临的两个酶切位点往往不能同时使用,因为一个位点切割后留下的碱基过少以至于影响旁边的酶切位点切割。
该如何添加保护碱基?添加保护碱基时,最关心的应该是保护碱基的数目,而不是种类。
什么样的酶切位点,添加几个保护碱基,是有数据可以参考的。
添加什么保护碱基,如果严格点,是根据两条引物的Tm值和各引物的碱基分布及GC含量。
如果某条引物Tm值偏小,GC%较低,添加时多加G或C,反之亦反。
为了解不同内切酶对识别位点以外最少保护碱基数目的要求,NEB采用了一系列含识别序列的短双链寡核苷酸作为酶切底物进行实验。
实验结果对于确定双酶切顺序将会有帮助(比如在多接头上切割位点很接近时),或者当切割位点靠近DNA末端时也很有用。
在本表中没有列出的酶,则通常需在识别位点两端至少加上6个保护碱基,以确保酶切反应的进行。
实验方法:用γ-[32P]ATP在T4多聚核苷酸激酶的作用下标记0.1A26单位的寡核苷酸。
PCR设计引物时酶切位点的保护
酶 寡核苷酸序列
切割率%
2 hr 20 hr
Acc I
GGTCGACC CGGTCGACCG CCGGTCGACCGG 0 0 0 0
0
0
Afl III
CACATGTG CCACATGTGG CCCACATGTGGG 0 >90 >90 0
>90
>90
Asc I
GGCGCGCC AGGCGCGCCT TTGGCGCGCCAA >90 >90 >90 >90
>90
>90
Ava I
CCCCGGGG CCCCCGGGGG TCCCCCGGGGGA 50 >90 >90 >90
>90
>90
BamH I
CGGATCCG CGGGATCCCG CGCGGATCCGCG 10 >90 >90 25
>90
>90
Bgl II
CAGATCTG GAAGATCTTC GGAAGATCTTCC 0 75 25 0
>90
>90
BssH II
GGCGCGCC AGGCGCGCCT TTGGCGCGCCAA 0 0 50 0
0
>90
BstE II
GGGT(A/T)ACCC 0 10
BstX I
AACTGCAGAACCAATGCATTGG AAAACTGCAGCCAATGCATTGGAA CTGCAGAACCAATGCATTGGATGCAT 0 25 25 0
50
>90
Cla I
CATCGATG GATCGATC CCATCGATGG CCCATCGATGGG 0 0 >90 50 0
0
>90
50
EcoR I
GGAATTCC CGGAATTCCG CCGGAATTCCGG >90 >90 >90 >90
>90
>90
Hae III
GGGGCCCC AGCGGCCGCT TTGCGGCCGCAA >90 >90 >90 >90
>90
>90
Hind III
CAAGCTTG CCAAGCTTGG CCCAAGCTTGGG 0 0 10 0
0
75
Kpn I
GGGTACCC GGGGTACCCC CGGGGTACCCCG 0 >90 >90 0
>90
>90
Mlu I
GACGCGTC CGACGCGTCG 0 25 0
50
Nco I
CCCATGGG CATGCCATGGCATG 0 50 0
75
Nde I
CCATATGG CCCATATGGG CGCCATATGGCG GGGTTTCATATGAAACCC GGAATTCCATATGGAATTCC GGGAATTCCATATGGAATTCCC 0 0 0 0 75 75 0
0
0
0
>90
>90
Nhe I
GGCTAGCC CGGCTAGCCG CTAGCTAGCTAG 0 10 10 0
25
50
Not I
TTGCGGCCGCAA ATTTGCGGCCGCTTTA AAATATGCGGCCGCTATAAA ATAAGAATGCGGCCGCTAAACTAT AAGGAAAAAAGCGGCCGCAAAAGGAAAA 0 10 10 25 25 0
10
10
90
>90
Nsi I
TGCATGCATGCA CCAATGCATTGGTTCTGCAGTT 10 >90 >90
>90
Pac I
TTAATTAA GTTAATTAAC CCTTAATTAAGG 0 0 0 0
25
>90
Pme I
GTTTAAAC GGTTTAAACC GGGTTTAAACCC AGCTTTGTTTAAACGGCGCGCCGG 0 0 0 75 0
25
50
>90
Pst I
GCTGCAGC TGCACTGCAGTGCA AACTGCAGAACCAATGCATTGG AAAACTGCAGCCAATGCATTGGAA CTGCAGAACCAATGCATTGGATGCAT 0 10 >90 >90 0 0
10
>90
>90
0
Pvu I
CCGATCGG ATCGATCGAT TCGCGATCGCGA 0 10 0 0
25
10
Sac I
CGAGCTCG 10 10
Sac II
GCCGCGGC TCCCCGCGGGGA 0 50 0
>90
Sal I
GTCGACGTCAAAAGGCCATAGCGGCCGC GCGTCGACGTCTTGGCCATAGCGGCCGCGG ACGCGTCGACGTCGGCCATAGCGGCCGCGGAA 0 10 10 0
50
75
Sca I
GAGTACTC AAAAGTACTTTT 10 75 25
75
Sma I
CCCGGG CCCCGGGG CCCCCGGGGG TCCCCCGGGGGA 0 0 10 >90 10
10
50
>90
Spe I
GACTAGTC GGACTAGTCC CGGACTAGTCCG CTAGACTAGTCTAG 10 10 0 0 >90
>90
50
50
Sph I
GGCATGCC CATGCATGCATG ACATGCATGCATGT 0 0 10 0
25
50
Stu I
AAGGCCTT GAAGGCCTTC AAAAGGCCTTTT >90 >90 >90 >90
>90
>90
Xba I
CTCTAGAG GCTCTAGAGC TGCTCTAGAGCA CTAGTCTAGACTAG 0 >90 75 75 0
>90
>90
>90
Xho I
CCTCGAGG CCCTCGAGGG CCGCTCGAGCGG 0 10 10 0
25
75
Xma I
CCCCGGGG CCCCCGGGGG CCCCCCGGGGGG TCCCCCCGGGGGGA 0 25 50 >90 0
75
>90
>90
注释:
1.如果要加在序列的5’端,就在酶切位点识别碱基序列(红色)的5’端加
上相应的碱基(黑色),相同如果要在3’端加保护碱基,就在酶切位点识
别碱基序列(红色)的3’端加上相应的碱基(黑色)。
2.切割率:正确识别并酶切的效率
3。加保护碱基时最好选用切割率高时加的相应碱基。