动态规划法——双序列比对
- 格式:ppt
- 大小:1.56 MB
- 文档页数:55


动态规划算法及其在序列比对中应用分析序列比对是生物信息学中一个重要的问题,用于比较两个或多个生物序列的相似性和差异性。
在序列比对过程中,动态规划算法是一种常用和有效的方法。
本文将介绍动态规划算法的基本原理和应用,并深入分析其在序列比对中的应用。
1. 动态规划算法基本原理动态规划算法是一种通过把问题分解为相互重叠的子问题,并通过将每个子问题的解存储起来来解决复杂问题的方法。
它通常用于处理具有重叠子问题和最优子结构特性的问题。
动态规划算法的核心思想是将原问题拆解成若干个子问题,通过计算每个子问题的最优解来得到原问题的最优解。
这个过程可以通过建立一个状态转移方程来实现,即找到子问题之间的关联关系。
2. 动态规划在序列比对中的应用序列比对是生物信息学研究中常见的任务之一,用于比较两个或多个生物序列的相似性和差异性。
动态规划算法在序列比对中被广泛应用,最为著名的例子是Smith-Waterman算法和Needleman-Wunsch算法。
2.1 Smith-Waterman算法Smith-Waterman算法是一种用于局部序列比对的动态规划算法。
它通过为每个可能的比对位置定义一个得分矩阵,并计算出从每个比对位置开始的最优比对路径来找到最优的局部比对。
Smith-Waterman算法的基本思路是从比对矩阵的右下角开始,根据得分矩阵中每个位置的得分值和其周围位置的得分值进行计算,并记录下最大得分值及其对应的路径。
最终,通过回溯从最大得分值开始的路径,得到最优的局部比对结果。
2.2 Needleman-Wunsch算法Needleman-Wunsch算法是一种用于全局序列比对的动态规划算法。
它通过为每个比对位置定义一个得分矩阵,并通过计算出从第一个比对位置到最后一个比对位置的最优比对路径来找到最优的全局比对。
Needleman-Wunsch算法的基本思路与Smith-Waterman算法类似,但不同之处在于需要考虑序列的开头和结尾对比对结果的影响。

利用动态规划算法进行两条序列比对实 验 目 的学会使用EMBOSS 软件包的NEEDLE 和WATER 进行两条序列比对。
实 验 内 容1. Get the mRNA and protein sequence of Chlorocebus sabaeus and Colobus angolensispalliates from NCBI.2. Use Needle to do the sequence alignment in default parameters.*****Protein Sequence alignment result*****Program: needle# Rundate: Sat 28 Sep 2019 16:43:43 # Commandline: needle # -auto # -stdout# -datafile EBLOSUM62# -gapopen 10.0 # -gapextend 0.5 # -endopen 10.0 # -endextend 0.5# -aformat3 pair # -sprotein1 # -sprotein2 # Align_format: pair # Report_file: stdout # Aligned_sequences: 2# 1: XP_008000870.1 # 2: XP_011811726.1 # Matrix: EBLOSUM62 # Gap_penalty: 10.0 # Extend_penalty: 0.5 # Length: 586# Identity: 561/586 (95.7%) # Similarity: 572/586 (97.6%) # Gaps: 4/586 ( 0.7%) # Score: 2906.0***** mRNA Sequence alignment result *****# Program: needle# Rundate: Sat 28 Sep 2019 16:47:19 # Commandline: needle # -auto # -stdout# -datafile EDNAFULL# -gapopen 10.0 # -gapextend 0.5 # -endopen 10.0 # -endextend 0.5 # -aformat3 pair # -snucleotide1# -snucleotide2# Align_format: pair # Report_file: stdout # Aligned_sequences: 2# 1: XM_008002679.1 # 2: XM_011956336.1 # Matrix: EDNAFULL# Gap_penalty: 10.0 # Extend_penalty: 0.5 # Length: 2594# Identity: 2505/2594 (96.6%) # Similarity: 2505/2594 (96.6%) # Gaps: 27/2594 ( 1.0%) # Score: 12207.5a. After alignment, we have found that the identity and the gaps between two mRNAs were higher than protein, but the similarity of mRNA was lower than protein.b. The two sequences are closely related.3. Use Water to do the sequence alignment in default parameters.*****Protein Sequence alignment result*****# Program: water# Rundate: Sat 28 Sep 2019 16:52:02 # Commandline: water # -auto # -stdout# -datafile EBLOSUM62# -gapopen 10.0 # -gapextend 0.5 # -aformat3 pair# -sprotein1 # -sprotein2 # Align_format: pair # Report_file: stdout# Aligned_sequences: 2 # Matrix: EBLOSUM62# Gap_penalty: 10.0 # Extend_penalty: 0.5 ## Length: 582# Identity: 561/582 (96.4%) # Similarity: 572/582 (98.3%) # Gaps: 0/582 ( 0.0%) # Score: 2906.0*****mRNA Sequence alignment result*****# Program: water# Rundate: Sat 28 Sep 2019 16:53:55 # Commandline: water # -auto # -stdout# -datafile EDNAFULL# -gapopen 10.0 # -gapextend 0.5 # -aformat3 pair# -snucleotide1 # -snucleotide2 # Align_format: pair # Report_file: stdout## Aligned_sequences: 2 # Matrix: EDNAFULL# Gap_penalty: 10.0 # Extend_penalty: 0.5 ## Length: 2592# Identity: 2505/2592 (96.6%) # Similarity: 2505/2592 (96.6%) # Gaps: 25/2592 ( 1.0%) # Score: 12207.5a. After alignment, we have found that the identity and the gaps between two mRNAs were higher than protein, but the similarity of mRNA was lower than protein. ( THE SAME TO Needle )b. The two sequences are closely related.c. Higher identity and similarity than Needle.4. Do the protein sequence alignment and change the original parameters.Eg: We used the needle to do the protein sequence alignment and changed the GAP_OPEN from 10 to 1.# Program: needle# Rundate: Sat 28 Sep 2019 16:58:37 # Commandline: needle # -auto # -stdout# -datafile EBLOSUM62# -gapopen 1.0 # -gapextend 0.5 # -endopen 10.0 # -endextend 0.5# -aformat3 pair# -sprotein1 # -sprotein2 # Align_format: pair # Report_file: stdout ## Aligned_sequences: 2 # Matrix: EBLOSUM62# Gap_penalty: 1.0 # Extend_penalty: 0.5 ## Length: 588 # Identity: 561/588 (95.4%) # Similarity: 572/588 (97.3%) # Gaps: 8/588 ( 1.4%)# Score: 2908.0a. We can get a consequence that the identity, gaps and similarity between the protein sequences become lower, but the score become higher. ( Were more distinct but got higher score )b. Moreover, we have changed other 4 parameters, but not got any distinct result.5. Global and local pairwise sequence alignment between human myoglobin and hemoglobin protein sequences.***Global pairwise sequence alignment***Protein Sequence Alignment between hemoglobin subunit alpha(beta) and myoglobin(Homo sapiens)***Local pairwise sequence alignment***Protein Sequence Alignment between hemoglobin subunit alpha(beta) and myoglobin(Homo sapiens)实验总结1.此次实验,我们基本掌握了如何对两条序列进行全局&局部比对,以及根据不同情况对具体参数进行调整的能力。

双序列比对算法综述作者:王沛来源:《学习与科普》2019年第12期摘要:在生物信息学中,基因序列比对是最基本、最重要的操作。
本文首先介绍了序列比对的划分方式,提出了双序列比对算法的研究意义;接着对典型的双序列比对算法的研究现状进行了较为详细的阐述,包括算法的原理、对比等;然后通过收集双序列比对算法的优化方案,总结出当前算法的发展趋势,得出结论。
关键词:生物信息,序列比对,双序列比对,动态规划,点阵图1 引言序列比对问题是指将基因序列进行比对,将其中相似性的部分标示出来,通过标示出的序列相似度来确定序列间的同源性关系。
在生物信息学中,基因序列的比对是最基本、最重要的操作,是进行基因识别、信息分析、结构预测等问题的前提。
本文将介绍一种最基础的比对方式——双序列比对。
2 背景与意义序列比对有多种划分方式。
根据比对数量的不同,可分为双序列比对和多序列比对。
双序列比对即通过两个基因序列的比对,找到相似的基因片段,从而推测目标基因可能具有的功能以及可能的分子进化关系。
而多序列比对通过多个基因序列的比对,寻找到它们相同的位点、区域,推测具有共同功能的序列模式。
就序列本身而言,对序列进行整体比对的方式称为全局比对,对序列进行部分比对的方式称为局部比对。
全局比对适用于总体相似度高的同源序列;局部比对适用于长度差别大、亲缘关系远的序列,可找出两条序列中相似度最高的片段。
由于双序列比对是基因序列比对最早采取的方式,也是生物信息学最基本的研究方法,所以我决定先从这种最基本的方式入手,了解双序列比对算法的研究现状及发展趋势,为进一步的学习做好铺垫。
3 双序列比对算法研究现状3.1 典型双序列比对算法介绍3.1.1 基于动态规划的双序列比对算法Needleman-Wunsch算法1970年,Needleman和Wunsch最早提出了一种基于动态规划思想的序列全局比对算法:使用迭代的方式求出两个基因序列之间的对比得分,并把结果存放在二维得分矩阵里面,然后运用动态规划方法在二维得分矩阵中进行回溯从而找到序列最佳比对路径,即序列比对的最优结果。

两序列比对算法摘要:序列比对是生物信息学研究的一个基本方法,对于发现生物序列中的功能、结构和进化信息具有重要的意义。
两序列比对中,典型的全局比对算法是Needleman—Wunsch算法;局部比对算法的基础是Smitll—Waterm an 算法,本文对典型的双序列比对算法进行描述。
关键词:生物信息学;两序列比对;算法引言:为了满足基因组中获得更多更有价值的信息,生物信息学迅速发展起来,生物信息学是一门多门科学交叉的学科,将数学、计算机科学应用于生物大分子信息的获取、加工、存储、分类、检索和分析等,以达到阐明和理解大量数据所蕴含的生物学意义的目的。
通过对DNA和蛋白质序列进行相似性比较,指明序列间的保守区域和不同之处,为进一步研究它们在结构、功能以及进化上的联系提供了重要的参考依据。
而序列比对就是运用某种特定的数学模型或算法,找出两个或多个序列之间的最大匹配碱基或残基数,比对的结果反映了算法在多大程度上反映了序列之间的相似性关系以及它们的生物学特征。
双序列比对算法双序列比对分为全局比对和局部比对,全局比对是考察两个序列之间的全局相似性,局部比对则比较序列片段之间的相似性。
Needleman—Wunsch算法是典型的全局比对算法,适用于全局水平上相似性程度较高的两个序列;Smitll—Waterman 算法适用于寻找局部相似序列对,该算法是目前被使用最广泛的序列相似性比较算法之一,由所熟悉的Needleman—Wunsch算法演变而来。
Needleman-Wunsch 算法使用迭代方法计算出两个序列的相似分值,存于一个得分矩阵中,然后根据这个得分矩阵,通过动态规划的方法回溯寻找最优的比对序列。
具有很高的灵敏度使用二维表格,一个序列沿顶部展开,一个序列沿左侧展开。
而且也能通过以下三个途径到达每个单元格:1.来自上面的单元格,代表将左侧的字符与空格比对。
2.来自左侧的单元格,代表将上面的字符与空格比对。

生物信息学中的基因组序列比对算法1. 引言生物信息学是研究生物学信息的存储、分析和应用的学科,其中基因组序列比对算法是重要的研究方向之一。
基因组序列比对是将一个序列与一个或多个目标序列进行比较,以寻找相似性和差异性的过程。
本文将介绍生物信息学中常用的基因组序列比对算法,包括Smith-Waterman算法、Needleman-Wunsch算法和BLAST算法。
2. Smith-Waterman算法Smith-Waterman算法是一种动态规划算法,可以用于比对两个序列之间的相似性。
它的基本思想是通过构建一个得分矩阵,计算两条序列中各个位置之间的得分,然后根据得分确定最佳比对。
具体步骤如下:(1) 构建一个得分矩阵,矩阵的行和列分别表示两条序列的每个字符。
(2) 初始化得分矩阵,将第一行和第一列的得分设为0。
(3) 根据特定的得分规则,计算得分矩阵中每个位置的得分。
得分规则可以根据具体情况进行调整,常见的得分规则包括替换得分、插入得分和删除得分。
(4) 从得分矩阵中找出最高得分的位置,得到最佳比对的结束位置。
(5) 追溯最佳比对的路径,得到最佳比对的开始位置。
Smith-Waterman算法的优点是可以寻找到最佳比对的局部相似性,适用于比对包含插入或删除的序列。
3. Needleman-Wunsch算法Needleman-Wunsch算法是一种全局序列比对算法,通过构建一个得分矩阵和得分规则,计算两个序列的全局相似性。
具体步骤如下:(1) 构建一个得分矩阵,矩阵的行和列分别表示两条序列的每个字符。
(2) 初始化得分矩阵,将第一行和第一列的得分设为特定值。
(3) 根据特定的得分规则,计算得分矩阵中每个位置的得分。
(4) 从得分矩阵中找出最高得分的位置,得到最佳比对的结束位置。
(5) 追溯最佳比对的路径,得到最佳比对的开始位置。
Needleman-Wunsch算法的优点是可以寻找到全局最佳比对,适用于比对两个序列之间的整体相似性。


双序列比对算法
/// 双序列比对用于研究两个序列定义的DNA有多少相似之处,或者蛋白质序列有多少相似之处。
/// 这种比较在DNA鉴定和遗传暗示步骤中是非常重要的,在生物信息学应用中,两个序列之间比较也特别重要,特别是在研究顺序的进化关系和鉴定功能。
///
/// 双序列比对算法主要用于非完全比对,因为完全比对可以利用穷举法,把序列中的元素两两比对,以期发现所需的最近分数最高的所需的比对对。
/// 双序列比对算法可以利用动态规划算法,其中首先定义一个矩阵,表示序列i和序列j的最佳比对,在这个矩阵中,每个条目用于表示以矩阵中元素为末尾的两个序列段的相似度/距离,元素i和元素j越来越相似,这个度量值越大,距离越小。
///
/// 动态规划法大概有两个步骤:第一个步骤是填充一个矩阵,第二个步骤是从矩阵中搜索出最有可能产生最高得分的比对对。

实验三:两条序列比对与多序列比对实验目的:学会使用MegAlign,ClustalX和MUSCLE进行两条序列和多条序列比对分析实验内容:双序列比对是使两条序列产生最高相似性得分的序列排列方式和空格插入方式。
两条序列比对是生物信息学最基础的研究手段。
第一次实验我们用dotplot方法直观地认识了两条序列比对。
但是dotplot仅仅是展示了两条序列中所有可能的配对,并不是真正意义上的序列比对。
这里介绍进行两条序列比对的软件-MegAlign。
多序列比对是将多条序列同时比对,使尽可能多的相同(或相似)字符出现在同一列中。
多序列比对的目标是发现多条序列的共性。
如果说序列两两比对主要用于建立两条序列的同源关系,从而推测它们的结构和功能,那么,同时比对多条序列对于研究分子结构、功能及进化关系更为有用。
多序列比对对于系统发育分析、蛋白质家族成员鉴定、蛋白质结构预测、保守模块的搜寻等具有非常重要的作用。
我们这节课主要学习多条序列比对的软件-ClustalX, MUSCLE。
一、MegAlignDNASTAR公司的Lasergene软件包是一个比较全面的生物信息学软件,它包含了7个模块。
其中MegAlign可进行两条或多条序列比对分析。
1. 两条序列比对1.1 安装程序解压DNASTAR Lasergene软件压缩包,双击Lasergene710WinInstall.exe文件,按照默认路径安装软件到自己电脑上。
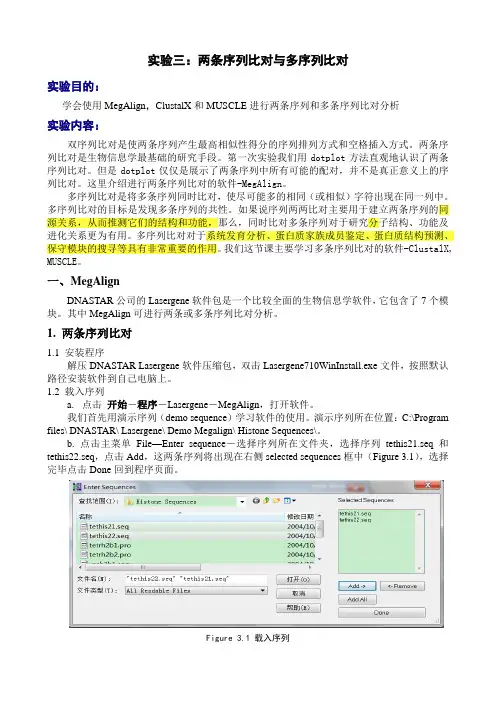
1.2 载入序列a.点击开始-程序-Lasergene-MegAlign,打开软件。
我们首先用演示序列(demo sequence)学习软件的使用。
演示序列所在位置:C:\Program files\ DNASTAR\ Lasergene\ Demo Megalign\ Histone Sequences\。
b. 点击主菜单File—Enter sequence-选择序列所在文件夹,选择序列tethis21.seq和tethis22.seq,点击Add,这两条序列将出现在右侧selected sequences框中(Figure 3.1),选择完毕点击Done回到程序页面。

生物信息学中的序列比对方法序列比对是生物信息学中的核心问题之一。
它是指将两个或多个序列进行比较,以寻找相似性或同源性。
序列比对方法的应用范围非常广泛,包括基因组学、蛋白质组学、微生物学、疫苗设计等领域。
序列比对的重要性自不必言,只有准确的序列比对才能够进行准确的结构预测、功能预测、演化分析等。
序列比对方法可以分为全局比对和局部比对。
全局比对是指将整个序列进行比对,而局部比对则只比对两个序列中的一部分。
全局比对一般用于比较相似的序列,而局部比对则用于比较不同长度和结构的序列。
根据序列比对的算法不同,序列比对方法又可分为动态规划法、启发式算法、图像算法等。
动态规划法是最常见的序列比对算法之一。
它是一种优秀的全局比对算法,在序列相似度计算和演化分析中经常使用。
使用动态规划法进行序列比对的过程非常复杂,需要处理大量的计算和数据。
它的基本思路是将整个序列划分为若干个子序列,然后计算每个子序列的得分,最后将所有子序列的得分相加。
在计算子序列得分的时候,需要考虑序列匹配、序列替换和序列插入删除等操作,通常采用得分矩阵来表示这些操作的得分。
得分矩阵通常由两个序列中的每个位置组成,其中每个位置有一定的得分,表示在这个位置进行匹配、替换、插入或删除操作的得分。
动态规划法的主要优点是它能够得到最优的序列比对结果。
但是,它的计算复杂度非常高,时间和空间占用也非常大,所以在大规模的序列比对中不太适用。
为了解决这个问题,启发式算法应运而生。
启发式算法是一种较快的局部比对算法。
它不断地比较序列中的一部分,直到找到最好的匹配。
由于启发式算法不需要计算整个序列,因此它的计算速度很快。
但是,启发式算法的缺点是它不能保证得到最佳的序列比对结果,可能会漏掉某些相似的序列区域。
图像算法是另一种常用的局部比对算法。
它将序列看作是一幅图像,然后将比对问题转化为图像匹配问题。
图像算法的主要优点是它可以处理大规模的序列比对,同时还可以对序列进行可视化展示。
生物信息学课后习题及答案(由10级生技一、二班课代表整理)一、绪论1.你认为,什么是生物信息学?采用信息科学技术,借助数学、生物学的理论、方法,对各种生物信息(包括核酸、蛋白质等)的收集、加工、储存、分析、解释的一门学科。
2.你认为生物信息学有什么用?对你的生活、研究有影响吗?(1)主要用于:在基因组分析方面:生物序列相似性比较及其数据库搜索、基因预测、基因组进化和分子进化、蛋白质结构预测等在医药方面:新药物设计、基因芯片疾病快速诊断、流行病学研究:SARS、人类基因组计划、基因组计划:基因芯片。
(2)指导研究和实验方案,减少操作性实验的量;验证实验结果;为实验结果提供更多的支持数据等材料。
3.人类基因组计划与生物信息学有什么关系?人类基因组计划的实施,促进了测序技术的迅猛发展,从而使实验数据和可利用信息急剧增加,信息的管理和分析成为基因组计划的一项重要的工作。
而这些数据信息的管理、分析、解释和使用促使了生物信息学的产生和迅速发展。
4简述人类基因组研究计划的历程。
通过国际合作,用15年时间(1990-2005)至少投入30亿美元,构建详细的人类基因组遗传图和物理图,确定人类DNA的全部核苷酸序列,定位约10万基因,并对其他生物进行类似研究。
1990,人类基因组计划正式启动。
1996,完成人类基因组计划的遗传作图,启动模式生物基因组计划。
1998完成人类基因组计划的物理作图,开始人类基因组的大规模测序。
Celera公司加入,与公共领域竞争启动水稻基因组计划。
1999,第五届国际公共领域人类基因组测序会议,加快测序速度。
2000,Celera公司宣布完成果蝇基因组测序,国际公共领域宣布完成第一个植物基因组——拟南芥全基因组的测序工作。
2001,人类基因组“中国卷”的绘制工作宣告完成。
2003,中、美、日、德、法、英等6国科学家宣布人类基因组序列图绘制成功,人类基因组计划的.目标全部实现。
2004,人类基因组完成图公布。
双序列对比的原理双序列对比是指对比两个序列之间的结构、相似性、差异性或演化关系的一种方法。
它广泛应用于生物学研究中,可以帮助研究人员理解不同物种的关系、进化过程以及功能区域等方面的信息。
双序列对比的原理基于两个序列的比对和分析。
在双序列对比中,两个序列通常是DNA、RNA或蛋白质序列。
在进行对比之前,需要先经过序列比对的步骤,即将两个序列按照一定的规则对齐,使得相同的部分对齐在一起并显示出来,不同的部分则可以通过间隔、替代或插入的方式标示出来。
比对的目标是找到两个序列之间的共同特征,以便更好地分析和解释它们之间的关系。
在进行双序列对比时,常用的比对算法包括Smith-Waterman算法和Needleman-Wunsch算法。
这些算法通过动态规划的方法,计算出两个序列之间的最优比对方案。
最优比对方案是指在符合一定规则的前提下,使得比对结果得分最高或最接近实际情况的方案。
通过比对算法计算得到的比对结果,可以用于查找相似性或共有区域,也可以用于推测序列之间的演化关系。
双序列对比还可以通过比对结果进行比对分析。
比对分析可以通过不同的方法和步骤来实现。
常见的比对分析方法包括序列标注、格局分析、功能预测和进化树构建等。
序列标注是指对比对结果中的不同部分进行注释和分析,比如标示出替代、插入和缺失的位置,这对于比对结果的解读非常重要。
格局分析是指对比对结果中重复出现的序列单元进行统计和分析,以便预测可能的功能区域。
功能预测是指根据比对结果推测序列的功能和结构特征,比如预测蛋白质序列中的位点、结构域和功能基序。
进化树构建是指基于比对结果推测不同序列之间的演化关系,从而揭示物种的进化历程和亲缘关系。
双序列对比的核心原理是在比对的基础上进行分析和解读。
通过比对两个序列,可以获得它们之间的结构和相似性信息,并进一步推测它们之间的差异性和演化关系。
双序列对比可以应用于多个领域,例如进化生物学、比较基因组学、疾病研究等。
常用序列比对
常用的序列比对方法包括:
1. 双序列比对:将两个序列进行比对,找到它们之间的相似性和差异性。
这是最基本的序列比对方法,常用于基因序列比对、蛋白质序列比对等。
2. 多序列比对:将多个序列进行比对,找到它们之间的共同特征和差异性。
这可以帮助研究人员发现不同物种或不同基因之间的进化关系。
3. 局部比对:在双序列或多序列比对中,只比较其中的一部分序列,而不是整个序列。
这种方法常用于寻找特定区域的相似性,例如蛋白质结构域的比对。
4. 动态规划比对:这是一种基于动态规划算法的比对方法,通过计算不同位置的相似性得分来找到最优比对。
这种方法可以有效地处理长序列比对,并在时间和空间复杂度上具有较好的性能。
5. Smith-Waterman 比对:这是一种经典的局部比对方法,通过在比对过程中引入空位罚分来处理插入和删除操作。
Smith-Waterman 比对常用于生物信息学领域,如基因序列比对和蛋白质序列比对。
6. 启发式比对:一些基于启发式规则的比对方法,如BLAST(Basic Local Alignment Search Tool)和 FASTA,通过使用索引和搜索算法来加速比对过程。
这些方法常用于大规模数据库搜索和序列相似性分析。
这些序列比对方法在不同的应用场景中具有各自的优势和适用范围。
选择合适的比对方法取决于具体的需求和问题的特点。
一、实验目的1. 掌握多序列比对的基本原理和方法。
2. 熟悉使用BLAST、CLUSTAL W等工具进行多序列比对。
3. 分析比对结果,了解序列间的进化关系。
二、实验原理多序列比对是指将两个或多个生物序列进行排列,以揭示序列间的相似性和进化关系。
通过比对,可以识别保守区域、功能域和结构域,为生物信息学研究和进化生物学研究提供重要依据。
多序列比对的方法主要包括以下几种:1. 动态规划法:通过构建一个动态规划表,计算最优比对路径,实现序列的比对。
2. 人工比对法:通过分析序列结构、功能域等信息,人工进行比对。
3. 基于启发式算法的比对:通过寻找序列间的相似性,快速进行比对。
三、实验材料1. 仿刺参EGFR基因氨基酸序列(Fasta格式)。
2. 同源序列数据库(如NCBI)。
3. 多序列比对软件(如BLAST、CLUSTAL W)。
四、实验步骤1. 使用BLAST工具进行同源序列搜索。
(1)在NCBI网站上,选择“BLAST”功能。
(2)将仿刺参EGFR基因氨基酸序列粘贴到“Query Sequence”框中。
(3)选择合适的比对参数,如“MegaBLAST”。
(4)点击“BLAST”按钮,等待结果。
(5)在结果页面,找到相似度最高的几个序列,下载下来。
2. 使用CLUSTAL W进行多序列比对。
(1)将下载的同源序列整合到一个Fasta格式的文本文件中。
(2)在CLUSTAL W软件中,选择“Multiple Sequence Alignment”功能。
(3)上传Fasta格式的文本文件。
(4)选择合适的比对参数,如“Gap Penalty”和“Gap Reward”。
(5)点击“Align”按钮,等待结果。
3. 分析比对结果。
(1)观察比对结果,分析序列间的相似性和进化关系。
(2)绘制系统进化树,展示序列的进化历程。
五、实验结果与分析1. 使用BLAST工具,找到与仿刺参EGFR基因氨基酸序列相似度最高的几个序列,如Anopheles gambiae、Nasonia vitripennis等。