R语言学习知识中的多元统计之判别分析
- 格式:doc
- 大小:139.26 KB
- 文档页数:12

有用的统计学Statistics第7讲分类方法中央财经大学统计与数学学院7.3判别分析和Logistic回归的R语言操作head(iris) #本例使用iris数据,展示iris数据的5个变量和前几行样本观测值summary(iris$Species) # 输出三个类的样本量by(iris[, 1:4], iris[,5], colMeans) #比较三类鸢尾花在4个变量上的均值library(MASS) #加载MASS包z <-lda(Species ~ ., iris, prior = c(1,1,1)/3) #Fisher 判别,设定三个类别的先验概率均为1/3z # 输出估计结果iris.lda.values<-predict(z) # 对现有样本做出预测ldahist(data = iris.lda.values$x[,1], g=iris$Species) #输出在第一个方向上、三个类别投影的直方图ldahist(data = iris.lda.values$x[,2],g=iris$Species) #输出在第二个方向上、三个类别投影的直方图图1在第一个方向上、三个类别投影的直方图图2在第二个方向上、三个类别投影的直方图Logistic回归library(ElemStatLearn)#加载ElemStatLearn包head(SAheart) #本例使用SAheart数据,这是关于冠状动脉心脏病的数据,展示SAheart数据的变量和前几行样本观测值summary(factor(SAheart$chd))#查看两个类的样本量glm.SA<-glm(chd~. , family=binomial(link="logit"), data= SAheart) #建立全模型summary(glm.SA) # 输出全模型结果glm.SA.step<-step(glm.SA, direction = "both") #逐步回归summary(glm.SA.step)# 输出逐步回归结果predict(glm.SA.step, SAheart, type = "response") # 预测样本的患病概率。

#第十章多元统计分析介绍#10.1 主成分分析与因子分析#10.1.1 主成分的简要定义与计算#10.1.2 主成分R 通用程序student<-data.frame(X1=c(148, 139, 160, 149, 159, 142, 153, 150, 151, 139, 140, 161, 158, 140, 137, 152, 149, 145, 160, 156,151, 147, 157, 147, 157, 151, 144, 141, 139, 148),X2=c(41, 34, 49, 36, 45, 31, 43, 43, 42, 31,29, 47, 49, 33, 31, 35, 47, 35, 47, 44,42, 38, 39, 30, 48, 36, 36, 30, 32, 38),X3=c(72, 71, 77, 67, 80, 66, 76, 77, 77, 68,64, 78, 78, 67, 66, 73, 82, 70, 74, 78,73, 73, 68, 65, 80, 74, 68, 67, 68, 70),X4=c(78, 76, 86, 79, 86, 76, 83, 79, 80, 74,74, 84, 83, 77, 73, 79, 79, 77, 87, 85,82, 78, 80, 75, 88, 80, 76, 76, 73, 78))student.pr<-princomp(student, cor=TRUE)summary(student.pr,loadings=TRUE)#10.1.3 因子分析的简要定义与计算#10.1.4 因子分析R 通用程序student<-read.table("e:/data/student.txt")names(student)=c("math", "phi", "chem", "lit", "his", "eng") fa<-factanal(student, factors=2)fa#10.2 判别分析#10.2.1 距离判别#10.2.2 Fisher 判别法#10.2.3 R 通用程序library(MASS)data(iris)attach(iris)names(iris)library(MASS)iris.lda <- lda(Species ~ Sepal.Length + Sepal.Width+ Petal.Length + Petal.Width)【原创】定制代写开发r/python/spss/matlab/WEKA/sas/sql/C++/stata/eviews数据挖掘和统计分析可视化调研报告程序等服务(附代码数据),咨询:3025393450@有问题到淘宝找“大数据部落”就可以了iris.ldairis.pred=predict(iris.lda) $ classtable(iris.pred, Species)detach(iris)w <- read.table("e:/data/disc.txt")names(w)=c("group", "x1", "x2", "x3", "x4")library(MASS)z <- lda(group~x1+x2+x3+x4, data=w, prior=c(1, 1)/2) newdata<-rbind(c(8.85, 3.38, 5.17, 26.10), c(28.60, 2.40, 1.20, 127.0),c(20.70, 6.70, 7.60, 30.20), c(7.90, 2.40, 4.30, 33.20),c(3.19, 3.20, 1.43, 9.90), c(12.40, 5.10, 4.43, 24.60),c(16.80, 3.40, 2.31, 31.30), c(15.00, 2.70, 5.02, 64.00)) dimnames(newdata)<-list(NULL, c("x1", "x2", "x3", "x4")) newdata<-data.frame(newdata)predict(z, newdata=newdata)#10.3 聚类分析#10.3.1 基本思想#10.3.2 R通用程序x<-c(1, 2, 4.5, 6, 8)dim(x)<-c(5, 1)d<-dist(x)hc1<-hclust(d, "single")hc2<-hclust(d, "complete")hc3<-hclust(d, "median")hc4<-hclust(d, "ward")opar<-par(mfrow=c(2, 2))plot(hc1, hang=-1);plot(hc2, hang=-1)plot(hc3, hang=-1);plot(hc4, hang=-1)par(opar)data(iris);attach(iris)iris.hc<-hclust(dist(iris[,1:4]))plot(iris.hc, hang = -1)plclust(iris.hc,labels = FALSE, hang=-1)re<-rect.hclust(iris.hc,k=3)iris.id <- cutree(iris.hc,3)table(iris.id,Species)#10.4 典型相关分析#10.4.1 基本思想#10.4.2 R通用程序invest=read.table("e:/data/invest.txt")names(invest)=c("x1", "x2", "x3", "x4", "x5", "x6", "y1", "y2", "y3", "y4", "y5")ca<-cancor(invest[, 1:6], invest[, 7:11])ca#x10.5 对应分析#10.5.1 基本思想#10.5.2 R通用程序x.df=data.frame(HighlyFor=c(2, 6, 41, 72, 24), For =c(17, 65, 220, 224, 61),Against=c(17, 79, 327, 503, 300), HighlyAgainst=c(5, 6, 48, 47, 41))rownames(x.df)<-c("BelowPrimary", "Primary", "Secondary", "HighSchool","College")library(MASS)biplot(corresp(x.df, nf=2))。

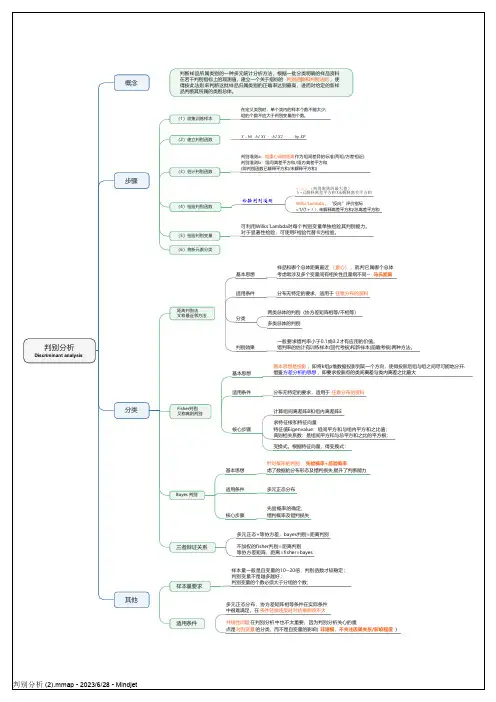
判别分析Discriminant analysis 概念判断样品所属类别的一种多元统计分析方法,根据一批分类明确的样品资料在若干判别指标上的观测值,建立一个关于指标的判别函数和判别法则,使得按此法则来判断这批样品归属类别的正确率达到最高,进而对给定的新样品判断其所属的类别总体。
步骤(1)收集训练样本在定义类别时,单个类内的样本个数不能太少;组的个数不应大于判别变量的个数。
(2)建立判别函数Y b0b1 X1b2 X2bp XP(3)估计判别函数判别准则a:组重心间的距离作为组间差异的标准(两组/方差相近)判别准则b:组间离差平方和/组内离差平方和(即判别函数已解释平方和/未解释平方和)(4)检验判别函数检验判别准则(判别准则的最大值)λ=已解释离差平方和/未解释离差平方和Wilks'Lambda,“反向”评价指标=1/(1+λ),未解释离差平方和/总离差平方和(5)检验判别变量可利用Wilks'Lambda对每个判别变量单独检验其判别能力。
对于显著性检验,可使用F检验代替卡方检验。
(6)将新元素分类分类距离判别法又称最近邻方法基本思想样品和哪个总体距离最近(重心),就判它属哪个总体考虑常涉及多个变量间有相关性且量纲不同--马氏距离适用条件分布无特定的要求,适用于任意分布的资料分类两类总体的判别(协方差矩阵相等/不相等)多类总体的判别判别效果一般要求错判率小于0.1或0.2才有应用的价值。
错判率的估计有训练样本(回代考核)和新样本(前瞻考核)两种方法。
Fisher判别又称典则判别基本思想基本思想是投影,即将k组p维数据投影到某一个方向,使得投影后组与组之间尽可能地分开.借鉴方差分析的思想,即要求投影点的类间离差与类内离差之比最大适用条件分布无特定的要求,适用于任意分布的资料核心步骤计算组间离差阵B和组内离差阵E求特征根和特征向量特征值Eigenvalue:组间平方和与组内平方和之比值;典则相关系数:是组间平方和与总平方和之比的平方根;变换式。



R语⾔中Fisher判别的使⽤⽅法最近编写了Fisher判别的相关代码时,需要与已有软件⽐照结果以确定⾃⼰代码的正确性,于是找到了安装⽅便且免费的R。
这⾥把R中进⾏Fisher判别的⽅法记录下来。
1. 判别分析与Fisher判别不严谨但是通俗的说法,判别分析(Discriminant Analysis)是⼀种多元(多个变量)统计分析⽅法,它根据样本的多个已知变量的值对样本进⾏分类的⽅法。
⼀般来说,判别分析由两个阶段构成——学习(训练)和判别。
在学习阶段,给定⼀批已经被分类好的样本,根据它们的分类情况和样本的多个变量的值来学习(训练)得到⼀种判别⽅法;在判别阶段⽤前⼀阶段得到的判别⽅法对其他样本进⾏判别。
Fisher判别(Fisher Discrimination Method)⼜被称为线性判别(LDA,Linear Discriminative Analysis),是判别分析的⼀种,历史可以追溯到1936年。
它的核⼼思想是将多维数据(多个变量)投影(使⽤线性运算)到⼀维(单⼀变量)上,然后通过给定阈值将样本根据投影后的单⼀变量进⾏分类。
Fisher判别的学习(训练)阶段,就是找到合适的投影⽅式,使得对于已经被分类好的样本,同⼀类的样本被投影后尽量扎堆。
学习阶段的结果是找到⼀系列的系数(Coeffcient),构成形如y=a1 * x1 + a2 * x2 + a3 * x3 + ... + an * xn其中:a1,a2,... an是系数,x1,x2,... ,xn是变量值。
的判别式和阈值。
⽽判别阶段可以根据这个判别式计算出y,并根据阈值将样本进⾏分类。
2. 在R中使⽤Fisher判别R中使⽤Fisher判别说起来很简单,但是我当初也放狗搜索了不短的时间才搞明⽩如何使⽤。
⾸先,它在R⾥不叫Fisher,⽤Fisher搜索多半误⼊歧途。
在R中,它叫LDA(Linear Discriminative Analysis)。

利用R语言如何判别和分类楼主在学习数据挖掘期间,老师讲了很多的判别和分类方法,只是没有平时时间整理,这次利用周末的时间特地整理自己以前的知识点,这篇文章会引用大量网上的图片和文字,若有侵权,及时告知,本人会马上修改。
这篇文章中的案例统一使用著名的鸢尾花数据。
若有错误,也请及时指出,大家相互学习,共同进步判别分析(discriminant analysis)是一种分类技术。
它通过一个已知类别的“训练样本”来建立判别准则,并通过预测变量来为未知类别的数据进行分类。
判别分析根据所采用的数据模型,可分为线性判别分析和非线性判别分析。
根据判别准则可分为Fisher判别、Bayes判别和距离判别。
其中最基本的Fisher判别方法也被称为线性判别方法。
该方法的主要思想是将多维数据投影到某个方向上,投影的原则是将总体与总体尽可能的分开,然后再选择合适的判别规则将新的样本分类判别。
Fisher判别会投影降维,使多维问题简化为一维问题来处理。
选择一个适当的投影轴,使所有的样品点都投影到这个轴上得到一个投影值。
对这个投影轴的方向的要求是:使每一组内的投影值所形成的组内离差尽可能小,而不同组间的投影值所形成的类间离差尽可能大。
Bayes 判别思想是根据先验概率求出后验概率,并依据后验概率分布作出统计推断。
距离判别思想是根据已知分类的数据计算各类别的重心,对未知分类的数据,计算它与各类重心的距离,与某个重心距离最近则归于该类。
1.线性判别当不同类样本的协方差矩阵相同时,我们可以在R中使用MASS 包的lda函数实现线性判别。
值得注意的是当分类只有两种且总体服从多元正态分布条件下,Bayes判别与Fisher判别、距离判别是等价的。
利用table函数建立混淆矩阵,比对真实类别和预测类别。
> library(MASS)> data(iris)> iris.lda=lda(Species~.,data=iris)> table(Species,predict(iris.lda,iris)$class)Species setosa versicolor virginicasetosa 50 0 0versicolor 0 48 2virginica 0 1 49> table<-table(Species,predict(iris.lda,iris)$class)> sum(diag(prop.table(table)))###判对率[1] 0.982.二次判别当不同类样本的协方差矩阵不同时,则应该使用二次判别。

多元统计分析公式主成分分析判别分析多元统计分析是一种通过收集和分析多个变量之间相互作用关系来帮助我们理解、解释和预测数据的方法。
其中,主成分分析和判别分析是常用的多元统计分析方法。
本文将对这两种方法的公式和应用进行介绍。
一、主成分分析主成分分析(Principal Component Analysis,简称PCA)是一种通过线性变换将一组可能存在相关性的变量转化为一组线性无关的新变量的方法。
它的基本思想是通过将原始变量进行线性组合来构建主成分,这些主成分能够解释原始数据中大部分的方差。
主成分分析的公式如下:X = A * T其中,X是原始数据矩阵,A是变量相关系数矩阵,T是主成分得分矩阵。
主成分分析的步骤如下:1. 标准化数据:将原始数据标准化,确保各个变量具有相同的尺度。
2. 计算相关系数矩阵:计算标准化后的数据的相关系数矩阵A。
3. 计算特征值和特征向量:对相关系数矩阵进行特征值分解,得到特征值和对应的特征向量。
4. 选择主成分:根据特征值的大小选择前n个主成分。
5. 计算主成分得分:将原始数据投影到所选的主成分上,得到主成分得分矩阵T。
主成分分析的应用十分广泛,常用于降维、数据可视化、变量选择等领域。
例如,在社会科学研究中,可以将大量的社会经济指标通过主成分分析进行降维,从而更好地理解社会现象。
二、判别分析判别分析(Discriminant Analysis)是一种帮助我们根据已知类别数据预测未知类别数据的方法。
判别分析通过寻找最佳投影方向,将不同类别的样本在投影后最大程度地分离开来,从而提高分类的准确性。
判别分析的公式如下:D = W * X其中,D是判别得分,W是权重系数,X是原始数据。
判别分析的步骤如下:1. 计算类内散度矩阵和类间散度矩阵:分别计算各个类别的散度矩阵。
2. 计算广义特征值和广义特征向量:对类内散度矩阵和类间散度矩阵进行广义特征值分解,得到广义特征值和对应的广义特征向量。

Equation Chapter 1 Section 1 Array《多元统计分析》Multivariate Statistical Analysis主讲:统计学院统计学院应用统计学教研室School of Statistics第三章 判别分析【教学目的】1. 让学生了解判别分析的背景、基本思想; 2. 掌握判别分析的基本原理与方法; 3. 掌握判别分析的操作步骤和基本过程; 4. 学会应用聚类分析解决实际问题。
【教学重点】1. 注意判别分析与聚类分析的关系(联系与区别); 2. 阐述各种判别分析方法。
§1 概述一、什么是判别分析1.研究背景科学研究中,经常会遇到这样的问题:某研究对象以某种方式(如先前的结果或经验)已划分成若干类型,而每一类型都是用一些指标()12,,,p X X X X '=来表征的,即不同类型的X 的观测值在某种意义上有一定的差异。
当得到一个新样本观测值(或个体)的关于指标X 的观测值时,要判断该样本观测值(或个体)属于这几个已知类型中的哪一个,这类问题通常称为判别分析。
也就是说,判别分析(discriminant analysis )是根据所研究个体的某些指标的观测值来推断该个体所属类型的一种统计方法。
判别分析的应用十分广泛。
例如,在工业生产中,要根据某种产品的一些非破坏性测量指标判别产品的质量等级;在经济分析中,根据人均国民收入,人均工农业产值,人均消费水平等指标判断一个国家的经济发展程度;在考古研究中,根据挖掘的古人头盖骨的容量,周长等判断此人的性别;在地质勘探中,根据某地的地质结构,化探和物探等各项指标来判断该地的矿化类型;在医学诊断中,医生要根据某病人的化验结果和病情征兆判断病人患哪一种疾病,等等。
值得注意的是,作为一种统计方法,判别分析所处理的问题一般都是机理不甚清楚或者基本不了解的复杂问题,如果样本观测值的某些观测指标和其所属类型有必然的逻辑关系,也就没有必要应用判别分析方法了。
基于R语言的判别分析方法和分类方法判别分析(discriminant analysis)是一种分类技术。
它通过一个已知类别的“训练样本”来建立判别准则,并通过预测变量来为未知类别的数据进行分类。
判别分析根据所采用的数据模型,可分为线性判别分析和非线性判别分析。
根据判别准则可分为Fisher判别、Bayes判别和距离判别。
其中最基本的Fisher判别方法也被称为线性判别方法。
该方法的主要思想是将多维数据投影到某个方向上,投影的原则是将总体与总体尽可能的分开,然后再选择合适的判别规则将新的样本分类判别。
Fisher判别会投影降维,使多维问题简化为一维问题来处理。
选择一个适当的投影轴,使所有的样品点都投影到这个轴上得到一个投影值。
对这个投影轴的方向的要求是:使每一组内的投影值所形成的组内离差尽可能小,而不同组间的投影值所形成的类间离差尽可能大。
Bayes判别思想是根据先验概率求出后验概率,并依据后验概率分布作出统计推断。
距离判别思想是根据已知分类的数据计算各类别的重心,对未知分类的数据,计算它与各类重心的距离,与某个重心距离最近则归于该类。
1.线性判别当不同类样本的协方差矩阵相同时,我们可以在R中使用MASS 包的lda函数实现线性判别。
值得注意的是当分类只有两种且总体服从多元正态分布条件下,Bayes判别与Fisher判别、距离判别是等价的。
利用table函数建立混淆矩阵,比对真实类别和预测类别。
> library(MASS)> data(iris)> iris.lda=lda(Species~.,data=iris)> table(Species,predict(iris.lda,iris)$class)Species setosa versicolor virginicasetosa 50 0 0versicolor 0 48 2virginica 0 1 49> table<-table(Species,predict(iris.lda,iris)$class)> sum(diag(prop.table(table)))###判对率[1] 0.982.二次判别当不同类样本的协方差矩阵不同时,则应该使用二次判别。
多元统计与r语言建模多元统计与R语言建模引言:多元统计分析是统计学中的一种重要方法,用于研究多个变量之间的关系和相互影响。
而R语言作为一种开源的统计计算和绘图软件,具有强大的数据分析和建模能力。
本文将介绍多元统计分析的基本概念和常用方法,并结合R语言进行建模实例。
一、多元统计分析的基本概念1. 多元统计分析的目的:多元统计分析旨在探索和解释多个变量之间的关系,以及变量与其他因素之间的关联。
2. 变量类型:在多元统计分析中,变量可以分为两大类:定性变量和定量变量。
定性变量是指具有类别或标签的变量,如性别、学历等;定量变量是指具有数值意义的变量,如年龄、收入等。
3. 多元统计方法:常用的多元统计方法包括:主成分分析、因子分析、聚类分析、判别分析、回归分析等。
二、R语言在多元统计分析中的应用1. R语言简介:R语言是一种功能强大的统计计算和绘图软件,具有丰富的数据分析函数和扩展包,可以进行各种统计分析和建模。
2. R语言的优势:R语言具有开源免费、社区活跃、生态丰富、可扩展性强等优势,使其成为统计学家和数据分析师的首选工具。
3. R语言的应用:R语言可以应用于数据预处理、描述性统计分析、假设检验、回归建模、分类与聚类分析等多元统计分析任务。
三、基于R语言的多元统计建模实例为了更好地理解多元统计分析方法和R语言的应用,我们将以一个实际案例展示如何使用R语言进行多元统计建模。
案例背景:某电商平台想要了解用户购买行为与用户特征之间的关系,以便制定个性化的推荐策略。
为此,我们收集了一份包含用户购买行为和用户特征的数据集。
数据准备:我们需要导入数据集并进行数据预处理。
这包括数据清洗、数据变换和缺失值处理等步骤。
在R语言中,可以使用各种函数和包来完成这些任务。
数据探索:在进行多元统计建模之前,我们需要对数据进行探索和描述性统计分析。
这可以帮助我们了解数据的分布、关联性和异常值等信息。
R 语言提供了丰富的可视化函数和统计函数,如直方图、散点图、相关系数等。
R:判别分析判别与聚类的⽐较:聚类分析和判别分析有相似的作⽤,都是起到分类的作⽤。
判别分析是已知分类然后总结出判别规则,是⼀种有指导的学习;聚类分析则是有了⼀批样本,不知道它们的分类,甚⾄连分成⼏类也不知道,希望⽤某种⽅法把观测进⾏合理的分类,使得同⼀类的观测⽐较接近,不同类的观测相差较多,这是⽆指导的学习。
所以,聚类分析依赖于对观测间的接近程度(距离)或相似程度的理解,定义不同的距离量度和相似性量度就可以产⽣不同的聚类结果判别分析基本原理:从已知的各种分类情况中总结规律(训练出判别函数),当新样品进⼊时,判断其与判别函数之间的相似程度(概率最⼤,距离最近,离差最⼩等判别准则)。
常⽤判别⽅法:最⼤似然法,Bayes判别法,距离判别法,Fisher判别法,逐步判别法等。
注意事项:1. 判别分析的基本条件:分组类型在两组以上,解释变量必须是可测的;2. 每个解释变量不能是其它解释变量的线性组合(⽐如出现多重共线性情况时,判别权重会出现问题);3. 各解释变量之间服从多元正态分布(不符合时,可使⽤Logistic回归替代),且各组解释变量的协⽅差矩阵相等(各组协⽅⽅差矩阵有显著差异时,判别函数不相同)。
相对⽽⾔,即使判别函数违反上述适⽤条件,也很稳健,对结果影响不⼤。
应⽤领域:对客户进⾏信⽤预测,寻找潜在客户(是否为消费者,公司是否成功,学⽣是否被录⽤等等),临床上⽤于鉴别诊断。
本⽂中分三个⽅法介绍判别分析,Bayes判别,距离判别,Fisher判别。
Bayes 和距离判别:都要考虑两个、或多个总体协⽅差(⽅差是协⽅差的⼀种)相等或不等的情况,由 var.equal= 的逻辑参数表⽰,默认是 FALSE,表⽰认为两总体协⽅差不等。
⽤样本的协⽅差可以估计总体的协⽅差。
在Bayes⽅法中我们把相等和不等的两个结果都列了出来,距离判别⾥我们默认两总体协⽅差不等。
⼀般使⽤时,我们都以两总体的协⽅差不等作为标准来进⾏后续计算。
.\ 前言 判别分析(discriminant analysis)是多元统计分析中较为成熟的一种分类方法,它的核心思想是“分类与判断”,即根据已知类别的样本所提供的信息,总结出分类的规律性,并建立好判别公式和判别准则,在此基础上,新的样本点将按照此准则判断其所属类型。例如,根据一年甚至更长时间的每天的湿度差及压差,我们可以建立一个用于判别是否会下雨的模型,当我们获取到某一天(建立模型以外的数据)的湿度差及压差后,使用已建立好的模型,就可以得出这一天是否会下雨的判断。
根据判别的组数来区分,判别分析可以分为两组判别和多组判别。接下来,我们将学习三种常见的判别分析方法,分别是:
• 距离判别 • Bayes判别 • Fisher判别
一、距离判别基本理论 假设存在两个总体和,另有为一个维的样本值,计算得到该样本到两个总体的距离和,如果大于,则认为样本属于总体,反之样本则属于总体;若等于,则该样本待判。这就是距离判别法的基本思想。 .\ 在距离判别法中,最核心的问题在于距离的计算,一般情况下我们最常用的是欧式距离,但由于该方法在计算多个总体之间的距离时并不考虑方差的影响,而马氏距离不受指标量纲及指标间相关性的影响,弥补了欧式距离在这方面的缺点,其计算公式如下:
,为总体之间的协方差矩阵
二、距离判别的R实现(训练样本) 首先我们导入数据 # 读取SAS数据 > library(sas7bdat) > data1 <- read.sas7bdat('disl01.sas7bdat') # 截取所需列数据,用于计算马氏距离 > testdata <- data1[2:5] > head(testdata,3) X1 X2 X3 X4 1 -0.45 -0.41 1.09 0.45 2 -0.56 -0.31 1.51 0.16 3 0.06 0.02 1.01 0.40 # 计算列均值 > colM <- colMeans(testdata) > colM .\ X1 X2 X3 X4 0.096304348 -0.006956522 2.033478261 0.431739130 # 计算矩阵的协方差 > cov_test <- cov(testdata) > cov_test X1 X2 X3 X4 X1 0.068183816 0.027767053 0.14996870 -0.002566763 X2 0.027767053 0.015363865 0.05878251 0.001252367 X3 0.149968696 0.058782512 1.01309874 0.028607150 X4 -0.002566763 0.001252367 0.02860715 0.033912464 # 样本的马氏距离计算 > distance <- mahalanobis(testdata,colM,cov_test) > head(distance,5) [1] 12.726465 11.224681 1.692702 1.347885 2.369820
这样,我们得到了距离判别中最关键的马氏距离值,在此基础上就可以进行进一步的判别分析了。不过我们介绍一个R的第三方包WMDB,该包的wmd()函数可以简化我们的距离判别过程,函数将输出样本的分类判别结果、错判的样本信息以及判别分析的准确度。 > library(WMDB) > head(data1,3) A X1 X2 X3 X4 1 1 -0.45 -0.41 1.09 0.45 2 1 -0.56 -0.31 1.51 0.16 3 1 0.06 0.02 1.01 0.40 .\ # 提取原始数据集的A列生成样品的已知类别 > testdata_group <- data1$A # 转换为因子变量,用于wmd()函数中 > testdata_group <- as.factor(testdata_group) > wmd(testdata,testdata_group) 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27
blong 1 1 1 1 1 1 1 1 1 1 1 1 1 1 2 2 1 1 1 2 1 1 1 1 2 2 2
28 29 30 31 32 33 34 35 36 37 38 39 40 41 42 43 44 45 46 blong 2 2 2 2 2 2 1 2 2 2 1 1 1 1 1 2 1 2 2 [1] "num of wrong judgement" [1] 15 16 20 22 23 24 34 38 39 40 41 42 44 [1] "samples divided to" [1] 2 2 2 1 1 1 1 1 1 1 1 1 1 [1] "samples actually belongs to" [1] 1 1 1 2 2 2 2 2 2 2 2 2 2 Levels: 1 2 [1] "percent of right judgement" [1] 0.7173913
由分析结果可知,根据已知分类的训练样品建立的判别规则,重新应用于训练样品后,出现了13个错判样品,拥有71.7%的准确度。
三、距离判别的R实现(测试样本) .\ 接着,当我们获取到未分类的新样本数据时,使用wmd()函数,在训练样本的基础上进行这些数据的距离判别 # 导入数据,一共10个样本 > data2 <- read.sas7bdat('disldp01.sas7bdat') # 截取所需列数据 > newtestdata <- data2[1:4] # 进行判别分析 > wmd(testdata,testdata_group,TstX = newtestdata) 1 2 3 4 5 6 7 8 9 10 blong 1 1 1 1 1 1 2 2 2 1
根据马氏距离判别分析得到的结果,10个待判样品中,第一类7个,第二类3个。
距离判别方法简单实用,它只要求知道总体的数字特征,而不涉及总体的分布,当总体均值和协方差未知时,就用样本的均值和协方差矩阵来估计,因此距离判别没有考虑到每个总体出现的机会大小,即先验概率,没有考虑到错判的损失。因此,我们进一步学习贝叶斯判别法。
一、贝叶斯判别基本理论 贝叶斯判别法的前提是假定我们已经对所要分析的数据有所了解(比如数据服从什么分别,各个类别的先验概率等),根据各个类别的先验概率求得新样本属于某类的后验概率。该算法应用到经典的贝叶斯公式,该公式为: .\ 假设有两个总体和,分别具有概率密度函数和,并且根据以往的统计分析,两个总体各自出现的先验概率为和,当一个样本发生时,求该样本属于某一类的概率,计算公式为:
这样,我们得到了该样本属于两类总体的概率,分别为和,属于哪一类总体的概率值大,我们则将样本划分到该类中。
二、贝叶斯判别的R实现 在R中,我们使用klaR包中的NaiveBayes()函数实现贝叶斯判别分析,函数调用公式如下: > NaiveBayes(formula, data, ..., subset, na.action = na.pass) # formula指定参与模型计算的变量,以公式形式给出,类似于y=x1+x2+x3 # na.action指定缺失值的处理方法,默认情况下不将缺失值纳入模型计算,也不会发生报错信息,当设为“na.omit”时则会删除含有缺失值的样本
# 数据准备,使用R内置数据集iris # 通过抽样建立训练样本(70%)和测试样本(30%) > index <- sample(2,size = nrow(iris),replace = TRUE,prob = c(0.7,0.3)) > train_data <- iris[index == 1,] > test_data <- iris[index == 2,] .\ # 载入所用包 > library(klaR) # 构建贝叶斯模型 > Bayes_model <- NaiveBayes(Species ~ ., data = train_data) # 进行预测 > Bayes_model_pre <- predict(Bayes_model, newdata = test_data[,1:4]) # 生成实际与预判交叉表 > table(test_data$Species,Bayes_model_pre$class)
setosa versicolor virginica setosa 20 0 0 versicolor 0 17 0 virginica 0 3 7
从上表生成的交叉表中,我们可以看到在该模型中错判了3个。 # 生成预判精度 > sum(diag(table(test_data$Species,Bayes_model_pre$class))) + / sum(table(test_data$Species,Bayes_model_pre$class)) [1] 0.9361702
三、Fisher判别基本理论 Fisher判别法的基本思想是“投影”,将组维的数据向低维空间投影,使其投影的组与组之间的方差尽可能的大,组内的方差尽可能的小。因