11月13日 引物合成的详解 4.需要什么级别的引物? 答:引物常用的纯化方式C18脱盐,OPC纯化,PAGE纯化,HPLC纯化。根据实验需要,确定订购引物的纯度级别。 应用引物长度要求纯度级别要求 一般PCR扩增<45 base OPC 一般PCR扩增>45 base PAGE 诊断PCR扩增< 40base OPC, PAGE DNA测序20base左右OPC 亚克隆,点突变等根据实验要求定OPC, PAGE,HPLC 根据实验要求定PAGE 基因构建(全基因合成) 反义核酸根据实验要求定PAGE PAGE, HPLC 修饰引物根据实验要求定 8.如何计算引物的浓度? 答:引物保存在高浓度的状况下比较稳定。引物一般配制成 10-50pmol/ul。一般情况下,建议将引物的浓度配制成50pmol/ul,加水的体积(微升)按下列方式计算:V (微升)= OD数*(乘)33 *(乘)*(乘)20000 / (除) 引物的分子量。引物的分子量可以从合成报告单上获得。如果需要配制成其他浓度,按上述公式换算。 注意:1 OD260= 33 ug/ml. 9.如何计算引物的Tm值? 答:引物设计软件都可以给出Tm,与引物长度、碱基组成、引物使用缓冲的离子强度有关。
长度为25mer以下的引物,Tm计算公式为:Tm = 4℃(G + C)+ 2℃(A + T) 对于更长的寡聚核苷酸,Tm计算公式为: Tm = 81.5 + 16.6 x Log10[Na+] + 0.41 (%GC) – 600/size 公式中,Size = 引物长度。 11.如何溶解引物? 答:干燥后的引物质地非常疏松,开盖前最好离心一下,或管垂直向上在桌面上敲敲,将引物粉末收集到管底。根据计算出的体积加入去离子无菌水或10mM Tris pH7.5缓冲液,室温放置几分钟,振荡助溶,离心将溶液收集到管底。溶解引物用的水一般不要用蒸馏水,因为有些蒸馏水的pH值比较低(pH4-5),引物在这种条件下不稳定。 12.如何保存引物? 答:引物合成后,经过一系列处理和纯化步骤,旋转干燥而成片状物质。引物在溶解前,室温状态下可以长期保存。溶解后的引物-20度可以长期保存。如果对实验的重复性要求较高,合成的OD数较大,建议分装,避免反复冻融。修饰荧光引物需要避光保存。 13.合成的引物5’端是否有磷酸化 答:合成的引物5’为羟基,没有磷酸基团。如果需要您可以用多核苷酸激酶进行5′端磷酸化,或者要求引物合成公司合成时直接在5′或3′端进行磷酸化,需要另外收费。 14.引物片段退火后不能连接到载体上是什么问题? 连接反应需要引物的5’磷酸基团。如果需要将合成的引物退火直接连
Primer 5.0搜索引物: 1.Primer Length我常设置在18-30bp,短了特异性不好,长了没有必要。当然有特殊要求的除外,如加个酶切位点什么的。 2.PCR Product size最好是100-500bp之间,小于100bp的PCR产物琼脂糖凝胶电泳出来,条带很模糊,不好看。至于上限倒也不必要求苛刻。 3.Search parameters还是选Manual吧,Search stringency应选High,GC含量一般是40-60%。其它参数默认就可以了。 4.搜索出来的引物,按Rating排序,逐个送Oligo软件里评估。当然,搜索出的引物,其扩增产物很短,你可以不选择它,或是引物3端≥2个A或T,或引物内部连续的G或C太多,或引物3端≥2个G或C,这样的引物应作为次选,没得选了就选它。对于这样的引物,如果其它各项指标还可以,我喜欢在引物末端去掉一个不满意的或加上一个碱基,看看引物的评估参数有没有变好点。 Oligo 6.0评估引物: 1.在analyze里,Duplex Formation不管是上游引物、下游引物还是上下游引物之间,The most stable 3’-Dimer绝对值应小于4.5kcal/mol, The most stable Dimer overall绝对值一般应小于多少kcal/mol跟PCR退火温度有关,我几次实验感觉在PCR退火温度在65°的时候,The most stable Dimer ove rall 6.7kcal/mol没有问题。 2.Hairpin Formation根据黄金法则 3.False priming sites: Primer的priming efficiency应该是错配地方的4倍左右,更多当然更好。 4.在PCR栏,个人感觉其所显示的optimal annealing temperature数值值得参考。在PCR摸索条件的时候,退火温度为其数值加减2的范围就可以了。 5.Internal stability很重要:我们希望引物的内部稳定性是中间高、两边低的弧形,最起码保证3端不要过于稳定。下图1引物3端过于稳定,很容易导致不适当扩增。△G参照黄金法则,这其实很好理解:把一滴水放到大海里,这滴水就会不停的扩散分布,扩散的越厉害越稳定,所以△G绝对值越大结构越稳定。 最后说一句,敢于尝试就会成功。 第二贴 --科室工作很多,小医生了,没有办法,所以肯怕不能满足很多战友的要求(qq聊或帮助设计),在此表示抱歉。就楼上的问题我试着回答一下,不一定正确,供参考吧。 --1、两个评价系统不一样,个人感觉oligo评价引物好点,primer出来的引物,我一般按效率排序,再结合退火温度和引物长度,选择引物到oligo测试。这是初步的选择,其实引物到了oligo里,退火温度也不一样。 --2、3端的二聚体应该避免,这个要看你的退火温度决定,一个50°的退火温度肯定和65°对二聚体的影响不一样了,一般来讲尽量控制在-4.5kcal/mol以下(个人观点,很多东西真得还是需要自己摸索)。 --3、个人感觉3端有A无A影响不大,3端有T的没有经验。有T是不是一定不行,个人感觉不见得。软件是评估,法则也不是没有例外,不是1+1=2那么确定。 --4、错配和二聚体谁轻谁重,个人觉得“到致命的程度”谁都重要,我也说不好。我设计的时候,尽量两个都不得罪。 --5、GC含量并非不重要,它直接影响引物各端稳定性,3端来两个G或C,稳定性就上去了,粘在模板上很牢。所以我设计的时候,尽量避免这样的情况出现。 谈一下我学这个引物设计的过程吧:
利用IN T ERN ET设计PCR引物举例 周咏东 华西医科大学附属第一医院眼科(610041) 国际互联网上信息资源十分丰富,给人们的生活、学习和工作带来了极大的方便。过去,需要设计PCR引物时,研究人员需要查阅大量文献,有时因无法查到原文,或无相关报道,会使研究工作一开始就不能顺利开展。笔者在科研中发现,有许多科研人员未能掌握I NT ERN ET上有关引物设计的共享资源,故结合实际应用经验予以介绍。使你的科研工作如虎添翼。 IN T ER NET上设计引物,分为两个步骤: 1 检索待扩增基因的DN A序列 首先接入IN T ERN ET,然后键入网址:w w w.ncbi.nlm. nih.g ov便进入了美国国家医学图书馆的生物技术信息中心的主页。在“Sear ch”右边的检索框内选择“G enBank”,然后在“fo r”右边的框内键入你检索的基因序列名,如“human Bcl-2cDN A sequence”,点击“Go”,检索就开始了。 出现的下一网页是“Cur rent Q uery”即告诉你检索出相关文献的数目。如你对结果不满意,该网页下半部分有“A dd T er m(s)to Q uer y”和“M o dify Cur rent Q uer y”两栏供你重新检索;如你对检检索结果满意,即可点击“Retr iev e XX”(X X 为查出的文献数)。接着即显示了刚才调出的文献名。你可选择一篇最符合的,然后将“Display”键右边的框内选择为FA ST A r epor t”(这是下一步设计引物所规定的),点击“D is-play”健,你要的序列就显示出来了。 最后,将这段序列全选,在“编辑”栏中点击“复制”,将此窗口最小化,重开窗口,进入下一步骤。 2 引物的设计 在新开的窗口中,健入网址w ww.g eno me.w https://www.doczj.com/doc/1c5272244.html, 你即进入了“Whitehead Instit ut e for Bio medical R eser ch/ M IT Cent er for Genome Resear ch”的主页。在主页中先找到“G enome Center Softw ar e”标题,在其中的标题为“Ex peri-mental W eb-based Softw ar e”中,点击“W WW.P rimer P ick-ing(Pr imer3)”,此项,我们将用此网上软件设计引物。 网页上显示为“P r imer3o ld V ersio n”及“Click Her e T o T r y N ex t Ver sio n”,这两个版本大同小异,随便用哪一个。关键是在“Paste sour ce sequence belo w”文字下方的大空框内,粘贴上第一步查出的那段序列。然后根据自己的要求,对列出的各项引物设计指标作相应变动,否则为默认。确认指标设定完毕后,点击粘贴序列框下方的“Pick Pr imer s”键,你即得到了所需的引物,显示在“P rimer3O utput”网页上,共有5对,你可按需任选其一。 通过上述两个步骤,你如愿以偿,是不是很简便、快捷?快动手试一试吧!。 编辑 陈小娜 2.1.2非标准数字影像设备上网直接接入模块 该模块用于解决一部分带有数字网络接口,但又仅符合生产厂家内部标准的设备上网问题。以往,生产数字影像设备的众多厂家由于没有统一的上网标准,使医院相当一部分数字影像设备难以上网,因此,该模块须克服许多困难和问题,在实践中逐渐地、局部性地予以实现和完善。 2.1.3 非标准数字影像设备上网间接接入模块 此模块专用于对医院过去引进的,生产厂商根本就没提供上网接口的一部分早期非标准数字影像设备,通过对影像重新A/D的方法让它们间接上网。 2.2 数字影像会诊中心模块 建立医院数字影像局域网与PA CS系统,其核心意义在于能够方便地汇集各种各类检查的数字影像,提供给专家进行综合会诊。因此,在PA CS局域网基础上,建立硬件设施过硬、图像显示清晰、显示技术优良,能同时方便地显示各种数字诊断影像的数字化影像会诊中心,在很大程度上将代表整个项目的临床诊断价值与水平。2.3 数字影像局域网网络工程模块 网络工程模块包括:网络服务器、数据存储与管理软件系统模式、网络布局与工作站分布、网络构架与布线等等。此部分模块归属于计算机Intr anet信息网络工程范畴,对当前医院影像局域网In-tr anet的软硬件性能指标及设备系统的可扩展性具有决定性的作用。 2.4 各工程模块的信息流与局域网系统P ACS软件模块 如果说硬件是骨架,软件就是血液。建立了良好的硬件平台以后,必须要建立合理的网络信息流向和配以优良的PA CS 软件系统,才能保证整个系统能够正常运行。因此,这是建立数字影像会诊中心时,必须很好建立的又一个重要模块。 总之,医院建立数字影像局域网,并逐步过度到最终院际广域网连接已是二十一世纪医院计算机网络发展的必然趋势。我们必须紧跟这一时代发展潮流,扎扎实实从手上工作做起,理清各个系统模块关系,做好各方面技术储备,为医院建立数字影像局域网及P A CS系统作好准备。 编辑 杨立新 142?医学信息2001年3月第14卷第3期 Internet应用●
引物设计的11条黄金法则
PCR引物设计的11条黄金法则 1.引物最好在模板cDNA的保守区内设计。DNA序列的保守区是通过物种间相似序列的比较确定的。在NCBI上搜索不同物种的同一基因,通过序列分析软件(比如DNAman)比对(Alignment),各基因相同的序列就是该基因的保守区。 2.引物长度一般在15~30碱基之间。 引物长度(primerlength)常用的是18-27bp,但不应大于38,因为过长会导致其延伸温度大于74℃,不适于TaqDNA聚合酶进行反应。 3.引物GC含量在40%~60%之间,Tm值最好接近72℃。 GC含量(composition)过高或过低都不利于引发反应。上下游引物的GC含量不能相差太大。另外,上下游引物的Tm值(meltingtemperature)是寡核苷酸的解链温度,即在一定盐浓度条件下,50%寡核苷酸双链解链的温度。有效启动温度,一般高于Tm值
5~10℃。若按公式Tm=4(G+C)+2(A+T)估计引物的Tm值,则有效引物的Tm为55~80℃,其Tm值最好接近72℃以使复性条件最佳。 4.引物3′端要避开密码子的第3位。 如扩增编码区域,引物3′端不要终止于密码子的第3位,因密码子的第3位易发生简并,会影响扩增的特异性与效率。 5.引物3′端不能选择A,最好选择T。 引物3′端错配时,不同碱基引发效率存在着很大的差异,当末位的碱基为A时,即使在错配的情况下,也能有引发链的合成,而当末位链为T 时,错配的引发效率大大降低,G、C错配的引发效率介于A、T之间,所以3′端最好选择T。 6.碱基要随机分布。 引物序列在模板内应当没有相似性较高,尤其是3’端相似性较高的序列,否则容易导致错误引发(Falsepriming)。降低引物与模板相似性的一种方法是,引物中四种碱基的分布最好是随机的,不要有聚嘌呤或聚嘧啶的存在。尤其3′端
引物设计和Primer-BLAST的应用 Lv Peng 2015.11.18
CONTENT 1.PCR-引物设计目的 2.引物设计原则 3.设计引物软件 4.在线设计工具 5.probeBase 简介
1.1PCR(Polymerase Chain Reaction) 聚合酶链式反应 1971 Khorana 提出设想 1985 Kary Mullis 发明了PCR 1986年5月 Mullis在冷 泉港实验室 做专题报告 冷泉港实验室(The Cold Spring Harbor Laboratory,缩写CSHL),又译为科尔德斯普林实验室。
几不同的PCR技术 1.扩增已知序列两侧DNA的PCR:反向PCR(Inverse PCR,IPCR)、锚定PCR(anchored PCR)、RACE(Rapid Amplification of cDNA Ends)、连接介导的PCR(ligation-mediated PCR,LM-PCR); 2.检测有限量稀有靶序列,即一对引物扩增产物不足以以通过凝胶电泳观察到的时:巢式PCR(nested PCR); 3.快速、灵敏、特异而准确定量的PCR:实时荧光定量PCR (real-time quantitative PCR,RQ-PCR)。
特性 优化 碱基组成 (G+C )含量应在40%-60%,4种碱基要分布均匀;长度 一般为18-27个核苷酸长度。上下游引物长度差别不能大于3bp ;重复和自身互补序列 不能有大于3bp 的反向重复序列或自身互补序列存在;上下游引物互补性一个引物的3’末端序列不能结合到另一个引物的任何位点上; 解链温度(Tm ) 两个引物的Tm 值相差不能大于5℃,扩增产物与引物的Tm 值相差不能大于10℃3’末端 引物3’末端碱基尽量为G 或C ,不能使3’末端有NNGC 或NNCG 序列引物序列不要有局部的GC rich 或AT rich (特别是3’端),避开T/C 或A/G 的连续结构 1.2引物设计原则 引物特性及优化设计
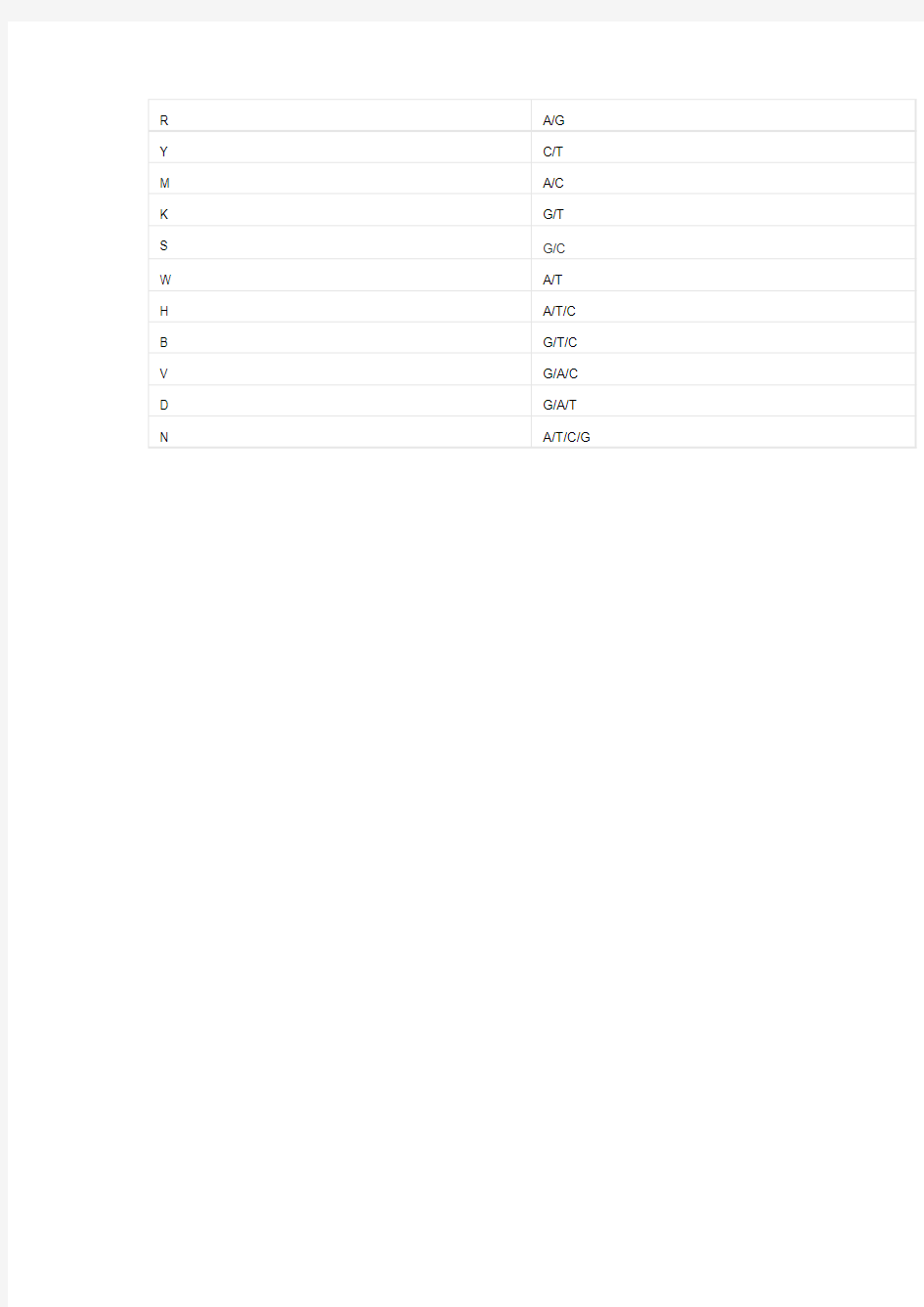
The central role of UDPGDH played in capsule and other polysaccharides synthesis. KPS, capsule polysaccharide; LPS,lipopolysaccharide 简并引物设计方法 (1)利用NCBI搜索不同物种中同一目的基因的蛋白质或cDNA编码的氨基酸序列因为密码子的关系,不同的核苷酸序列可能表达的氨基酸序列是相同的,所以氨基酸序列才是真正保守的。首先利用NCBI的Entrez检索系统,查找到一条相关序列即可。随后利用这一序列使用BLASTP(通过蛋白查蛋白),在整个NR数据库中查找与之相似的氨基酸序列。 (2)对所有的序列进行多序列比对将搜索到的同一基因的不同氨基酸序列进行多序列比对,可选工具有Clustal W/X,也可在线分析。所有序列的共有部分将会显示出来。“*”表示保守,“:”表示次保守。 (3)确定合适的保守区域设计简并引物至少需要上下游各有一个保守区域,且两个保守区域相距50~400个氨基酸残基为宜,使得PCR产物在150~1200bp 之间,最重要的是每一个保守区域至少有6个氨基酸的保守区,因为每条引物至少18bp左右。 若比对结果保守性不是很强很可能找不到6个氨基酸序列的保守区,这时可以根据物种的亲缘关系,选择亲缘关系近的物种进行二次比对,若保守性仍达不到要求,则需进行三次比对,总之,究竟要选多少序列来比对,要根据前一次的结果反复调整。最终目的就是有两个6个氨基酸且两者间距离合适的保守区域。 (4)利用软件设计引物当得到保守区域后,就可以利用专业的软件来设计引物了,其中Primer 5.0 支持简并引物的设计,将参与多序列比对的序列中的任一条导入Primer 5.0 中,将其翻译成核苷酸序列,该序列群可用一条有简并性的核苷酸链来表示(其中R=A/G,Y=C/T,M=A/C,K=G/T,S=C/G,W=A/C/T,B=C/G/T,V=A/C/G,D=A/G/T,N=A/C/G/T,该具有简并性的核苷酸链必然包含上一步中找到的氨基酸保守区域的对应部分,在Primer 5.0 中修改参数,令其在两个距离合适的保守的nt区域内寻找引物对,总之要保证上下游引物都落在该简并链的保守区域内,结果会有数对,分数越高越好。 (5)对引物的修饰若得到的引物为: 5-NAGSGNGCDTTANCABK-3 则简并度=4×2×4×3×4×3×2=2304,很明显该条引物的简并度很高不利于PCR,可以通过次黄嘌呤代替N(因为次黄嘌呤可以很好的和4种碱基配对)和根据物种密码子偏好这两种方法来降低简并度。 这样设计出来的简并引物对,适用于比对的氨基酸序列所属物种及与这些物种分类地位相同的其他物种。 简并引物设计原则
1-2890(引物1) #1: Product of length 640 (rating: 171) Contains region of the molecule from 1 to 640 Tm: 72.1 C TaOpt: 48.8 C GC: 32.3 Sense Primer: CCTGGTTAATCCAAATCAC Similarity: 100.0% Length: 19 Tm: 44.1 C GC: 42.1 dH: -142.0 kcal/mol dS: -372.4 cal/mol dG: -29.2 kcal/mol Antisense Primer: GACAGGCCCTAATTAAGTT Similarity: 100.0% Length: 20 Tm: 45.0 C GC: 42.1 dH: -158.0 kcal/mol dS: -418.4 cal/mol dG: -31.5 kcal/mol Tm Difference: 0.9 GC Difference: 0 #1: Product of length 540 (rating: 171) Contains region of the molecule from 1 to 540 Tm: 72.2 C TaOpt: 49.4 C GC: 33.1 Sense Primer: CCTGGTTAATCCAAATCACT Similarity: 100.0% Length: 20 Tm: 45.8 C GC: 40.0 dH: -149.8 kcal/mol dS: -393.2 cal/mol dG: -30.8 kcal/mol Antisense Primer: ATAAGATTTGAGGTCAGCCA Similarity: 100.0% Length: 20 Tm: 46.4 C GC: 40.0 dH: -147.7 kcal/mol dS: -386.3 cal/mol dG: -30.7 kcal/mol Tm Difference: 0.6 GC Difference: 0.0 1-2890(引物2) #1: Product of length 603 (rating: 171) Contains region of the molecule from 514 to 1116 Tm: 73.9 C TaOpt: 50.3 C GC: 37.0 Sense Primer: TTGAAGATGGCTGACCT Similarity: 100.0% Length: 18 Tm: 42 C GC: 47.1 dH: -129.6 kcal/mol dS: -335.0 cal/mol dG: -27.9 kcal/mol Antisense Primer: GGAGGCCCTTTAACTTAA
引物设计step by step 1、在NCBI上搜索到目的基因,找到该基因的mRNA,在CDS选项中,找到编码区所在位置,在下面的origin中,Copy该编码序列作为软件查询序列的候选对象。 2、用Primer Premier5搜索引物 ①打开Primer Premier5,点击File-New-DNA sequence, 出现输入序列窗口,Copy目的序列在输入框内(选择As),此窗口内,序列也可以直接翻译成蛋白。点击Primer,进入引物窗口。 ②此窗口可以链接到“引物搜索”、“引物编辑”以及“搜索结果”选项,点击Search按钮,进入引物搜索框,选择“PCR primers”,“Pairs”,设定搜索区域和引物长度和产物长度。在Search Parameters里面,可以设定相应参数。一般若无特殊需要,参数选择默认即可,但产物长度可以适当变化,因为100~200bp的产物电泳跑得较散,所以可以选择300~500bp. ③点击OK,软件即开始自动搜索引物,搜索完成后,会自动跳出结果窗口,搜索结果默认按照评分(Rating)排序,点击其中任一个搜索结果,可以在“引物窗口”中,显示出该引物的综合情况,包括上游引物和下游引物的序列和位置,引物的各种信息等。 ④对于引物的序列,可以简单查看一下,避免出现下列情况:3’不要出现连续的3个碱基相连的情况,比如GGG或CCC,否则容易引起错配。此窗口中需要着重查看的包括:Tm 应该在55~70度之间,GC%应该在45%~55%间,上游引物和下游引物的Tm值最好不要相差太多,大概在2度以下较好。该窗口的最下面列出了两条引物的二级结构信息,包括,发卡,二聚体,引物间交叉二聚体和错误引发位置。若按钮显示为红色,表示存在该二级结构,点击该红色按钮,即可看到相应二级结构位置图示。最理想的引物,应该都不存在这些二级结构,即这几个按钮都显示为“None”为好。但有时很难找到各个条件都满足的引物,所以要求可以适当放宽,比如引物存在错配的话,可以就具体情况考察该错配的效率如何,是否会明显影响产物。对于引物具体详细的评价需要借助于Oligo来完成,Oligo自身虽然带有引物搜索功能,但其搜索出的引物质量感觉不如Primer5. ⑤在Primer5窗口中,若觉得某一对引物合适,可以在搜索结果窗口中,点击该引物,然后在菜单栏,选择File-Print-Current pair,使用PDF虚拟打印机,即可转换为Pdf文档,里面有该引物的详细信息。 3、用Oligo验证评估引物 ①在Oligo软件界面,File菜单下,选择Open,定位到目的cDNA序列(在primer中,该序列已经被保存为Seq文件),会跳出来两个窗口,分别为Internal Stability(Delta G)窗口和Tm窗口。在Tm窗口中,点击最左下角的按钮,会出来引物定位对话框,输入候选的上游引物序列位置(Primer5已经给出)即可,而引物长度可以通过点击Change-Current oligo length来改变。定位后,点击Tm窗口的Upper按钮,确定上游引物,同样方法定位下游引物位置,点击Lower按钮,确定下游引物。引物确定后,即可以充分利用Analyze 菜单中各种强大的引物分析功能了。
PCR引物设计与分析 摘要:本文简单的介绍了PCR技术以及PCR引物设计原则和技巧,并以一段序列为例,介绍两种引物设计软件的使用方法。一般性引物自动搜索可采用“Premier Primer 5”软件,而引物的评价则可采用“Oligo 6”软件。 关键词:PCR;引物设计;软件; 1PCR 聚合酶链式反应(英文全称:Polymerase Chain Reaction),简称PCR。聚合酶链式反应(PCR)是体外酶促合成特异DNA片段的一种方法,由高温变性、低温退火(复性)及适温延伸等几步反应组成一个周期,循环进行,使目的DNA 得以迅速扩增,具有特异性强、灵敏度高、操作简便、省时等特点。它不仅可用于基因分离、克隆和核酸序列分析等基础研究,还可用于疾病的诊断或任何有DNA,RNA的地方。 PCR又称无细胞分子克隆或特异性DNA序列体外引物定向酶促扩增技术。 类似于DNA的天然复制过程,其特异性依赖于与靶序列两端互补的寡核苷酸引物。PCR由变性--退火(复性)--延伸三个基本反应步骤构成:①模板DNA的变性:模板DNA经加热至90~95℃一定时间后,使模板DNA双链或经PCR扩增形成的双链DNA解离,使之成为单链,以便它与引物结合,为下轮反应作准备;②模板DNA与引物的退火(复性):模板DNA经加热变性成单链后,温度降至55~60℃,引物与模板DNA单链的互补序列配对结合;③引物的延伸:DNA模板--引物结合物在DNA聚合酶的作用下,于70~75℃,以dNTP为反应原料,靶序列为模板,按碱基配对与半保留复制原理,合成一条新的与模板DNA链互补的半保留复制链重复循环变性--退火--延伸三过程,就可获得更多的“半保留复制链”,而且这种新链又可成为下次循环的模板。每完成一个循环需2~4分钟,2~3小时就能将待扩目的基因扩增放大几百万倍。 参加PCR反应的物质主要有五种即引物、酶、dNTP、模板和Mg2+。引物是PCR特异性反应的关键,PCR 产物的特异性取决于引物与模板DNA互补的程度。理论上,只要知道任何一段模板DNA序列,就能按其设计互补的寡核苷酸链做引物,利用PCR就可将模板DNA在体外大量扩增。 2引物设计原则及注意
本文叙述了一种用于甲基化分析的探针法定量PCR的引物和探针设计方法,目前用于甲基化检测的引物探针设计工具非常多,都有使用成功的案例,经过初步多方尝试,本文中叙述的为本人认为较为靠谱的方法。Oligo7的优势在于专业,参数详尽且可自由设置,模块化设计,学会后使用便利。专业的活就是要专业的用专业的工具干。
首先是进行序列转换,有较多的在线工具和联机软件都可实现,这里使用https://www.doczj.com/doc/1c5272244.html,/methprimer/,较为简单直观。
直接将目标序列放入如上图的编辑框中,此也可直接用于相关引物的设计,不过本人没使用过,因为不能设计探针。submit后就有转化后的序列信息,如下图: 以上详细标记了CpG位置和非CpG位置的C,可直接复制到Word标注使用,下面就可以使用Oligo7利用上边的序列设计引物和探针了,如果是设计非甲基化引物探针,则使用原始序列。
关于引物和探针的一些主要参数,主要参考invtrogen的建议: Primer设计的基本原则: a)引物长度一般在18-35mer。 b)G-C含量控制在40-60%左右。 c)避免近3’端有酶切位点或发夹结构。 d)如果可能避免在3’端最后5个碱基有2个以上的G或C。 e)如果可能避免在3’端最后1个碱基为A。 f)避免连续相同碱基的出现,特别是要避免GGGG或更多G出现。 g)退火温度Tm控制在58-60C左右。 h)如果是设计点突变引物,突变点应尽可能在引物的中间。 T aqMan 探针设计的基本原则: a)T aqMan 探针位置尽可能靠近扩增引物(扩增产物50-150bp),但不能与引物重叠。 b)长度一般为18-40mer 。 c)G-C含量控制在40-80%左右。 d)避免连续相同碱基的出现,特别是要避免GGGG或更多G出现。 e)在引物的5’端避免使用G。 f)选用比较多的碱基C。 g)退火温度Tm控制在68-70℃左右。 另:目标变异碱基最好在3’末端或3’末端-1位置,保证扩增特异性,对于甲基化,则最好是C。
LAMP原理及引物设计与实例 .LAMP引物的设计 LAMP引物的设计主要是针对靶基因的六个不同的区域,基于靶基因3' 端的F3c、F2c和Flc区以及5' 端的Bl、B2和B3区等6个不同的位点设计4种引物。 FIP(Forward Inner Primer):上游内部引物,由F2区和F1C区域组成,F2区与靶基因3’端的F2c区域互补,F1C区与靶基因5' 端的Flc区域序列相同。 F3引物:上游外部引物(Forward Outer Primer),由F3区组成,并与靶基因的F3c区域互补。 BIP引物:下游内部引物(Backward Inner Primer ),由B1C和B2区域组成,B2区与靶基因3' 端的B2c区域互补,B1C域与靶基因5' 端的Blc区域序列相同. B3引物:下游外部引物(Backward Outer Primer ),由B3区域组成,和靶基因的B3c区域互补。 2.扩增原理 60-65℃是双链DNA复性及延伸的中间温度,DNA在65℃左右处于动态平衡状态。因此,DNA在此温度下合成是可能的。利用4种特异引物依靠一种高活性链置换DNA聚合酶。使得链置换DNA合成在不停地自我循环。扩增分两个阶段。 第1阶段为起始阶段,任何一个引物向双链DNA的互补部位进行碱基配对延伸时,另一条链就会解离,变成单链。上游内部引物FIP的F2序列首先与模板F2c结合(如图B所示),在链置换型DNA聚合酶的作用下向前延伸启动链置换合成。外部引物F3与模板F3c结合并延伸,置换出完整的FIP连接的互补单链(如图C所示)。FIP上的F1c与此单链上的Fl 为互补结构。自我碱基配对形成环状结构(如图C所示)。以此链为模板。下游引物BIP与B3先后启动类似于FIP和F3的合成,形成哑铃状结构的单链。迅速以3' 末端的Fl区段为起点。以自身为模板,进行DNA合成延伸形成茎环状结构。该结构是LAMP基因扩增循环的起始结构。 第2阶段是扩增循环阶段。以茎环状结构为模板,FIP与茎环的F2c区结合。开始链置换合成,解离出的单链核酸上也会形成环状结构。迅速以3’末端的B1区段为起点,以自身为模板。进行DNA合成延伸及链置换.形成长短不一的2条新茎环状结构的DNA,BIP引物上的B2与其杂交。启动新一轮扩增。且产物DNA长度增加一倍。在反应体系中添加2条环状引物LF和LB,它们也分别与茎环状结构结合启动链置换合成,周而复始。扩增的最后产物是具有不同个数茎环结构、不同长度DNA的混合物。且产物DNA为扩增靶序列的交替反向重复序列。 https://www.doczj.com/doc/1c5272244.html,MP的特点 LAMP与以往的核酸扩增方法相比具有如下优点: (1)操作简单,LAMP核酸扩增是在等温条件下进行,对于中小医院只需要水浴锅即可,产
引物设计的原理与方法 This model paper was revised by the Standardization Office on December 10, 2020
PCR引物设计的原理及方法 阎振鑫S111666(四川大学生命科学学院细胞生物学成都 610014) 摘要:自20世纪后期发展了PCR技术以来,PCR已经改变了整个生物学研究的进程。而PCR反应的第一步就是设计引物,引物设计的好坏直接关系到PCR的成败。PCR引物设计有许多的原则必须要遵循:引物与引物之间避免形成稳定的二聚体或发夹结构,引物与模板的序列要紧密互补。引物不能在模板的非目的位点引发DNA聚合反应等。另外,引物的设计方法也越来越多,出现了许多专门的设计软件和网站,如:PrimerPremier5.0等。 关键词:PCR 引物原理方法 NCBI PrimerPremier5.0 PCR primer design principle and method YanZhenxin (sichuan Univercity, Life science college cell biology chengdu 610014 ) Abstract: When PCR technology was find, PCR has changed all of the program in research of biology. The design of primer is the frist step of PCR. It is relation to the fate of PCR. There are some principals must be obey: dipolymer and hairpin structure must be avoid between different primers. The DNA polymerization reaction should not be triggered at the wrong site. Therefore, there are more and more methods of design primer, include the professional softwares and professional web site. Key word: PCR primer principle NCBI PrimerPremier5.0 聚合酶链式反应(Polymerase chain reaction。PCR)是20世纪后期发展起来的 一种体外扩增特异DNA片断的技术。具有快速、简便及高度敏感等优点,能极大地缩短目的基因扩增时间[1]。因此,其一直是生物学者们致力于构建cDNA文库、基因克隆以及表达调控研究的必要前提和基础[2]。PCR的第一步就是引物设计。引物设计的好坏,直接影响了PCR的结果,因此这一步很关键。成功的PCR反应既要高效,又要特异性扩增产物,因此对引物设计提出了较高的要求。引物设计需要注意的地方很多,在大多数情况下,我们都是在知道已知模板序列时进行PCR扩增的。在某些情况比如构建文库的时候也会在不知道模板序列的情况下进行设计。这个时候随机核苷酸序列
20个测序常见的问题 1.为什么需要新鲜的菌液? 首先,新鲜的菌液易于培养,可以获得更多的DNA,同时最大限度地保证菌种的纯度。2.如何提供菌液? 如果您提供新鲜菌液,用封口膜封口以免泄漏;也可以将培养好的4~5ml菌液沉淀下来,倒去上清以方便邮寄。同时邮寄时最好用盒子以免邮寄过程中压破。 3.如何制作穿刺菌? 用灭菌过1.5ml或2ml离心管加入LB琼脂(7g/L)斜面凝固,用接种针挑取分散良好的单菌落穿过琼脂直达管底,不完全盖紧管盖适当温度培养过夜,然后盖紧盖子加封口膜,室温或4度保存。 4.PCR产物直接测序有什么要求? (1)扩增产物必须特异性扩增,条带单一。如果扩增产物中存在非特异性扩增产物,一般难以得到好的测序结果; (2)必须进行胶回收纯化; (3)DNA纯度在1.6—2.0之间,浓度50ng/ul以上。 5.为什么PCR产物直接测序必须进行Agarose胶纯化? 如果不进行胶纯化而直接用试剂盒回收,经常会导致测序出现双峰甚至乱峰,这主要是非特异性扩增产物或者原来的PCR引物去除不干净所导致。大多所谓的PCR“纯化试剂盒”实际上只是回收产物而不能起到纯化的作用的。对于非特异性扩增产物肯定无法去除,而且通常他们不能够完全去除所有的PCR引物,这会造成残留的引物在测序反应过程中参与反应而导致乱峰。 6.如何进行PCR产物纯化? PCR产物首先必须用Agarose胶电泳,将特异扩增的条带切割下,然后纯化。使用凝胶回收试剂盒回收,产物用ddH2O溶解。 7.PCR产物直接测序的好处? (1) PCR产物直接测序可以反映模板的真实情况; (2) 省去克隆的实验费用和时间; (3) PCR产物测序正确的片段进行下一步克隆实验使结果更有保障; (4) 混合模板进行PCR的产物直接测序可以发现其中的点突变。 8.对用于测序的质粒DNA的要求有哪些? 对测序模板DNA的一般要求:(1)DNA纯度要求高,1.6—2.0之间,不能有混合模板,也不能含有RNA,染色体DNA,蛋白质等;(2)溶于ddH2O中,溶液不能含杂质,如盐类,或EDTA等螯合剂,将干扰测序反应正常进行。 9.如何鉴定质粒DNA浓度和纯度? 我们使用水平琼脂糖凝胶电泳,并在胶中加入0.5ug/ml的EB(电泳缓冲液中不必加E,加一个已知浓度的标准样品。电泳结束以后在紫外灯下比较亮度,判断浓度和纯度。此方法可以更直接、准确地判断样品中是否含有染色体DNA、RNA等,也可以鉴别抽提的质粒DNA 的不同构型。 质粒DNA的3种构型是指在抽提质粒DNA过程中,由于各种原因的影响,使得超螺旋的共价闭合环状结构的质粒(SC)的一条链断裂,变成开环状(OC)分子,如果两条链发生断裂,就变成为线状(L)分子。这3种分子有不同的迁移率,通常,超螺旋型(SC)迁移速度最快,其次为线状(L)分子,最慢为开环状(OC)分子。使用紫外分光光度计检测,或者用溴乙锭-标准浓度DNA比较法只能检测抽提到的产物中的浓度,甚至由于抽提的质粒DNA中含有RNA、蛋白质、染色体DNA等因素的干扰,浓度检测的数值也是没有多少意义的。
PCR引物的设计原则: ①引物应用核酸系列保守区内设计并具有特异性。 ②产物不能形成二级结构。 ③引物长度一般在15~30碱基之间。 ④G+C含量在40%~60%之间。 ⑤碱基要随机分布。 ⑥引物自身不能有连续4个碱基的互补。 ⑦引物之间不能有连续4个碱基的互补。 ⑧引物5′端可以修饰。 ⑨引物3′端不可修饰。 ⑩引物3′端要避开密码子的第3位。 1.引物的特异性 引物与非特异扩增序列的同源性不要超过70%或有连续8个互补碱基同源。 2.避开产物的二级结构区 某些引物无效的主要原因是引物重复区DNA二级结构的影响,选择扩增片段时最好避开二级结构区域。用有关计算机软件可以预测估计mRNA 的稳定二级结构,有助于选择模板。实验表明,待扩区域自由能(△G°)小于58.6lkJ/mol时,扩增往往不能成功。若不能避开这一区域时,用7-deaza-2′-脱氧GTP取代dGTP对扩增的成功是有帮助的。 3.长度
寡核苷酸引物长度为15~30bp,一般为18~27mer。引物的有效长度:Ln=2(G+C)+(A+T),Ln值不能大于38,因为>38时,最适延伸温度会超过Taq DNA聚合酶的最适温度(74℃),不能保证产物的特异性。 4.G+C含量 G+C含量一般为40%~60%。其Tm值是寡核苷酸的解链温度,即在一定盐浓度条件下,50%寡核苷酸双链解链的温度,有效启动温度,一般高于Tm值5~10℃。若按公式Tm=4(G+C)+2(A+T)估计引物的Tm 值,则有效引物的Tm为55~80℃,其Tm值最好接近72℃以使复性条件最佳。上下游引物的GC含量不能相差太大。 5.碱基随机分布 引物中四种碱基的分布最好是随机的,不要有聚嘌呤或聚嘧啶的存在。尤其3′端不应超过3个连续的G或C,因这样会使引物在G+C富集序列区错误引发。 6.引物自身 引物自身不应存在互补序列,否则引物自身会折叠成发夹状结构引物本身复性。这种二级结构会因空间位阻而影响引物与模板的复性结合。若用人工判断,引物自身连续互补碱基不能大于3bp。 7.引物之间 两引物之间不应具有互补性,尤应避免3′端的互补重叠以防引物二聚体的形成。一对引物间不应多于4个连续碱基的同源性或互补性。
Rosetta系列的表达菌株可以提供T7 RNA聚合酶,它能表达PET系列载体上的外源基因。。。pGEX系列载体上的外源基因不需要T7 RNA聚合酶,普通的大肠杆菌经IPTG诱导即可表达 Tac启动子是一组由Lac和trp启动子人工构建的杂合启动子,受Lac阻遏蛋白的负调节,它的启动能力比Lac和trp都强。其中Tac 1是由Trp启动子的-35区加上一个合成的46 bp DNA片段(包括Pribnow 盒)和Lac操纵基因构成,Tac 12是由Trp的启动子-35区和Lac 启动子的-10区,加上Lac操纵子中的操纵基因部分和SD序列融合而成 蛋白标签:
pGEX4T1载体基本信息 出品公司: GE 别名: pGEX-4T-1, pGEX4T1, pGEX 4T 1 质粒类型: 大肠杆菌蛋白表达载体 表达水平: 高拷贝 启动子: Tac 克隆方法: 多克隆位点,限制性内切酶 载体大小: 4969 bp 5' 测序引物及序列: pGEX5': GGGCTGGCAAGCCACGTTTGGTG 3' 测序引物及序列: pGEX3': CCGGGAGCTGCATGTGTCAGAGG 载体标签: N-GST 载体抗性: Ampicillin 氨苄 备注: 复制子是pMB1 产品目录号: 27-4580-01 稳定性: 瞬时表达 Transient 组成型: 诱导表达 病毒/非病毒: 非病毒 pGEX4T1载体质粒图谱和多克隆位点信息 原核生物DNA复制起始点,是DNA链上独特的具有起始DNA复制功能的碱基序列。大肠杆菌的复制起
pGEX4T1载体简介 pGEX4T1载体序列 LOCUS pGEX-4T-1 4969 bp DNA circular SYN DEFINITION pGEX-4T-1 ACCESSION KEYWORDS SOURCE ORGANISM other sequences; artificial sequences; vectors. COMMENT This file is created by Vector NTI https://www.doczj.com/doc/1c5272244.html,/ COMMENT VNTAUTHORNAME|https://www.doczj.com/doc/1c5272244.html,| FEATURES Location/Qualifiers source 1..4969 /organism="pGEX-4T-1" /mol_type="other DNA" promoter 184..212 /label="tac_promoter" misc_feature 224..246 /label="M13_pUC_rev_primer" gene 258..977 /label="GST (variant)" /gene="GST (variant)" CDS 258..977 /label="ORF frame 3"