real time PCR 数据分析excel模板
- 格式:xls
- 大小:16.00 KB
- 文档页数:2
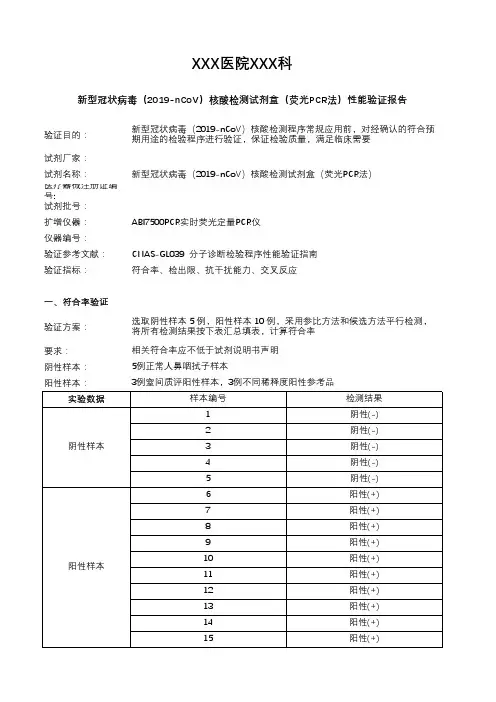
验证目的:试剂厂家:试剂名称:医疗器械注册证编号:试剂批号:扩增仪器:仪器编号:验证参考文献:验证指标:一、符合率验证验证方案:要求:阴性样本:阳性样本:XXX医院XXX科新型冠状病毒(2019-nCoV)核酸检测试剂盒(荧光PCR法)性能验证报告新型冠状病毒(2019-nCoV)核酸检测程序常规应用前,对经确认的符合预期用途的检验程序进行验证,保证检验质量,满足临床需要CNAS-GL039 分子诊断检验程序性能验证指南符合率、检出限、抗干扰能力、交叉反应选取阴性样本 5 例,阳性样本 10 例,采用参比方法和候选方法平行检测,将所有检测结果按下表汇总填表,计算符合率相关符合率应不低于试剂说明书声明新型冠状病毒(2019-nCoV)核酸检测试剂盒(荧光PCR法)ABI7500PCR实时荧光定量PCR仪5例正常人鼻咽拭子样本3例窒间质评阳性样本,3例不同稀释度阳性参考品二、检出限验证验证方案:要求:标准物质浓度:稀释液:稀释浓度:厂家声明检出限:三、抗干扰能力验证验证方案:要求:干扰物质1:加入量:生理盐水200copies/mL5次结果均为阳性,检出率100%使用定值标准物质(如:国际参考品、国家参考品、厂家参考品),梯度稀释至厂家声明的检出限浓度,重复测定 5 次根据临床需求及实际情况,选取相应干扰物质,适量加入到待测弱阳性样本中,按常规样本操作流程,重复测定 3 次3次检测结果均为阳性,验证通过人全血0.5ml干扰物质2:加入量:四、交叉反应验证验证方案:要求:加入病原体1:加入病原体2:验证日期:操作者:复核者原始数据保存位置:原始数据文件名:原始数据附图:XX鼻腔喷雾剂0.2ml乙型流感病毒取一定浓度与新型冠状病毒(2019-nCoV)核酸可能存在交叉反应的病原体加入样本保存液或经确认为阴性的样本中,按常规样本操作流程,重复测定3次3次检测结果均为阴性,验证通过SARS 冠状病毒。

real time PCR 数据分析real-timepcr数据分析无论所使用的real-timepcr是何种型号,正确的数据分析对于获得有效的实验结果都是至关重要的。
这里介绍有关real-timepcr数据分析的知识。
在探讨基本分析过程之前,先了解如何设计一个不好的实验。
如果你就是自己设计的引物和探针,那有利于下一步的工作。
但是在有些情况下,人们采用出版发行文献上的序列可以更便利。
忘记,即便就是出版物提供更多的序列也无法确保可以获得优化的实验结果。
而且排印错误的可能性也须要考量在内。
所以步入实验室之前采用blast对全部序列展开核实保证他们就是恰当的。
下订单前先检察引物和探针的序列和tm值就是实验设计的基本建议。
标准曲线是判断实验质量的重要手段。
使用一个已知的模板,pcr产物,合成的寡核苷酸或转录的rna做个标准曲线能够确定pcr的效率,敏感性,动态范围和其他的参数。
建立标准曲线时使用od260的模板样本。
模板的总量以dna分子的数量来描述,把质量转化为dna含量的公式如下:(质量(克)*阿伏伽德罗常数)每个碱基的平均值质量*模板的长度。
例如,合成70-mer的单链dna,样本质量为0.8*10?-11gm。
代入公式得:(0.8*10?-11*6.023*10?23molecules/mole)330gm/mole/base*70base。
如果使用双链的模板,则碱基的平均质量为660gm/mole/base。
标准曲线采用的模板含量从1*10?7已经开始已连续吸收7次每次吸收10倍,最终获得10个模板拷贝。
这样的浓度有利于获得最低的δrn和最高的ct。
用excel画曲线时以模板数量的对数值为x,ct(cyclethreshold)值y轴。
标准曲线的计算公式如下:y=mx+b。
y就是ct,m是斜率,x=log10templateamount,b=y-intercept。
用斜率排序出来实验效率efficiency【10?(-1/斜率)】-1。

Real-time qPCR 检测操作手册1/ 6一、实验概述Real-time Quantitative PCR Detecting System ,即实时荧光定量核酸扩增检测系统,也叫实时定量基因扩增荧光检测系统,简称qPCR 。
是一种在PCR 反应体系中加入荧光基团,利用荧光信号积累实时监测整个PCR 进程,最后通过特定数学原理对未知模板进行定量分析的方法,实现了PCR 从定性到定量的飞跃。
二、实验流程三、实验材料: 1.主要试剂试剂名称 试剂来源 Cat.No. Trizol 上海普飞 3101-100 M-MLV promega M1705 dNTPs promega U1240 oligo dT 上海生工 B0205 Bulge-LoopTM miRNA广州锐博 详见实验报告 qPCR Primer Set Rnase Inhibitor promega N2115 Primer(R&F) 上海生工 详见实验报告 SYBR Master MixtureTAKARADRR041B2/ 6样本收集Trizol 法抽取RNARNA 反转录获取cDNAReal-time qPCR2.主要器材器材名称来源Cat.NoNanodrop 分光光度计Thermo 2000/2000C稳压电泳仪上海天能EPS-600超细匀浆机FLUKO公司F6/10Real time PCR 仪器Agilent公司MX3000p反转录耗材Axygen四、实验步骤:1.总RNA 抽提(1)收取样品,Trizol 裂解。
细胞样品:收集细胞(6 孔板 80%细胞密度),2000 rpm 离心5min,去上清,细胞沉淀中加入1mL Trizol,充分混匀后室温静置 5 min,然后转移至新的 1.5 mL EP 管中;组织样品:将待研磨的组织样品从液氮或者-80℃冰箱中取出,无菌刀片于干冰上将组织样品切割成约 3 mm×3 mm×3 mm 大小,置于装有 1 mL Trizol 裂解液的 1.5mL EP 管中。

实时荧光定量PCR的数据分析方法
作者:易健明, 屈武斌, 张成岗, YI Jian-Ming, QU Wu-Bin, ZHANG Cheng-Gang
作者单位:易健明,张成岗,YI Jian-Ming,ZHANG Cheng-Gang(军事医学科学院放射与辐射医学研究所,蛋白质组学国家重点实验室,全军军事认知与心理卫生研究中心,北京100850;安徽医科大学研究生院,安徽合肥230032)
, 屈武斌,QU Wu-Bin(军事医学科学院放射与辐射医学研究所,蛋白质组学国家重点实验室,全军军事认知
与心理卫生研究中心,北京100850)
刊名:
生物技术通讯
英文刊名:Letters in Biotechnology
年,卷(期):2015,26(1)
引用本文格式:易健明.屈武斌.张成岗.YI Jian-Ming.QU Wu-Bin.ZHANG Cheng-Gang实时荧光定量PCR的数据分析方法[期刊论文]-生物技术通讯 2015(1)。


LightCycler ® 480 系统快速操作指南1 一.配制好反应体系,封好膜。
接通LC480与电脑电源,电脑帐号operator ,密码LC480,点击LC480软件,登录帐号user ,密码Master1。
二.开始实验:点击,在列表中选择对应的程序,如H1N1或HBV ,点击窗口右下角的,点击软件界面右下角的,输入实验文件名点击窗口右下角的开始实验。
三.编辑子集:点击subset editor ,点击左下角的,按ctrl 键的同时鼠标选择本次实验的孔,最后点击应用。
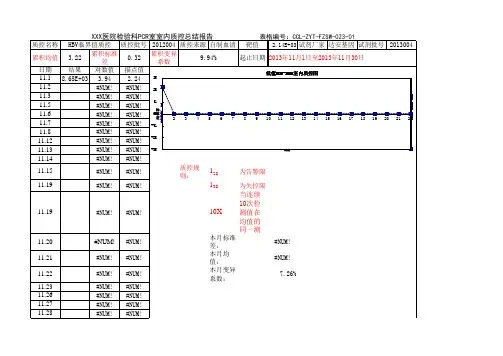
注意,一次实验可以同时运行相同扩增参数的多个实验(如H1N1、HIV 与HCV ),那么可以分别设置多个子集设置样品:先在subset 中选择本次实验的子集,点击左上角的,样品输入样品名,再选择标准品类型和浓度或者阳性对照/阴性对照四.数据分析:实验运行完成后进入,选择Abs Quant/Fit Ppoint ,在窗口中的Subset (第二行)中选择本次实验的子集,点击软件界面中上方的,在Noise Band 中调节noise band 高度,使之处于对数增长期并高于所有噪音信号,如右图。
再点击进入,点击软件界面中间偏右的再点击软件界面左下角的值或浓度值等以及平均值标准误等信息,该部分可以鼠标拖动滑块或鼠标拖曳、鼠标点击软件右上角的为本次分析命名,如输入H1N1或其它名称五.报告输出打印:点击软件右侧的保存当前实验的分析结果,再点击软件左侧的,点击软件中部左侧的detailed 选择需要输出的分析结果,如H1N1, 分析中建议选择results 或standard curve ,点击软件界面左下方的,生成PDF 报告,如装了打印机,直接点击软件中间上方的,或者点击,选择pdf 文件保存位置后点击save 保存。

RealTimePCR结果分析与实验设计做Real Time PCR的大体有四个目的。
1. 检测某个基因表达水平的上调或下调。
2. 检测siRNA靶基因的下调量。
3. 观测某个基因在一系列细胞中的表达模式。
4. 别出心裁的实验设计。
四种实验对应的数据处理方法是不同的。
但原理是一样的,即都基于这个公式:Ct = -k * log2(X0) + b (1)其中k=1/log2(1+E),其中E代表PCR的扩增效率,如每个循环扩增的原DNA的90%,则E=0.9。
b是个无关紧要的常量,除非是绝对定量rt PCR,否则是用不到b的。
所谓的2 delta方法是这样的:如有基因A,内参基因C,样品甲和乙。
欲知基因A在甲中的表达量相对于乙是上调还是下调,上调或下调了多少。
应得到四个Ct值:Ct(甲A),Ct(甲C),Ct(乙A)和Ct (乙C)。
ΔCt(甲)=Ct(甲C)-Ct(甲A)=k*log2[X(甲A)/X(甲C)]ΔCt(乙)=Ct(乙C)-Ct(乙A)=k*log2[X(乙A)/X(乙C)]ΔΔCt=ΔCt(甲)-ΔCt(乙)=k*log2[A在甲中的表达量/A在乙中的表达量]如此则:R=A在甲中的表达量/A在乙中的表达量=2^(ΔΔCt/k)这样一来,只要知道k值就可以了。
k=1/log2(1+E),如果扩增效率是100%,则E=1,k=1。
否则只要做个浓度梯度,就可把它计算出来。
1. 做siRNA时,R<1,(1-R)*100%即siRNA下调靶基因的量。
2. 表达模式分析只要一个delta就行了。
3. 一通百通,无论怎么弄,都有解决办法。
实验设计时,每板重复三次,做三个板,是为了消除PCR本身和样品本身的误差,是必不可少的。
如同微阵列一样,三次是最少次数。
取平均值可以,中数也可以。
无论如何,实验设计一定要严谨。



一般来讲,进行real-time qPCR MasterMix都是2×的浓缩液,只需要加入模板和引物就可以。
由于real-time qPCR灵敏度高,所以每个样品至少要做3个平行孔,以防在后面的数据分析中,由于Ct相差较多或者SD太大,无法进行统计分析。
通常来讲,反应体系的引物终浓度为100-400mM;模板如果是总RNA一般是10ng-500,如果cDNA,通常情况下是1ul或者1ul的10倍稀释液,要根据目的基因的表达丰度进行调整。
当然这些都是经验值,在操作过程中,还需要根据所用MasterMix,模板和引物的不同进行优化,达到一个最佳反应体系。
在反应体系配置过程中,有下面几点需要注意:1. MasterMix不要反复冻融,如果经常使用,最好溶解后放在4度。
2. 更多的配制Mix进行,减少加样误差。
最好能在冰上操作。
一般来讲,进行real-time qPCR MasterMix都是2×的浓缩液,只需要加入模板和引物就可以。
由于real-time qPCR灵敏度高,所以每个样品至少要做3个平行孔,以防在后面的数据分析中,由于Ct相差较多或者SD太大,无法进行统计分析。
通常来讲,反应体系的引物终浓度为100-400mM;模板如果是总RNA一般是10ng-500,如果cDNA,通常情况下是1ul或者1ul的10倍稀释液,要根据目的基因的表达丰度进行调整。
当然这些都是经验值,在操作过程中,还需要根据所用MasterMix,模板和引物的不同进行优化,达到一个最佳反应体系。
在反应体系配置过程中,有下面几点需要注意:1. MasterMix不要反复冻融,如果经常使用,最好溶解后放在4度。
3. 每管或每孔都要换新枪头!不要连续用同一个枪头加样!4. 所有成分加完后,离心去除气泡。
5. 每个样品至少3个平行孔。
参比或者校正染料(reference dye,passive dye)常用的是ROXTM(现在已经是ABI的注册商标了!)或者其他染料,只要不影响检测PCR产物的荧光值就可以。
一.配制好反应体系,封好膜。
接通LC480与电脑电源,电脑帐号operator,密码LC480,点击LC480软件,登录帐号user,密码Master1。
二.开始实验:点击,在列表中选择对应的程序,如H1N1或HBV,点击窗口右下角的,点击软件界面右下角的,输入实验文件名点击窗口右下角的开始实验。
三.编辑子集:点击subset editor,点击左下角的,按ctrl键的同时鼠标选择本次实验的孔,最后点击应用。
注意,一次实验可以同时运行相同扩增参数的多个实验(如H1N1、HIV与HCV),那么可以分别设置多个子集设置样品:先在subset中选择本次实验的子集,点击左上角的,样品输入样品名,再选择标准品类型和浓度或者阳性对照/阴性对照四.数据分析:实验运行完成后进入,选择Abs Quant/Fit Ppoint,在窗口中的Subset(第二行)中选择本次实验的子集,点击软件界面中上方的,在Noise Band中调节noise band高度,使之处于对数增长期并高于所有噪音信号,如右图。
再点击进入,点击软件界面中间偏右的自动设置阈值或手工调节阈值(鼠标左键按住拖动或在输入阈值大小),再点击软件界面左下角的,软件左下方出现每个样品的Ct值或浓度值等以及平均值标准误等信息,该部分可以鼠标拖动滑块或鼠标拖曳、鼠标点击软件右上角的为本次分析命名,如输入H1N1或其它名称五.报告输出打印:点击软件右侧的保存当前实验的分析结果,再点击软件左侧的,选择需要输出的分析结果,如H1N1,点击软件中部左侧的detailed分析中建议选择results或standard curve,点击软件界面左下方的,生成PDF报告,如装了打印机,直接点击软件中间上方的,或者点击,选择pdf文件保存位置后点击save保存。
荧光定量PCR实验及数据分析一、概述荧光定量PCR(Quantitative Realtime PCR,简称qPCR)是一种结合了PCR技术的高灵敏度和荧光探针技术的实时定量特性的分子生物学分析方法。
该方法通过实时监测PCR反应过程中荧光信号的变化,对模板DNA或RNA的初始浓度进行定量分析。
荧光定量PCR技术在基因表达研究、病原体检测、基因突变分析以及药物疗效评估等领域具有广泛的应用价值。
在荧光定量PCR实验中,通常使用特异性引物和荧光探针来识别并扩增目标序列。
荧光探针的设计是关键步骤之一,它必须能够与目标序列特异性结合并在PCR过程中产生可检测的荧光信号。
实验过程中还需严格控制反应条件,包括温度、时间、引物和探针的浓度等,以确保实验的准确性和可重复性。
数据分析是荧光定量PCR实验不可或缺的一部分。
通过对实验数据的收集、整理和分析,可以获取目标序列的初始浓度信息,进而对实验结果进行解读和评估。
数据分析方法包括相对定量和绝对定量两种,前者通过比较不同样本间目标序列的相对表达量来评估差异,后者则通过标准曲线法或质粒拷贝数法等方法来确定目标序列的绝对浓度。
荧光定量PCR技术是一种高效、灵敏且特异的分子生物学分析方法,对于研究基因表达、病原体检测等领域具有重要意义。
通过不断优化实验操作和数据分析方法,可以进一步提高荧光定量PCR实验的准确性和可靠性,为科学研究和临床实践提供有力支持。
1. 荧光定量PCR技术的概述荧光定量PCR技术,是一种基于DNA聚合酶链式反应的分子生物学技术,它通过引入荧光标记,实时监测PCR过程中目标DNA片段的扩增情况,从而实现对特定基因拷贝数的精确量化。
该技术结合了PCR的高效扩增能力与荧光信号的灵敏检测,使得微量DNA分子的检测成为可能,并在遗传学、分子生物学、医学诊断等领域中发挥着重要作用。
荧光定量PCR技术主要依赖于特异性引物和探针的设计,使得PCR扩增过程具有高度的特异性。
谈谈如何分析RealtimePCR的结果一直瞎白话,作为一名知识分子,好歹也得干点儿正经事,既然做科研,就得严谨,就得实事求是。
言归正传,写点儿如何分析Realtime PCR结果的体会。
为什么想这个呢?我们实验室现在做这个的越来越多,但是看到他们做Presentation时的结果分析的统计图就知道,他们没踏实搞清楚如何去分析结果,就上去糊弄老板了,我曾严肃的当场指出过,但是师姐气得直瞪眼,丫就不明白我是真为她好,我也就懒得跟她纠缠了。
但是作为当代,我国,世界级著名学府(校领导自称的),一流专业(就是没什么人报名,一半是调剂的),牛B 教授(这个是实话)的不用心学生,我还是忍不住要说一说。
关于Realtime PCR的所有情况的结果如何分析,我不都懂,也不想都懂,但是我需要的关于基因表达差异的2-ΔΔCt分析方法,这一点我还是比较清楚的。
原理:其实就是这个公式:X n=X o× (1+E x)nX n,表示n个循环后的分子数;X o,表示初始模板分子数;E x,表示分子扩增效率;n,表示循环数。
首先我们要明白,Realtime PCR的终极目的是要检测其初始模板的量的相对或绝对量(这就是半定量或定量Realtime PCR的区别)。
还有一个最为重要的概念就是Ct值,是指目标扩增产物达到所设定的阈值时所经历循环数。
再回到刚才的公式中,我们可以得到下面的式子:X t = X o× (1+E x)Ctx = K x (1)X t,表示到达阈值时的分子数;K x,是个常数,因为这是人为设定的。
其他同前式。
那么,同理,内参也有一个这样的式子:R t = R o× (1+E x)CtR = K R (2)把结果均一化,所谓均一化就是通过内参的比较,去除加入模板时的人为差异,即(1)除以(2)得出,X t / R t = X o / R o × (1+E x)CtX- CtR = K x / K R进一步推出我们关注的初始模板均一化值:X o / R o = K x / K R × (1+E x)-(CtX - CtR),令CtX - CtR =ΔCt,因此,可得X o / R o = K x / K R × (1+E x)ΔCt(3)至此,我们得到了组内的模板的均一化相对或绝对值,下一步是组间的差异比较。
由于Real-time qPCR 的众多优点,现在已是生命科学领域的一项常规技术。
越来越多的研究文章中涉及RT-PCR 的实验,也基本上被real-time qPCR 所代替。
由于real-time aPCR 输出的数据不同于常规的PCR 电泳检测,不少没有做过real-time qPCR 的研究者往往感到高深莫测,不知从何入手;甚至一些做过次实验的研究者也会对数据处理分析感到迷惑,不知所措。
本文就从real-time qPCR 的发展史说起,包括real-time qPCR 的原理,实验设计,实际操作,数据分析,常见问题解答五个方面,手把手教你从各个方面了解real-time qPCR,彻底的从菜鸟到高手!一、Real-time qPCR 发展史Real-time qPCR 就是在PCR 扩增过程中,通过荧光信号,对PCR 进程进行实时检测。
由于在PCR 扩增的指数时期,模板的Ct 值和该模板的起始拷贝数存在线性关系,所以成为定量的依据。
由于常规的PCR 的缺点,real-time qPCR 由于其操作简便,灵敏度高,重复性好等优点发展非常迅速。
现在已经涉及到生命科学研究的各个领域,比如基因的差异表达分析,SNP 检测,等位基因的检测,药物开辟,临床诊断,转基因研究等。
在Real-time qPCR 技术的发展过程中,定量PCR 仪的发展起了至关重要的作用。
1995 年,美国PE 公司(已经并入Invitrogen 公司)成功研制了Taqman 技术,1996 年推出了首台荧光定量PCR 检测系统,通过检测每一个循环的荧光强度,通过Ct 值进行数据分析。
从而荧光定量PCR 获得广泛应用。
现在的定量PCR 仪有ABI7000、7300、7500,7700、7900HT、StepOnePlusTM、StepOneTM、PRISM@StepOneTM 系列;BIO-RAD 的CFX96、iCycler iQ5@、MyiQ@、MJ Research Chromo4TM Opticon 系列;Stratagene MxTM 系列;Roche LightCycler@ 系列;Eppendorf Masercycler@;Corbett Rotor-GeneTM;Cepheid SmartCycler@和BIOER 的LineGene 系列。
罗氏r e a l t i m e P C R操作指南(总2页)本页仅作为文档封面,使用时可以删除This document is for reference only-rar21year.March一.配制好反应体系,封好膜。
接通LC480与电脑电源,电脑帐号operator,密码LC480,点击LC480软件,登录帐号user,密码Master1。
二.开始实验:点击,在列表中选择对应的程序,如H1N1或HBV,点击窗口右下角的,点击软件界面右下角的,输入实验文件名点击窗口右下角的开始实验。
三.编辑子集:点击subset editor,点击左下角的,按ctrl键的同时鼠标选择本次实验的孔,最后点击应用。
注意,一次实验可以同时运行相同扩增参数的多个实验(如H1N1、HIV与HCV),那么可以分别设置多个子集设置样品:先在subset中选择本次实验的子集,点击左上角的,样品输入样品名,再选择标准品类型和浓度或者阳性对照/阴性对照四.数据分析:实验运行完成后进入,选择Abs Quant/FitPpoint,在窗口中的Subset(第二行)中选择本次实验的子集,点击软件界面中上方的,在Noise Band中调节noise band高度,使之处于对数增长期并高于所有噪音信号,如右图。
再点击进入,点击软件界面中间偏右的自动设置阈值或手工调节阈值(鼠标左键按住拖动或在输入阈值大小),再点击软件界面左下角的,软件左下方出现每个样品的Ct值或浓度值等以及平均值标准误等信息,该部分可以鼠标拖动滑块或鼠标拖曳、鼠标点击软件右上角的为本次分析命名,如输入H1N1或其它名称五.报告输出打印:点击软件右侧的保存当前实验的分析结果,再点击软件左侧的,点击软件中部左侧的detailed选择需要输出的分析结果,如H1N1,分析中建议选择results或standard curve,点击软件界面左下方的,生成PDF报告,如装了打印机,直接点击软件中间上方的,或者点击,选择pdf文件保存位置后点击save 保存。