利用STRING 数据库进行Cytoscape蛋白互作网络绘制步骤详解
- 格式:docx
- 大小:1.96 MB
- 文档页数:23

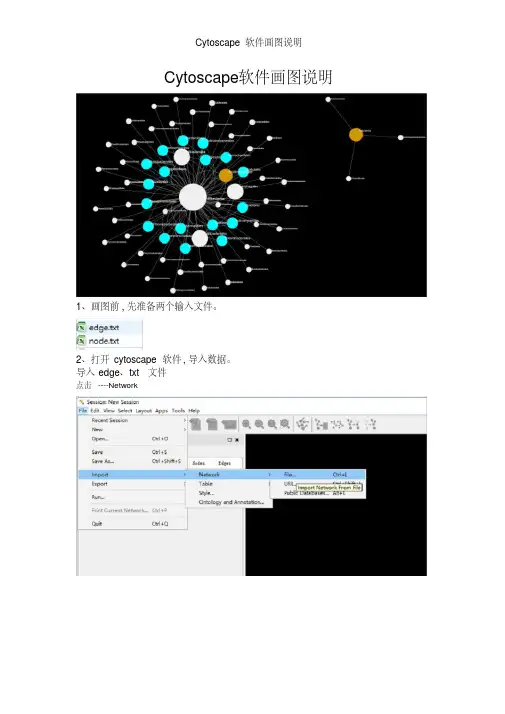
Cytoscape软件画图说明1、画图前,先准备两个输入文件。
2、打开cytoscape软件,导入数据。
导入edge.txt文件点击File ----Import ----Network点击ok得到原始图形节点1,文件中第一列节点2,文件中第2列连接类型,文件中第3列点击layout ----Apply Preferred Out 改变图形排列方式此处可以用鼠标在画布中拖动图形到合适的位置。
改变画布背景。
点击左侧Contro Panel ----Style---network ---Backgroud paint设置节点之间连线的宽度和颜色Contro Panel ----Style---Edge颜色宽度导入node.txt个文件点击File ----Import ----Table设置节点图形属性Contro Panel ----Style---Node1、node大小通过设置Height和Width来控制大小2、Node性状通过设置Shape来控制3、Node填充色通过设置Fill Color来控制4、Node标签的设置点击Properties -- Label Position点击Label Position来设置标签位置。
这里是一个示范操作,要细致调整标签位置还是要设置Column和Mapping Type两个参数。
设置完成之后图形调节节点之间的距离点击layout ----Scale鼠标拖动最后,画图导出成pdf文件点击File ----Export----Network View as Graphics精品文档。
11欢迎下载欢迎您的下载,资料仅供参考!致力为企业和个人提供合同协议,策划案计划书,学习资料等等打造全网一站式需求。


STRING数据库的蛋白质相互作用(PPI)网络分析STRING(Search Tool for the Retrieval of InteractingGenes/Proteins)数据库是一个用于存储蛋白质相互作用(PPI)信息的在线资源。
PPI网络分析是研究蛋白质之间相互作用的一种方法。
通过分析PPI网络,研究者可以了解蛋白质的功能和作用机制,揭示生物系统的复杂性。
在本篇文章中,我们将探讨PPI网络分析的意义、方法和应用。
PPI网络分析的意义在于帮助我们理解蛋白质的功能和相互作用。
蛋白质是细胞中最重要的功能分子之一,它们通过相互作用形成复杂的网络结构,从而参与调控细胞的生理和病理过程。
通过构建PPI网络并进行分析,我们可以了解蛋白质在细胞中的相互关系,进而找到调控的关键因子。
PPI网络的构建通常基于实验数据或计算机预测。
实验方法包括酵母双杂交、共免疫沉淀和质谱分析等。
这些实验技术可以检测到蛋白质之间的物理相互作用。
计算机预测方法则基于已知蛋白质结构和序列信息,通过算法判断蛋白质之间是否可能相互作用。
STRING数据库整合了多种实验和计算方法生成的PPI数据,提供了更全面的PPI网络信息。
PPI网络分析通常包括网络图的构建和网络特性的分析。
网络图由节点和边组成,其中节点代表蛋白质,边表示蛋白质之间的相互作用。
网络构建可以基于已知的实验数据或计算机预测结果。
网络特性分析包括节点度数、网络连通性、模块化等指标的计算。
这些指标可以帮助我们了解网络的结构和特点。
PPI网络分析的应用非常广泛。
首先,它可以帮助我们预测蛋白质的功能。
蛋白质的功能通常与其相互作用的伙伴密切相关。
通过分析PPI网络,我们可以推断一个未知蛋白质的功能,并为后续实验提供指导。
其次,PPI网络分析还可以帮助我们识别关键的调控通路和靶点。
在许多疾病中,蛋白质相互作用的异常可能是病理过程的关键因素。
通过分析PPI网络,我们可以找到与疾病相关的节点和模块,并设计针对性的治疗策略。

Cytoscape软件画图说明
1、画图前,先准备两个输入文件。
2、打开cytoscape软件,导入数据。
导入edge、txt文件
点击----Network
节点1,文件中第一列
节点2,文件中
第2列
连接类型,文件
中第3列
点击ok得到原始图形
点击layout ----Apply Preferred Out 改变图形排列方式
此处可以用鼠标在画布中拖动图形到合适的位置。
改变画布背景。
点击左侧Contro Panel ----Style---network ---Backgroud paint
设置节点之间连线的宽度与颜色Contro Panel ----Style---Edge
导入node、txt个文件点击----Table 宽度颜色
设置节点图形属性
Contro Panel ----Style---Node
1、node大小
通过设置Height与Width来控制大小
2、Node性状
通过设置Shape来控制
3、Node填充色
通过设置Fill Color来控制
4、Node标签的设置
点击Properties -- Label Position
点击Label Position来设置标签位置。
这里就是一个示范操作,要细致调整标签位置还就是要设置Column与Mapping Type两个参数。
设置完成之后图形
调节节点之间的距离
点击layout ----Scale
鼠标拖动
最后,画图导出成pdf文件点击View as Graphics。
STRING网站Cytoscape软件制作精美蛋白互作网络图(PPI)之前小编为大家推送了利用DAVID网站进行差异基因的GO和KEGG分析,链接:DAVID&Metascape:专注于基因功能注释和富集通路分析的网站。
而基因功能注释后就可以寻找蛋白表达之间的关系了,在生信分析中,常常会使用STRING网站+Cytoscape软件来制作蛋白互作网络图(PPI)。
今天小编奉上一部PPI制作教程,让我们一起细细咀嚼吧!首先我们进入STRING网站的官网(网址为/),界面很简单明了,左侧一栏是网站的输入方式,右侧一栏是我们的蛋白输入框。
通常我们在做PPI时选择输入方式是“Multiple proteins”,即多个蛋白的输入。
选择好蛋白的输入方式后,输入一列差异基因,STRING网站对基因的限制条件是不超过2000个基因,格式也是每行一个基因名(或者说是蛋白名字吧,总之就是差异基因所表达的蛋白)。
简而言之,这个页面上的操作的步骤是先选择“Multiple protenins”,然后输入基因名,最后选择“Homo sapiens”。
然后点击“SEARCH”选项,千万别停下来,继续点击“CONTINUE”选项。
这样就做好了一张PPI图,但是看上去还是比较杂乱的,因此我们需要通过Cytoscape软件对PPI图进一步做美化处理。
下拉当前页面,可以看到一行菜单,Legend菜单里面是关于网络图中Nodes和Edges的注释,了解一下。
Settings菜单功能比较重要,比如我们对Edges的选择,“confidence”是通过线条的粗细来反映蛋白之间相互作用的强弱。
如果我们制作的网络图比较分散,我们可以通过设置“minimum required interaction score”将conbined_score调高来调整PPI,使图形看上去更紧密。
如果我们觉得网络图上的蛋白数量较少,我们可以通过设置蛋白数量的上限,比如我们将其设置为“no more than 50 interactors”,看下效果。

STRING蛋白网络分析操作流程蛋白网络分析是一种系统生物学方法,用于研究蛋白质相互作用网络。
它可以帮助科学家解析蛋白质相互作用、信号传导和代谢途径,从而更好地理解细胞的功能和调控机制。
以下是一个关于蛋白网络分析的操作流程,包括数据准备、网络构建和分析、结果解释和验证等几个主要步骤。
1.数据准备:首先,需要收集和整理相关的蛋白质相互作用数据。
这些数据可以来自公开数据库,如STRING(Search Tool for the Retrieval of Interacting Genes/Proteins)、BioGRID、IntAct等。
可以根据研究对象的物种和特定的研究领域选择相应的数据库。
确保所选择的数据可靠性和准确性。
2.数据预处理:在进行网络构建之前,需要对原始数据进行预处理,以去除噪音和无效的信息。
这通常包括去除重复的相互作用、修复格式错误和标准化数据格式等。
此外,还可以根据研究的目的进行数据筛选和过滤,如选择特定阈值的相互作用等。
3.网络构建和分析:接下来,使用预处理后的数据构建蛋白网络。
最常用的方法是基于相互作用的网络,其中蛋白质与其他蛋白质之间的相互作用表示为网络中的节点和边。
可以使用网络分析软件,如Cytoscape等,进行网络可视化和分析。
可以计算网络的节点度、聚集系数、连通性等基本拓扑特征,并进行模块和子网络的发现,以揭示蛋白质相互作用网络的组织结构和功能模块。
4.结果解释:对于网络中的重要节点和模块,可以使用注释数据库(如Gene Ontology)来解释其生物学功能和关联的代谢途径。
这可以帮助科学家理解蛋白质网络的功能和调控机制,从而提出相关的假设和研究问题。
5.结果验证:为了验证通过蛋白网络分析得到的结果,可以采用多种实验技术,如免疫共沉淀、基因敲除、RNA干扰等。
这些实验可以用来验证网络预测的相互作用和功能,进一步验证和揭示蛋白质网络的生物学意义和调控机制。
需要注意的是,蛋白网络分析是一个复杂的过程,需要综合运用多种工具和技术,并结合实验验证来解析蛋白质相互作用网络。

利用生物大数据技术开展蛋白质互作网络分析的方法与技巧引言:在生物学研究中,蛋白质互作网络是理解细胞内分子之间相互作用的关键工具。
生物大数据技术的出现为我们提供了大规模蛋白质互作网络数据和分析工具。
本文将介绍利用生物大数据技术开展蛋白质互作网络分析的方法与技巧。
一、数据获取蛋白质互作网络分析的第一步是获取蛋白质互作网络的数据。
目前,公共数据库如STRING、BioGRID、HPRD、MINT等提供了大量的蛋白质互作数据。
研究人员可以通过访问这些数据库,根据自己的研究兴趣和需要获取相关数据。
此外,也可以利用生物大数据技术从大规模基因组学研究中获得蛋白质互作网络数据。
二、数据预处理蛋白质互作网络数据的预处理对于后续的分析至关重要。
预处理的目的是去除噪声和无关信息,提高分析的准确性和可靠性。
预处理的步骤包括数据清洗、数据归一化和数据筛选等。
数据清洗主要是去除无效数据和错误数据,如重复数据、无效交互等。
数据归一化用于消除数据集之间的差异,将数据调整到统一的尺度上。
数据筛选是根据研究目的和假设筛选出与研究有关的蛋白质互作数据。
三、网络分析方法基于生物大数据的蛋白质互作网络分析有多种方法可选,本文将介绍常用的两种方法:网络可视化和网络分析工具。
1. 网络可视化网络可视化是将蛋白质互作网络数据以图形方式呈现,帮助研究人员直观地理解蛋白质之间的相互关系。
常用的网络可视化工具有Cytoscape、Gephi等。
这些工具可以根据互作关系绘制节点和边,节点代表蛋白质,边代表蛋白质之间的互作关系。
网络可视化不仅可以展示整体的蛋白质互作网络,还可以根据需求进行扩展和收缩,分析特定的互作子网络。
2. 网络分析工具网络分析工具是用于发现蛋白质互作网络的特征和模式的工具。
常用的网络分析工具有CentiScaPe、MAGI和STRING等。
这些工具可以对蛋白质互作网络进行拓扑结构分析、模块检测、节点中心性分析等。
通过网络分析工具,可以发现互作网络中的关键节点、功能模块和互作模式,为深入理解蛋白质相互作用提供重要线索。

STRING蛋白网络分析操作流程蛋白网络分析是一种重要的生物信息学方法,用于研究蛋白质相互作用网络和蛋白质功能与调控机制。
下面是一个关于蛋白网络分析的操作流程,涵盖了数据收集、网络构建、网络分析和结果解释四个主要步骤。
1.数据收集:蛋白网络分析所需的数据主要包括蛋白质相互作用实验数据、蛋白质序列和结构信息、蛋白表达数据等。
这些数据可以从公共数据库(如STRING、BioGRID等)、文献以及实验室内部实验获得。
2.网络构建:使用蛋白质相互作用数据构建蛋白网络。
可以使用多种网络构建方法,包括物理相互作用实验数据、预测相互作用数据和文献挖掘方法。
常用的方法有基于实验数据的亲和屏蔽分析、酵母双杂交实验等。
3.网络分析:运用网络分析方法揭示蛋白网络的拓扑结构、模块化特征和功能调控机制。
网络拓扑参数如节点度、聚集系数、介数中心性等可以用来描述网络的特征。
模块检测方法可以识别网络中的功能模块,揭示蛋白相互作用网络的组织与功能。
通过富集分析,可以对网络中的功能组件进行功能注释和生物学解释。
4.结果解释:通过对网络分析结果进行解释来深入理解蛋白网络的生物学意义。
可以使用基因本体论(Gene Ontology)和通路富集分析等方法,对蛋白网络中的蛋白功能进行聚类和分析,揭示蛋白质功能的调控机制。
此外,还可以通过叠加其他类型的数据,如表达数据、遗传突变等,进一步解释蛋白网络的生物学含义。
蛋白网络分析操作流程中的主要注意事项包括数据的质量评估、网络构建方法的选择、网络分析算法的列队以及结果的解释解释。
此外,还应该关注蛋白网络分析方法的局限性和未知的潜在问题。
总结以上所述,蛋白网络分析是一个复杂而有用的研究领域。
通过对蛋白质相互作用网络的构建和分析,我们可以揭示蛋白质功能和调控机制,为疾病治疗和生物学研究提供有价值的信息。
但需要注意的是,在进行蛋白网络分析时,我们应该结合多种数据和方法,谨慎地进行结果解释,以获得可靠的研究结论。
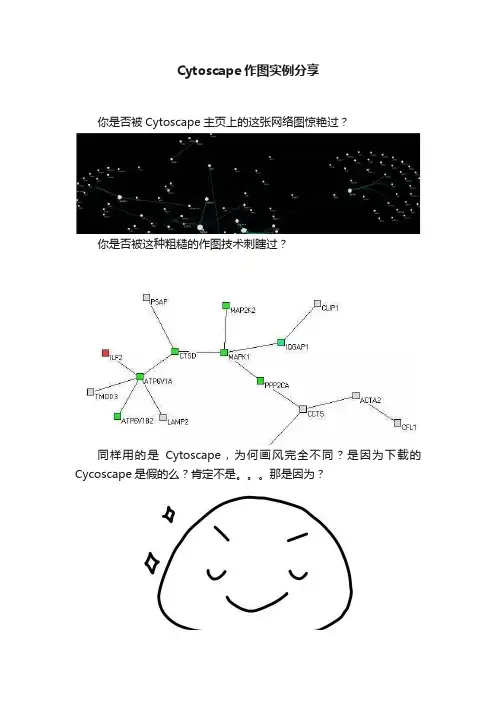
Cytoscape作图实例分享
你是否被Cytoscape主页上的这张网络图惊艳过?
你是否被这种粗糙的作图技术刺瞎过?
同样用的是Cytoscape,为何画风完全不同?是因为下载的Cycoscape是假的么?肯定不是。
那是因为?
因为没有看过本宫写的帖子!
既然封面上的这张图这么养眼,今天就实操演示一下它的画法,希望大家能够学会这套“眼保健操”!
封面这张图是一张蛋白质互作网络图,所以本宫找了一组基因去STRING上分析了一下,然后导出了互作关系的txt文件(上传到网盘中了,链接见文章底部)。
导入txt文件,把孤立节点Edit>>Cut掉
调整一下整体网络风格(选了一种最接近于目标网络图的风格,在此基础上进行微调)
这里顺便介绍一下,Border选项可以在节点边缘添加边框效果
接下来我们调整一下布局样式(选择这个样式是因为边缘的节点样式比较接近于封面图片),中间的节点比较密集,我们稍微拖一下,让它们分散点
封面图片的节点大小是根据节点的Degree来调整的,所以我们先用Centiscape这个插件算出节点的Degree
然后根据节点的Dgree调整节点大小
渐变选项(Continuous Mapping)大致调成10-50
下面开始调整Edge,先把Edge变成曲线,这里用默认选项即可
接下来调整Edge的样式,先调颜色(调了半天,选了这么个颜色~~只能说差不多吧)
后来用PS的拾色器取了看了一下参数
然后调整透明度,根据String数据库分析的combine_score渐变,参数见下图
再调整一下Edge的粗细,还是根据combine_score渐变。

蛋白质交互作用网络的建模和分析在生物大分子之间,互相作用的蛋白质数不胜数。
了解蛋白质之间的相互作用是研究许多生物学问题的关键,例如细胞周期、信号转导、代谢调节等。
因此,构建蛋白质相互作用网络模型并对其进行分析,以深入挖掘蛋白质间相互作用的规律和机制,已成为生物学、生物信息学等学科领域的热点和难点之一。
一、蛋白质相互作用网络模型的构建蛋白质相互作用网络模型是指将不同蛋白质之间的相互作用关系用图形表示出来。
这种图形通常被称为无向图或有向图,其中节点代表蛋白质,连接代表蛋白质之间的相互作用关系。
常用的构建蛋白质相互作用网络的方法有两种,其一是基于实验数据的方法,即利用实验手段如酵母双杂交、蛋白质质谱法、共沉淀等,获得蛋白质相互作用信息,并根据这些信息构建蛋白质相互作用网络模型;其二是基于预测的方法,即利用蛋白质序列、结构和功能等信息,通过计算或机器学习方法预测蛋白质之间的相互作用关系,进而构建蛋白质相互作用网络模型。
二、蛋白质相互作用网络模型的分析1、网络特征分析网络特征分析是对蛋白质网络模型进行全面描述和定量分析,以揭示网络的基本特征和规律。
蛋白质相互作用网络模型通常具有无标度网络、小世界网络等特征,这些特征能够用来解释蛋白质功能复杂性和调控机制,同时也为网络的仿真模拟提供了数学基础。
2、模块化分析模块化分析是将网络分为一系列具有内部紧密联系且与整个网络相对松散联系的互补部分。
蛋白质相互作用网络往往可以分为多个模块,这些模块反映了蛋白质相互作用的基本规律和生物学功能。
3、网络动力学模拟网络动力学模拟是对蛋白质网络模型动态行为进行定量分析的一种方法。
该方法不仅能够描述网络节点和连接的变化过程,而且能够通过模拟来预测网络的演化趋势和稳态结构,为揭示蛋白质网络的分子机制提供了重要途径。
三、蛋白质相互作用网络模型的应用1、蛋白质功能鉴定蛋白质相互作用网络模型可为蛋白质功能鉴定提供帮助,该模型可通过预测和分析节点的邻近节点来推断该节点与特定生物学功能相关的蛋白质。
蛋白质交互作用网络的构建和分析蛋白质是生命中核心的分子之一,它们在细胞内发挥着各种重要的生物学功能。
蛋白质通常通过与其他蛋白质相互作用,形成各种生物学体系。
因此,构建蛋白质交互作用网络是了解生物学过程中极其重要的一环。
本文将介绍蛋白质交互作用网络的构建和分析。
1. 蛋白质交互作用网络的构建蛋白质交互作用网络可以通过多种方法来构建,例如蛋白质芯片、酵母双杂交等。
其中最常用的方法是基于大规模蛋白质质谱测定技术的方法,这种方法无需进行基因重组和体内表达,只需要将蛋白质纯化后进行质谱测定即可。
在蛋白质质谱测定的过程中,会测定蛋白质的相互作用情况。
通过统计蛋白质之间的相互作用,构建出蛋白质交互作用网络。
这种构建方法可以高通量地测定大量蛋白质之间的相互作用情况。
2. 蛋白质交互作用网络的性质蛋白质交互作用网络是一种无标度网络。
无标度网络是指网络中少数节点之间有着极其密集的连接,而大多数节点之间的连接则比较稀疏。
蛋白质交互作用网络中也存在着这种情况,少数蛋白质之间的连接极其密集,而大多数蛋白质之间的连接则比较稀疏。
此外,蛋白质交互作用网络还具有模块化的特点。
模块是指网络中密集连接的节点群,这些节点在结构上具有相似的特征。
在蛋白质交互作用网络中,模块一般对应于特定的生物学功能或者生化途径,例如代谢途径、信号转导等等。
3. 蛋白质交互作用网络的分析蛋白质交互作用网络的分析可以揭示出各种有关生物学过程的重要信息。
下面将介绍常用的几种分析方法。
第一种分析方法是中心度分析。
中心度是指一个节点在网络中的重要程度。
在蛋白质交互作用网络中,节点的中心度与其在生物学过程中的重要性有关,例如高中心度的节点可能在生物过程中扮演着关键性的角色。
第二种分析方法是模块分析。
模块是网络中密集连接的节点群,这些节点在结构上具有相似的特征。
模块分析可以揭示出网络中不同模块对应的生物学过程或者生化途径。
第三种分析方法是网络可视化。
网络可视化可以将复杂的网络结构以可视化的方式呈现出来,帮助人们理解网络的结构和相互关系,进而揭示出网络中的生物学意义。
蛋白质互作网络的构建与分析一、引言蛋白质互作网络是指在细胞中相互作用的蛋白质之间形成的一种复杂网络结构,它是细胞内信号传导、代谢调节、基因表达等方面的重要调控机制。
通过对蛋白质互作网络的构建和分析可以揭示细胞内分子间的相互关系、生命现象的本质以及疾病的发生机制,具有很高的科学研究价值。
二、蛋白质互作网络的构建蛋白质互作网络的构建过程主要包括以下几个方面。
1. 数据收集数据的收集是构建蛋白质互作网络的关键步骤。
目前,常用的数据来源有生物实验数据和数据库数据。
生物实验数据可以通过蛋白质共沉淀、Y2H、GST-pull down等技术获得,但由于实验操作的复杂性和成本的高昂,通常是通过公开的实验数据集进行分析。
数据库数据主要包括基因组、转录组、蛋白质组等数据,这些数据是通过文献分析、生物实验等手段获得的。
2. 数据清洗由于大部分实验数据的准确度和可靠性不高,因此需要进行数据清洗,剔除不可靠的数据,确保后续分析的准确性和可靠性。
数据清洗通常会去掉具有异常值或错误值的数据,去除表达值过低或过高的基因,剔除具有非特异性的基因等。
3. 特征提取特征提取是将数据转化为能够用于构建蛋白质互作网络的特征向量的过程。
通常采用的方法是从每个蛋白质的表达谱数据中提取局部和全局特征,例如局部特征可以是蛋白质结构域的相关信息,全局特征可以是表示蛋白质发生生物学过程的表达谱数据。
4. 网络构建网络构建是将数据转化为网络结构的过程。
常用的方法有无监督方法和监督方法。
无监督方法主要包括互相关联方法、K-means聚类方法等,监督方法主要包括神经网络、贝叶斯网络、SVM等。
三、蛋白质互作网络的分析蛋白质互作网络的分析主要包括以下几个方面。
1. 网络特征分析网络特征分析是评估蛋白质互作网络复杂性的过程。
通过对网络中节点数、边数、平均度数、聚集系数等多维度指标的计算和分析,可以评估不同网络之间的拓扑性质,从而深刻理解蛋白质互作的规律和原理。
生物大数据技术解析蛋白质互作网络的流程和技巧蛋白质是生物体内最重要的分子之一,它们在细胞功能的发挥和调控中起着至关重要的作用。
蛋白质通过相互作用构建网络,这些互作网络在细胞中调控着各种生物学过程。
随着生物大数据技术的发展,研究者们能够更好地解析蛋白质互作网络,并从中揭示细胞的运作机制。
本文将介绍利用生物大数据技术分析蛋白质互作网络的流程和技巧。
解析蛋白质互作网络的流程可以分为以下几个步骤。
首先,需要获取蛋白质序列数据和互作信息。
蛋白质序列数据可以通过公开的生物数据库如UniProt得到,而蛋白质互作信息可以通过多种实验手段获得,如质谱联用技术和酵母双杂交等。
其次,对蛋白质序列进行特征提取和编码。
蛋白质序列具有相应的生物学特征,如氨基酸组成、残基重要性和结构域等,这些特征可以用于编码蛋白质序列。
然后,进行蛋白质互作网络的构建。
利用蛋白质互作信息,可以建立蛋白质互作网络,其中蛋白质是网络的节点,互作关系是网络的边。
最后,对构建好的蛋白质互作网络进行分析和解读。
可以通过网络拓扑分析、模块发现和功能注释等方法,揭示蛋白质互作网络的结构特征、模块功能和关键节点等信息。
在分析蛋白质互作网络时,有几个技巧是特别重要的。
首先是特征选择和蛋白质互作网络的重构。
在处理大规模的生物数据时,为了提高计算效率和降低数据的噪声,需要选择合适的特征和方法来重构蛋白质互作网络。
特征选择可以基于统计学、信息论和机器学习等方法进行,帮助我们找到最具有代表性和信息含量的特征。
其次是网络分析的方法选择。
蛋白质互作网络的分析方法非常多样,包括网络拓扑分析、聚类分析和动态模拟等。
选择合适的方法可以更好地理解蛋白质互作网络的结构和功能。
另外,多模态数据的整合也是非常重要的技巧之一。
蛋白质互作网络的构建可以基于多种数据源,如基因表达数据、遗传变异数据和药物作用数据等。
集成不同数据源的信息可以提高蛋白质互作网络的准确性和可解释性。
生物大数据技术为解析蛋白质互作网络提供了强大的工具和方法。
如何利⽤STRING数据库分析蛋⽩间相互作⽤(PPI)?相信很多⼈在做蛋⽩分析的时候,经常被蛋⽩与蛋⽩间的互作⽹络所烦恼,那今天,我们就来给⼤家介绍⼀个神器,帮助⼤家能简单快捷地完成蛋⽩与蛋⽩互作⽹络。
这个软件是STRING。
STRING本⾝就收录了2031个物种,9.6 Million个蛋⽩和1380 Million种相互作⽤。
出来能进⾏蛋⽩-蛋⽩间互作⽹络外,这个数据库还能⽤来查找关注的蛋⽩的调控因⼦,共表达,基因组共线性,物种共存在,⽂本挖掘,实验验证信息等等,是⼀个对科研⼈员来说⼗分实⽤的数据库。
⾸页我们直接点击SEACH,进⾏搜索⾃⼰想要查找的蛋⽩质。
可以按蛋⽩名字搜索,也可以按序列搜索,都可以输⼊多个或多条,也可以按照蛋⽩家族或者物种浏览。
那我们就以trpA为例进⾏说明⼀下吧~这是搜索出来的结果。
每个点代表⼀个蛋⽩,他们都是可以进⾏拖动的。
点击其中⼀个蛋⽩,我们可以查看其详细注释信息,结构信息,功能域信息,序列信息,同源基因,还能以此基因为核⼼重构⽹络(与其相互作⽤最强的基因的⽹络展⽰)等。
由下边的图例解释说明可见,不同颜⾊的线代表相互作⽤确定的依据,有基于认证过的数据库,实验验证,基因邻近,共表达,同源推测,⽂本挖掘等。
如果关联的蛋⽩很多的情况下,我们还能直接通过点的颜⾊,直接找到相关蛋⽩。
同时,我们还可以进⾏筛选,调整线型的含义,相互作⽤的数⽬,数据来源,可信度筛选, 互作点数⽬限制等的不同操作。
⾯对这么多的基因,我们当然希望能做个功能富集分析,这样就能更加直接地看到每个蛋⽩的功能、偏好性等等的信息。
我们也可以直接点击“Analysis”进⾏选择分析。
调整完毕,最后就可以点击Exports输出结果。
今天的介绍就到这⾥了,希望能帮到⼤家喔~。
String:蛋白互作网络(PPI)分析数据库String数据库是一个搜索已知蛋白质之间和预测蛋白质之间相互作用的数据库,该数据库可应用于2031个物种,包含960万种蛋白和1380万中蛋白质之间的相互作用。
它除了包含有实验数据、从PubMed摘要中文本挖掘的结果和综合其他数据库数据外,还有利用生物信息学的方法预测的结果。
研究蛋白之间的相互作用网络,有助于挖掘核心的调控基因,目前已经有很多的蛋白质相互作用的数据库,而string绝对是其中覆盖的物种最多,相互作用信息做大的一个,网址如下:/打开搜索界面简洁明了,有多种方式进行检索:好了下面我们来举个栗子,点击#1进行检索,图中每个节点表示一个蛋白,默认情况下节点的颜色分成红色和白色,红色代表是你的查询蛋白,白色代表与查询蛋白具有相互作用关系的其他蛋白。
由于白色不太好看,string会根据与相互作用的score值对颜色进行映射。
在Legend页面,可以看到每个蛋白的颜色和对应的score值,示意图如下:点击圆圈节点就可以显示该蛋白的具体信息,如下图:圆圈节点之间的直线代表该直线链接的两个蛋白之间的相互作用关系,如图:不同颜色对应不同的相互作用类型,当然你也可以在Settings进行设置,只展示需要的相互作用类型,如图:在Analysis页面,对于蛋白质相互作用网络中的基因,提供了GO和KEGG富集分析的结果,如图:在Exports页面,可以导出相互作用网络的图片,支持PNG, SVG 格式,也可以导出对应的相互作用表格和蛋白序列,注释等信息,如图:如果有多个感兴趣的目标蛋白,想查看它们之间的相互作用关系,则可在搜索界面选择多个蛋白质检索,把目标蛋白的名称输入搜索框,点击搜索。
需要注意的是,如果我们输入的是单个蛋白质名称,数据库将会输出与该蛋白质互作的所有蛋白质的互作图;但如果我们一次输入多个蛋白质名称或者序列,数据库将只输出输入蛋白质之间的互作网络图。
利用STRING 数据库进行蛋白互作预测步骤详解一、简介
STRING (Search Tool for the Retrieval of Interacting Genes/Proteins )数据库(/)是一个搜寻已知蛋白质之间和预测蛋白质之间相互作用的系统,这种相互作用既包括蛋白质之间直接的物理的相互作用,也包括蛋白质之间间接功能的相关性。
STRING 数据库除了包含有实验数据、从PubMed 摘要中文本挖掘的结果和综合其他数据库数据外,还有利用生物信息学的方法预测的结果。
所应用的生物信息学的方法有:染色体临近、基因融合、系统进化谱和基于芯片数据的基因共表达。
系统中利用一个打分机制对这些不同方法得来的结果给予一定的权重,最终给出一个综合的得分。
二、使用方法
1. 打开STRING 网站/
图 2.1
2. 支持多种类型文件来搜索,如名称、蛋白序列、多个名称、多条蛋白序列。
这里,我们
以多个猪转录本Ensembl的ID称为例。
图 2.2
3.输入转录本名称,按“GO!”,进入下个界面;
图 2.3
4.点击“GO”之后会出现如下界面:
图 2.4
5.匹配到的蛋白会自动勾选出来,如图显示
图 2.5
6. 按“Continue”,获得蛋白网络图;如图2.6 所示,可以选择不同的表达类型。
图 2.6
7.不同颜色的圆点,代表不同的蛋白;图2.7 所示为蛋白的注释信息。
图 2.7
8.如图2.8 所示,为蛋白互作关联分析结果,线条的粗细表示关联程度的强弱。
图 2.8
9.保存数据分析的结果;
10.利用Cytoscape去画基因蛋白互作关系;
首先我们要确定与基因互作的蛋白节点关系,这个关系可以从之前的结果页面下载,选择Other fomats:
11.点击Other fomats之后会出现一系列的下载选项,选择下载Text Summary:
12.summary结果展示,前两列即为节点文件,导入Cytoscape即可作图:
14.用cytoscape作图操作流程:
首先我们要安装cytoscape,cytoscape程序官方网站:/
点击进去之后用户可以根据自己的实际情况选择不同的版本,安装没有特别需要注意的地方,一直点击NEXT就可以了,这里我们以cytoscape2.8.3版本进行演示。
1)安装好了点击运行,会出现如下界面:
2)点击左上角的File选项中的Import选项,再选择文本格式输入:
3)选择文本格式输入之后,会出现如下界面,点击Select File(s):
4)选择已经准备好的节点文件,总共两列,每一行代表一种对应关系:
5)选择好文件之后会在界面中展示文件中的内容,如下所示:
6)然后选择互作中的第一列元素,也就是Select Source node column:
7) 再选择第二列元素,也就是Target Interaction的输入数据:
8)其中的Interaction Type选择默认,选择好了之后点击Import:
9)cytoscape会根据我们输入的节点文件自动绘制出互作网络图:
10)根据需要我们可以将网络图认为调整得更加美观(直接点击图中的红色圆圈可以随意拖动),下图为调整后的图形:
11)保存图片:点击File-Export-Current Network View As Graphics(或者直接快捷键:Ctrl+shift+P)
12)选择保存类型,有好多种类型可以自行选择,选择之后点击OK:
13.更多的数据展示,请登录/ 查阅。
三、参考文献
Franceschini et al., STRING v9.1: protein-protein interaction networks, with increased
coverage and integration. Nucleic Acids Res. 2013 Jan。