用ECRBrowser预测转录因子结合位点的方法
CST中国公司学术经理 李振亚 博?
经常会有一些朋友因为转录相关研究而需要进行转录因子结合位点的预测,以通过染色质免疫沉淀(ChIP)方法或其他研究转录调控的方法进行验证。我在这里给大家分享一个我经常使用的在线工具—
ECRBrowser(https://https://www.doczj.com/doc/b517463985.html,),并介绍一下如何用这个工具进行转录因子结合位点的预测。
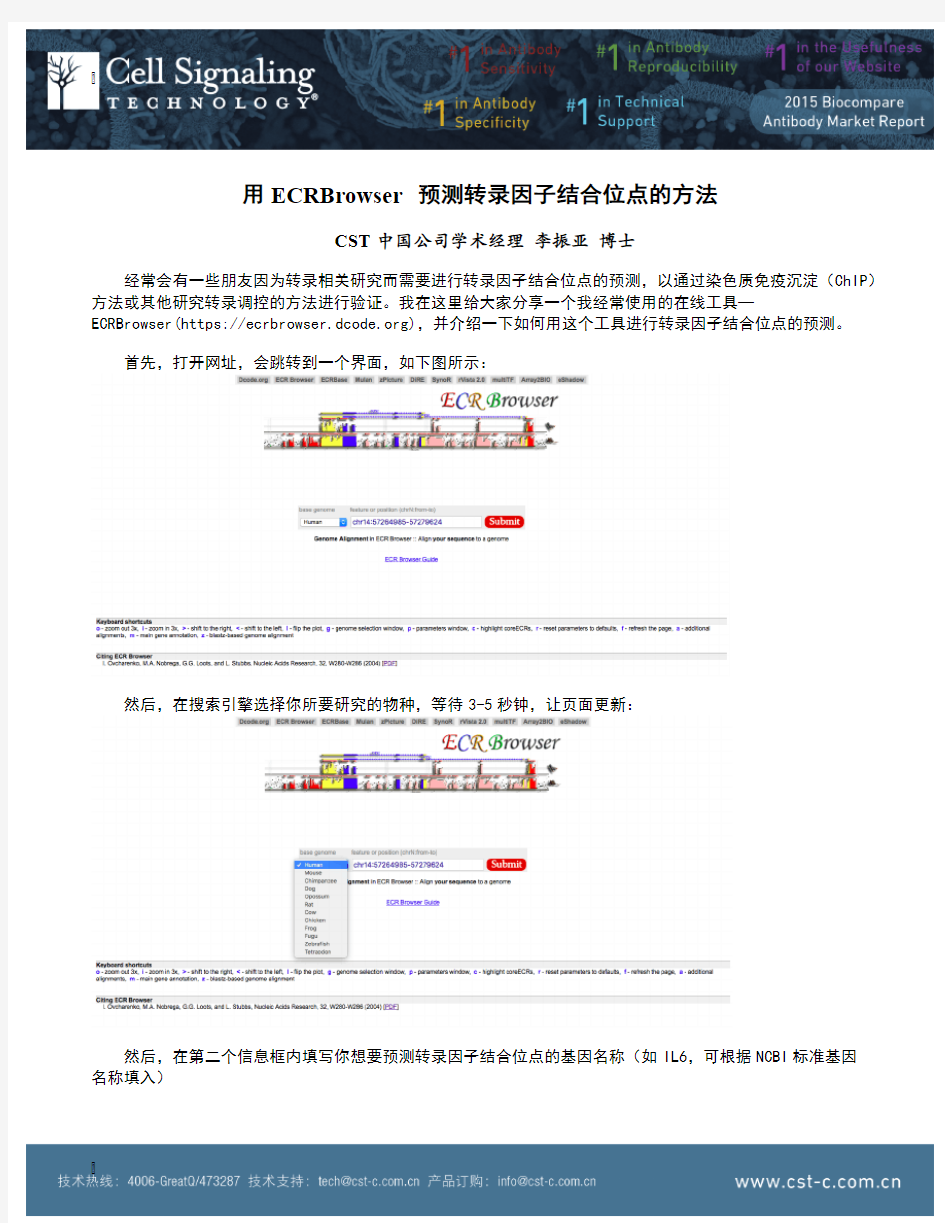
首先,打开网址,会跳转到一个界面,如下图所示:
然后,在搜索引擎选择你所要研究的物种,等待3-5秒钟,让页面更新:
然后,在第二个信息框内填写你想要预测转录因子结合位点的基因名称(如IL6,可根据NCBI标准基因
名称填入)
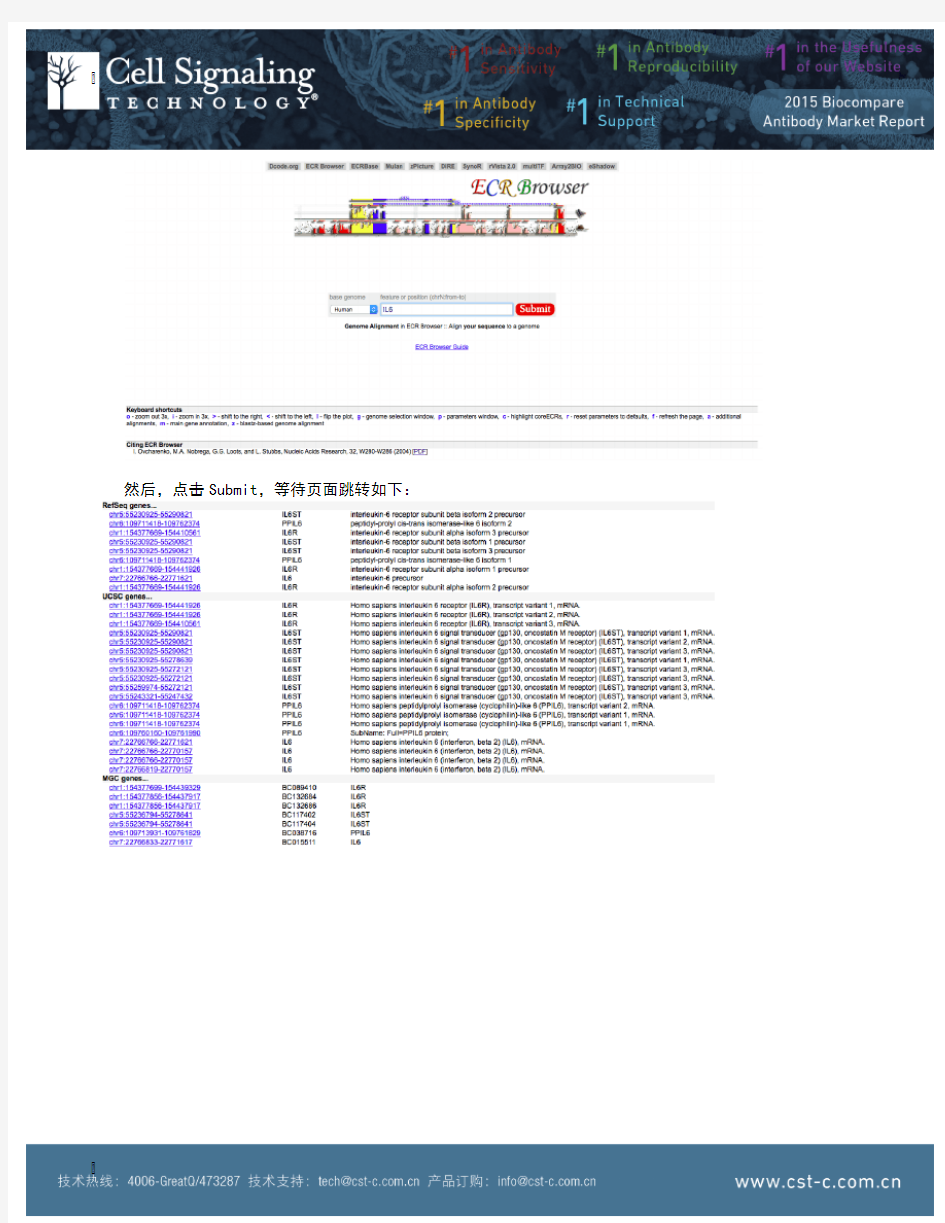
然后,点击Submit,等待页面跳转如下:
在其中选择正确的对应基因名称,由于我一般喜欢用Refseq数据库,所以我点击了套红的那个链接,即chr7:22766766-22771621。
等待页面跳转如下:
此时,这个页面显示的是整个IL6基因的转录区域,即mRNA全长(含内含子)所对应的基因组区域。注意,这个区域并不包括该基因的启动子区域。
那可能大家就要问了,我要预测转录因子结合位点,得需要基因启动子区域啊,那怎么做呢?
按照很多主流转录组学研究的核心启动子区纳入范围,都是在mRNA的起始位点的上游2kb以内。我也是按照这个标准去获取核心启动子区域。对于这个基因IL6,如果要在IL6假定的启动子区域搜索转录因子结合
位点,那么需要手动更改基因组的起止位点。请大家注意,现在页面的显示区域是chr7:22766766-22771621,起始位点是22766766,终止位点是22771621。由于这个基因位于正链(即从左向右转录),我按照上述核心
启动子区域的选定标准,把基因组区域显示范围更改为:chr7:22764766-22766766。大家发现什么规律没有?对啦,就是原来基因组显示的起始位置对应的一串数字变成基因组显示区域终止位置的数字,然后将这串数字减去2000,即得到需要的假定启动子区域起始位置对应的数字了!
那可能有人会问了,如果某一个基因位于负链呢?这个时候你首先会发现这个基因对应的mRNA会显示为
从右向左转录。然后再获取这样的基因假定的启动子区域时,就需要把原来基因组显示的终止位置对应的一串数字变成基因组显示区域起始位置的数字,然后将这串数字加上2000,即得到需要的需要的假定启动子区域
终止位置对应的数字了!
当然,大家可以选择更大的范围(如包含启动子和部分mRNA区域或者全部mRNA区域)进行相关比对和预测,本文只选择了启动子区域进行后续分析。
确定了某一个基因假定启动子区域的起始和终止位置后,点击submit,页面跳转如下:
然后点击页面上部的Synteny/Alignments按钮,如下图所示高亮区域对应按钮:
跳转后的页面如下图所示:
然后点击select all(默认情况下),或者根据自己的需要通过在选择框勾选,而进行和某一个或几个种属的对应基因区域的比对。选定后,点击页面下侧的Mulan按钮,页面会进一步跳转。但有些情况下由于select all获取数据可能会导致运算错误,可能无法使用。因此,我这次选择了进化上相对较近的Rhesus macaque [rheMac2]进行了比对和后续conserved TFBS的预测。如下图所示:
然后点击对应种属那一行的Mulan按钮,页面跳转后如下:
然后点击Submit,会进入运算过程,如图所示:
等待一段时间后,页面会跳转如下:
然后,点击选择框右侧的粉色Continue按钮,页面跳转后如下:
然后点击紫色按钮MULTITF,页面跳转如下:
按照默认设置,或更改一些自己需要的设置后,点击SUBMIT,页面会跳转如下:
页面安装转录因子的名称字母排序,大家可以在这张列表里勾选自己感兴趣的转录因子,也可以按照默认设置,在页面底部选择SELECT ALL(本文是按照SELECT ALL进行运算)
然后,点击SUBMIT,页面进入运算过程,如图所示:
点击CHECK IT,获取数据列表如下
大家可以根据自己需要点击不同按钮,获得不同数据集,默认情况下选择multi-conserved那个按钮,页
面跳转后如下:
这个结果显示的转录因子结合位点所在的DNA链(+或-)以及起止位点。而这些起止位点是指选定的2001个bp内从左到右排序。大家可以利用刚才的基因组显示区域的设置,如图所示:
在页面上部点击DNA按钮,下载这2001bp的假定启动子区域的序列,再进行仔细分析。如图所示:
如果确定某一个转录因子的位点和文献报道的保守序列接近,即可在上图中标记出来这个结合位点,并在此结合位点向上下游各选择300bp,共计约600bp,进行引物设计。一般可以利用NCBI的PrimerBLAST进行设计。既可以将这约600bp的序列粘贴后进行设计,也可以通过限制引物搜索范围进行设计。
10生物学通报2005年第40卷第11期 2003年即Watson和Crick发表DNA双螺旋结构50周年,宣布了人类基因组计划的完成,与此同时,其他许多生物的基因组计划已完成或在进行中,在此过程中产生的大量数据库对科学研究的深远影响是以前任何人未曾预料到的。然而遗憾的是,许多生物学家、化学家和物理学家对这些数据库的使用甚至去何处寻找这些数据库都只有一个比较模糊的概念。 基因转录是遗传信息传递过程中第一个具有高度选择性的环节,近20年来对基因转录调节的研究一直是基因分子生物学的研究中心和热点,因此亦产生了大量很有价值的数据库资源,对这些数据库的了解将为进一步研究带来极大便利,本文对其中一些数据库进行简要介绍。 1DBTSS DBTSS(DataBaseofTranscriptionalStartSites)由东京大学人类基因组中心维护,网址:http://dbtss.hgc.jp。最初该数据库收集用实验方法得到的人类基因的TSS(TranscriptionalStartSites,转录起始位点)数据。对转录起始位点(TSS)的确切了解具有非常重要的意义,可更准确的预测翻译起始位点;可用于搜索决定TSS的核苷酸序列,而且可更精确地分析上游调控区域(启动子)。自2002年发布第一版以来已作了多次更新。目前包含的克隆数为190964个,含盖了11234个基因,在SNP数据库中显示了人类基因中的SNP位点,而且现在含包含了鼠等其他生物的相关数据。DBTSS最新的版本为3.0。 在该最新的版本中,还新增了人和鼠可能同源的启动子,目前可以显示3324个基因的启动子,通过本地的比对软件LALIGN可以图的形式显示相似的序列元件。另一个新的功能是可进行与已知转录因子结合位点相似的部位的定位,这些存贮在TRANSFAC(http://transfac.gbf.de/TRANSFAC/index.html)数据库中,免费用于研究,但TRANSFAC专业版是商业版本。 DBTSS对匿名登录的用户是免费的,该网站要求用户在使用前注册,用户注册后即可使用。主页分为2个区域,一个介绍网站的部分信息和用户注册,另一区域为用户操作区,该区约分为10个部分,可分别进行物种和数据库的选择、BLAST、SNP以及TF(转录因子)结合部位搜索等部分。后者的使用可以见网页中的Help部分,里面有比较详细的介绍。DBTSS还提供了丰富的与其他相关网站的链接,如上文提到的TRANSFAC数据库、真核生物启动子数据库(Eukaryot-icPromoterDatabase,http://www.epd.isb-sib.ch/)以及人类和其他生物cDNA全长数据库等。 2JASPAR JASPAR是有注释的、高质量的多细胞真核生物转录因子结合部位的开放数据库。网址http://jaspar.cgb.ki.se。所有序列均来源于通过实验方法证实能结合转录因子,而且通过严格的筛选,通过筛选后的序列再通过模体(motif)识别软件ANN-Spec进行联配。ANN-Spec利用人工神经网络和吉布斯(Gibbs)取样算法寻找特征序列模式。联配后的序列再利用生物学知识进行注释。 目前该数据库收录了111个序列模式(profiles),目前仅限于多细胞真核生物。通过主页界面,用户可进行下列操作:1)浏览转录因子(TF)结合的序列模式;2)通过标识符(identifier)和注解(annotation)搜索序列模式;3)将用户提交的序列模式与数据库中的进行比较;4)利用选定的转录因子搜索特定的核苷酸序列,用户可到ConSite服务器(http://www.phylofoot.org/consite)进行更复杂的查询。JASPAR数据库所有内容可到主页下载。 与相似领域数据库相比,JASPAR具有很明显优势:1)它是一个非冗余可靠的转录因子结合部位序列模式;2)数据的获取不受限制;3)功能强大且有相关的软件工具使用。JASPAR与TRANSFAC(一流的TF数据库)有较明显的差异,后者收录的数据更广泛,但包含不少冗余信息且序列模式的质量参差不齐,是商业数据库,只有一部分是可以免费使用。用户在使用过程中会发现二者的差异,这主要是由于二者对数据的收集是相互独立的。另外该数据库还提供了相关的链接:如MatInspector检测转录因子结合部位,网址http://transfac.gbf.de/programs/matinspector/;TESS转录元件搜索系统,网址http://www.cbil.upenn.edu/tess/。 转录调节位点和转录因子数据库介绍! 张光亚!!方柏山 (华侨大学生物工程与技术系福建泉州362021) 摘要转录水平的调控是基因表达最重要的调控水平之一,对转录调节位点和转录因子的研究具有重要意义。介绍了DBTSS、JASPAR、PRODORIC和TRRD等相关数据库及其特征、内容和使用。 关键词转录调节位点转录因子数据库生物信息学 !基金项目:国务院侨办科研基金资助项目(05QZR06) !!通讯作者
ChIP-Seq技术在转录因子结合位点分析的应用 摘要:染色质免疫沉淀(Chromatin immunoprecipitaion, ChIP)技术是用来研究细胞 内特定基因组区域特定位点与结合蛋白相互作用的技术。将ChIP与第二代高通量测序技术相结合的染色质免疫沉淀测序(chromatin immunoprecipitation followed by sequencing,ChIP-Seq)技术能在短时间内获得大量研究数据,高效地在全基因组范围内检测与组蛋白、转录因子等相互作用的DNA区段,在细胞的基因表达调控网络研究中发挥重要作用。本文 简要介绍了ChIP-Seq技术的基本原理、实验设计和后续数据分析,以及ChIP-Seq技术在 研究转录因子结合位点中的。 关键词:ChIP-Seq;转录因子; 引言 染色质是真核生物基因组DNA主要存在形式,为了阐明真核生物基因表达调控机制,对于蛋白质与DNA在染色质环境下的相互作用的研究是基本途径。转录因子是参与基因表达调控的一类重要的细胞核蛋白质,基因的转录调控是生物基因表达调控层次中最关键的一层,转录因子通过特异性结合调控区域的DNA序列来调控基因转录过程。转录因子由基础转录因子和调控性转录因子两类组成,其中基础转录因子在转录起始位点附近的启动子区,与RNA聚合酶相互作用实现基因的转录;而调控性转录因子一般与位置多样的增强子序列结合,再通过形成增强体在组织发育、细胞分化等基因表达水平调控中发挥极其重要的作用[1]。 ChIP-Seq是近年来新兴的将ChIP与新一代测序技术相结合,在全基因s组范围内分析转录因子结合位点(transcription factor binding sites,TFBS)、组蛋白修饰(histone modification)、核小体定位(nucleosome positioning)和DNA 甲基化(DNA methylation)的高通量方法[2-4]。其中ChIP是全基因组范围内识别DNA与蛋白质体内相互作用的标准方法[5],最初用于组蛋白修饰研究[6],后来用于转录因子[7]。同时,新一代测序技术的迅猛发展也将基因组学水平的研究带入了一个新的阶段,使得许多基于全基因组的研究成为可能。相对于传统的基于芯片的ChIP-chip (chromatin immunoprecipitation combined with DNA tiling arrays),ChIP-seq 提供了一种高分辨率、低噪音、高覆盖率的研究蛋白质-DNA 相互作用的手段[8],可以应用到任何基因组序列已知的物种,可以研究任何一种DNA 相关蛋白与其靶定DNA 之间的相互作用,并能确切得到每一个片段的序列信息.随着测序成本的降低,ChIP-seq 逐步成为研究基因调控和表观遗传机制的一种常用手段。此外,为了达到更好的检测效果和更为完整的信息,近年来,将ChIP-Seq和ChIP-chip两者融合的研究具有很好的应用前景[9,10]。 转录因子在器官发生过程中起至关重要的作用,在全基因组水平将转录因子定位于靶基因DNA是认识转录调控网络的有效方法之一,了解基因转录调控的关键是识别蛋白质与DNA的相互作用。ChIP-Seq技术能够揭示转录因子的结合位点和确定直接的靶基因序列,可在体内分析特定启动子的分子调控机制,因此被广泛应用于转录调控机制的研究。本文主要就这一技术在转录因子结合位点研究中的基本原理、实验设计和数据分析等技术层面、以及实际应用层面进行讨论。 1 ChIP-seq基本原理及实验设计 1.1 ChIP技术 蛋白质与DNA相互识别是基因转录调控的关键,也是启动基因转录的前提。ChIP是在全基因组范围内检测DNA与蛋白质体内相互作用的标准方法[11],该技术由Orlando等[12]于1997年创立,最初用于组蛋白修饰的研究,后来广泛应用到转录因子作用位点的研究中[13]。ChIP的基本原理为:活细胞采用甲醛交联后裂解,染色体分离成为一定大小的片段,然后用特异性抗体免疫沉淀目标蛋白与DNA交联的复合物,对特定靶蛋白与DNA片段进行
转录因子 ? 1 简介 ? 2 方法 ? 3 转录因子 转录因子-简介 基因转录有正调控和负调控之分。如细菌基因的负调控机制是当一种阻遏蛋白(repressor protein)结合在受调控的基因上时,基因不表达;而从靶基因上去除阻遏蛋白后,RNA聚合酶识别受调控基因的启动子,使基因得以表达,这是正调控。这种阻遏蛋白是反式作用因子。 转录因子(transcription factor)是起正调控作用的反式作用因子。转录因子是转录起始过程中RNA聚合酶所需的辅助因子。真核生物基因在无转录因子时处于不表达状态,RNA聚合酶自身无法启动基因转录,只有当转录因子(蛋白质)结合在其识别的DNA序列上后,基因才开始表达。 转录因子的结合位点(transcription factor binding site,TFBS)是转录因子调节基因表达时,与mRNA结合的区域。按照常识,转录因子(transcription factor,TF)的结合位点一般应该分布在基因的前端,但是,新的研究发现,人21和22号染色体上,只有22%的转录因子结合位点分布在蛋白编码基因的5'端。 转录因子-方法 这篇文章的试验方法是,通过高密度的寡核苷酸芯片,反映出人21和22号染色体的几乎所有的非重复序列,通过这种芯片,检测三种转录因子,Sp1、 cMyc、和p53的结合位点。结果表明,每种转录因子都有大量的TFBS与之结合。然而,只有22%的转录因子结合位点分布在蛋白编码基因的5'端, 36%的TFBS分布在蛋白编码基因的中部或3'端,并且这36%的TFBS常常和基因组中的非蛋白编码RNA分布在一起。这暗示,在人的基因组中,不仅包含蛋白编码基因,也包含数量相当的非编码基因(noncoding genes),他们都受常见的转录因子所调控。 真核生物在转录时往往需要多种蛋白质因子的协助。一种蛋白质是不是转录机构的一部分往往是通过体外系统看它是否是转录起始所必须的。一般可将这些转录所需的蛋白质分为三大类: (1)RNA聚合酶的亚基,它们是转录必须的,但并不对某一启动子有特异性。 (2)某些转录因子能与RNA聚合酶结合形成起始复合物,但不组成游离聚合酶的
转录因子 基因转录有正调控和负调控之分。如细菌基因的负调控机制是当一种阻遏蛋白(repressor protein)结合在受调控的基因上时,基因不表达;而从靶基因上去除阻遏蛋白后,RNA聚合酶识别受调控基因的启动子,使基因得以表达,这是正调控。这种阻遏蛋白是反式作用因子。而顺式作用因子则指的是基因上与反式作用因子结合的对基因表达起调控作用的基因序列。 转录因子(transcription factor)是起正调控作用的反式作用因子。转录因子是转录起始过程中RNA聚合酶所需的辅助因子。真核生物基因在无转录因子时处于不表达状态,RNA聚合酶自身无法启动基因转录,只有当转录因子(蛋白质)结合在其识别的DNA序列上后,基因才开始表达。 转录因子的结合位点(transcription factor binding site,TFBS)是转录因子调节基因表达时,与mRNA结合的区域。按照常识,转录因子(transcription factor,TF)的结合位点一般应该分布在基因的前端,但是,新的研究发现,人21和22号染色体上,只有22%的转录因子结合位点分布在蛋白编码基因的5'端。 真核生物在转录时往往需要多种蛋白质因子的协助。一种蛋白质是不是转录机构的一部分往往是通过体外系统看它是否是转录起始所必须的。一般可将这些转录所需的蛋白质分为三大类: (1)RNA聚合酶的亚基,它们是转录必须的,但并不对某一启动子有特异性。 (2)某些转录因子能与RNA聚合酶结合形成起始复合物,但不组成游离聚合酶的成分。这些因子可能是所有启动子起始转录所必须的。但亦可能仅是譬如说转录终止所必须的。但是,在这一类因子中,要严格区分开哪些是R NA聚合酶的亚基,哪些仅是辅助因子,是很困难的。 (3)某些转录因子仅与其靶启动子中的特异顺序结合。如果这些顺序存在于启动子中,则这些顺序因子是一般转录机构的一部分。如果这些顺序仅存在于某些种类的启动子中,则识别这些顺序的因子也只是在这些特异启动子上起始转录必须的。 黑腹果蝇的RNA聚合酶需要至少两个转录因子方能在体外起始转录。其中一个是B因子,它与含TATA盒的部位结合。人的因子TFⅡD亦和类似的部位结合。同样,CTF(CAAT结合因子)则与腺病毒的主要晚期启动子中与CAAT盒同源的部位相结合。结合在上游区的另一个转录因子是USF(亦称MLTF),则可以识别腺病毒晚期启动子中靠近-55的顺序。转录因子Sp1则能和GC盒相结合。在SC40启动子中有多个GC盒,位于-70到-110之间。它们均能和Sp1相结合。然而含有GC盒的不同的DNA顺序与Sp1的亲和力却各不相同。可见GC盒两侧的顺序对Sp1-GC盒的结合究竟如何能影响转录。有时候需要几个转录因子才能起始转录。例如胞苷激酶的启动子需要S p1与GC盒结合和CTF与CAAT盒结合;腺病毒晚期启动子需要TFⅡD与TATA盒结合和USF与其邻近部位相结合。以上所述的因子是一般转录都需要的,似乎并没有什么调节功能。另一些转录因子则可以调控一组特殊基因的转录。热休克基因就是一个很好的例子。真核生物的热休克基因在转录起始点的上游15bp处有一个共同顺序。H STF因子仅在热休克细胞中有活性。它与包括热休克共同顺序在内的一段DNA相结合,所以这个因子的激活可以引起约包括20个基因的一组基因起始转录。在这里,转录因子和RNA聚合酶Ⅱ之间关系很类似细菌的σ因子与核心酶之间的关系。 转录因子是一种具有特殊结构、行使调控基因表达功能的蛋白质分子,也称为反式作用因子。植物中的转录因子分为二种,一种是非特异性转录因子,它们非选择性地调控基因的转录表达,如大麦(Hordeum vulgare) 中的HvCBF2 (C-repeat/DRE binding factor 2) (Xue et al., 2003)。还有一种称为特异型转录因子,它们能够选择性调控某种或某些基因的转录表达。典型的转录因子含有DNA结合区(DNA-binding domain)、转录调控区(acti vation domain)、寡聚化位点(oligomerization site) 以及核定位信号(nuclear localization signal) 等功能区域。这些功能区域决定转录因子的功能和特性(Liu et al., 1999)。DNA结合区带共性的结构主要有:1)HTH 和HL H 结构:由两段α-螺旋夹一段β-折叠构成,α-螺旋与β-折叠之间通过β-转角或成环连接,即螺旋-转角-螺旋结构和螺旋-环-螺旋结构。2)锌指结构:多见于TFIII A 和类固醇激素受体中,由一段富含半胱氨酸的多肽链构成。每四个半光氨酸残基或组氨酸残基螯合一分子Zn2+ ,其余约12-13 个残基则呈指样突出,刚好能嵌入DNA 双螺旋的大沟中而与之相结合。3)亮氨酸拉链结构:多见于真核生物DNA 结合蛋白的 C 端,与癌基因表达调控有关。由两段α - 螺旋平行排列构成,其α - 螺旋中存在每隔7 个残基规律性排列的亮氨酸残基,亮氨酸侧链交替排列而呈拉链状,两条肽链呈钳状与DNA 相结合。
关于组蛋白、甲基化、转录因子、结合位点和CHIP-Seq 1)染色质:真核细胞分裂间期的细胞核内的一种物质,这种物质的基本化学成分为脱氧核 糖核酸核蛋白(核蛋白就是由DNA或RNA与蛋白质形成的复合体),主要由DNA和组蛋白构成,也含有少量的非组蛋白和RNA。由于它可以被碱性的染料染色,所以称为染色质。在细胞的有丝分裂期,染色质经过螺旋、折叠,包装成了染色体。 2)核小体:核小体是染色体的基本结构单位,由DNA和组蛋白(histone)构成,是染色质(染 色体)的基本结构单位。由4种组蛋白H2A、H2B、H3和H4,每一种组蛋白各二个分子,形成一个组蛋白八聚体,约200 bp的DNA分子盘绕在组蛋白八聚体构成的核心结构外面,形成了一个核小体。这时染色质的压缩包装比(packing ratio)为6左右,即DNA 由伸展状态压缩了近6倍。200 bp DNA为平均长度;不同组织、不同类型的细胞,以及同一细胞里染色体的不同区段中,盘绕在组蛋白八聚体核心外面的DNA长度是不同的。如真菌的可以短到只有154 bp,而海胆精子的可以长达260bp,但一般的变动范围在180bp到200bp之间。在这200bp中,146 bp是直接盘绕在组蛋白八聚体核心外面,这些DNA不易被核酸酶消化,其余的DNA是用于连接下一个核小体。连接相邻2个核小体的DNA分子上结合了另一种组蛋白H1。组蛋白H1包含了一组密切相关的蛋白质,其数量相当于核心组蛋白的一半,所以很容易从染色质中抽提出来。所有的H1被除去后也不会影响到核小体的结构,这表明H1是位于蛋白质核心之外的。 3)染色体:在细胞的有丝分裂的分裂期由染色质经螺旋折叠形成,呈线状或棒状。 4) 有丝分裂:真核细胞的染色质凝集成染色体、复制的姐妹染色单体在纺锤丝的牵拉下分 向两极,从而产生两个染色体数和遗传性相同的子细胞核的一种细胞分裂类型。分裂具有周期性。即连续分裂的细胞,从一次分裂完成时开始,到下一次分裂完成时为止,为一个细胞周期。一个细胞周期包括两个阶段:分裂间期和分裂期,(这两个阶段所占的时间相差较大,一般分裂间期占细胞周期的90%-95%;分裂期大约占细胞周期的5%-10%。细胞种类不同,一个细胞周期的时间也不相同。)分裂期又分为分裂前期、分裂中期、分裂后期和分裂末期。细胞在分裂之前,必须进行一定的物质准备。细胞增殖包括物质准备和细胞分裂整个过程。有丝分裂是一个连续的过程按先后顺序划分为间期、前期、中期、后期和末期五个时期,在前期和中期之间有时还划分出一个前中期。 5) 分裂间期:主要完成DNA的复制和蛋白质的合成,DNA复制时边解旋编复制。 6) 姐妹染色单体:姐妹染色单体是指染色体在细胞有丝分裂(包括减数分裂)的间期进 行自我复制,形成由一个着丝点连接着的两条完全相同的染色单体。(若着丝点分裂,则就各自成为一条染色体了)。每条姐妹染色单体含1个DNA。 7) 同源染色体:二倍体细胞中染色体以成对的方式存在, 一条来自父本,一条来自母本, 且形态、大小相同,并在减数分裂前期相互配对的染色体。含相似的遗传信息。 8) 组蛋白:一组进化上非常保守的碱性蛋白质,其中碱性氨基酸(Arg,Lys)约占25%,存 在于真核生物染色质,分为5种类型(H1,H2A,H2B,H3,H4),后4种各2个形成组蛋白八聚体,构成核小体的核心,占核小体质量的一半。组蛋白的基因非常保守。亲缘关系较远的种属中,四种组蛋白(H2A、H2B、H3、H4)氨基酸序列都非常相似。 9) 甲基化(methylation):从活性甲基化合物(如S-腺苷基甲硫氨酸)上催化其甲基转移到其 他化合物的过程。可形成各种甲基化合物,或是对某些蛋白质或核酸等进行化学修饰形成甲基化产物。甲基化是蛋白质和核酸的一种重要的修饰,调节基因的表达和关闭,与癌症、衰老、老年痴呆等许多疾病密切相关,是表观遗传学的重要研究内容之一。最常见的甲基化修饰有DNA甲基化和组蛋白甲基化。DNA甲基化是指生物体在DNA甲基转移酶(DNA methyltransferase,DMT) 的催化下,以s-腺苷甲硫氨酸(SAM)为甲基
角朊细胞 角朊细胞的增殖和分化是一个受到精细调节的过程,并伴随着一系列形态学和生化改变,最终形成角质细胞,这就必然涉及到许多结构基因的同时活化与灭活,即基因表达的调控,而转录水平的调控尤为重要。现已发现许多转录因子如AP1、AP2、Sp1、POU结构域及C/EBP等可调节角朊细胞基因的表达。 目录
转录水平、翻译水平及翻译后水平,其中最常见的调控方式就是转录调控。现已发现AP1、AP2、NFκB、C/EBP、ets、Sp1及POU结构域等转录因子可作为表皮中的调控蛋白,从而调节编码套膜蛋白(involucrin, iNV)、转谷氨酰胺酶(transglutaminase,TG)、SPRR2A、兜甲蛋白(loricrin)、角蛋白及BPAG1等蛋白的基因的表达。本文就与角朊细胞基因表达有关的转录因子作一简要综述。 编辑本段转录因子的一般特征 转录因子(transcription factor)是能与位于转录起始位点上游50~5000bp的顺式作用元件(cis-acting elements)、沉默子(silencer)或增强子(enhancer)结合并参与调节靶基因转录效率的一组蛋白,并能将来自细胞表面的信息传递至核内基因。转录因子通常有几个功能域,可分为DNA结合域、转录调控域及自身活性调控域,DNA结合域可与特定的DNA序列(一般长8~20bp)相互作用,使转录因子与靶基因结合起来,随之转录调控域就可发挥其激活或抑制作用,通常这些结构域在结构与功能上是独立分开的。不同的转录因子还可结合于紧密相邻的DNA序列而形成一种多聚体结构来调节基因表达,这种组合调控(combinatorial regulation)不论转录因子是否激活及其含量多少均可激活基于靶基因中特定转录因子结合位点的转录。除启动基础转录活性外,转录因子还能整合从细胞表面经信号转导途径传递而来的信号[2]。 编辑本段激活角朊细胞基因表达的转录因子 (一)AP1 AP1转录因子通常以jun(c-jun、junB、junD)与Fos(Fra-1、Fra-2、c-fos、fosB)家族成员组成的同源或异源二聚体表达其活性,即结合于5’-GTGAGCTCAG-3’序列。目前已知AP1位点对于编码角蛋白(K1、K5、 K6及K19)、丝聚合蛋白原(profilaggrin)基因的最适转录活性十分重要[3,7],编码角质化包膜(cornified envelope)相关蛋白-TG1、兜甲蛋白及INV的基因也含有功能性AP1 位点[8,9],如hINV基因启动子在其转录起始位点上游2.5kb内有5个AP1共有结合位点(AP1-1~5),其中2个AP1位点AP1-1和AP1-5若同时发生突变时角朊细胞的转录水平就可下降80%;佛波酯(TPA)则可使AP1与hINV启动子处AP1-1及AP1-5位点的结合能力增强10~100倍,后经点突变实验证实AP1-1和AP1-5位点可部分介导佛波酯(TPA)诱导的效应[10]。丝聚合蛋白原、K1、兜甲蛋白及K19基因中的AP1位点可活化转录[3,6,7],
启动子分析-----------转录因子结合位点 启动子分析-----------转录因子结合位点 启动子是DNA分子可以与RNA聚合酶特异结合的部位,也就是使转录开始的部位。在基因表达的调控中,转录的起始是个关键。常常某个基因是否应当表达决定于在特定的启动子起始过程。启动子一般可分为两类: (1)一类是RNA聚合酶可以直接识别的启动子。这类启动子应当总是能被转录。但实际上也不都如此,外来蛋白质可对其有影响,即该蛋白质可直接阻断启动子,也可间接作用于邻近的DNA结构,使聚合酶不能和启动子结合。 (2)另一类启动子在和聚合酶结和时需要有蛋白质辅助因子 的存在。这种蛋白质因子能够识别与该启动子顺序相邻或甚至重叠的DNA顺序。 因此,RNA聚合酶能否与启动子相互作用是起始转录的关键问题,似乎是蛋白质分子如何能识别DNA链上特异序列。例如,RNA聚合酶分子上是否有一个活性中心能够识别出DNA双螺旋上某特异序列的化学结构?不同启动子对RNA 聚合酶的亲和力各不同。这就可能对调控转录起始的频率,亦即对基因表达的程度有重要不同。DNA链上从启动子直到
终止子为止的长度称为一个转录单位。一个转录单位可以包括一个基因,也可以包括几个基因。启动子预测软件大体分为三类,第一类是启发式的方法,它利用模型描述几种转录因子结合部位定向及其侧翼结构特点,它具有挺高的特异性,但未提供通用的启动子预测方法;第二类是根据启动子与转录因子结合的特性,从转录因子结合部位的密度推测出启动子区域,这方法存在较高的假阳性;另一类是根据启动子区自身的特征来进行测定,这种方法的准确性比较高。同时,还可以结合是否存在CpG岛,而对启动子预测的准确性做出辅助性的推测。 启动子预测软件有:PromoterScan ; Promoter 2.0 ; NNPP ;EMBOSS Cpgplot ; CpG Prediction 启动子及转录因子结合位点数据库及预测工具 冷泉港启动子分析程序介绍 https://www.doczj.com/doc/b517463985.html,/links/ch_09_t_6.html 在线预测和分析基因启动子(promoter) 一般在公共数据库中,如NCBI、UCSC、Ensembl给出的人类基因序列都没有对基因进行详细的标注。不过,有
转录因子 摘要:随着众多生物基因组计划的完成及其蛋白质组学研究的不断深入,人类步入了系统生物学时代。基因组计划的完成提供了大量的DNA内在信息,解析出基因组中可能存在的全部基因的阅读框架,因此,接下来研究基因的表达调控特别是转录调控就显得非常迫切。另一方面,蛋白组学研究的突飞猛进给我们描绘出了细胞的蛋白质表达谱和网络谱,接下来研究蛋白质与蛋白质,蛋白质与DNA的相互作用将成为现在及以后相当长一段时间内的研究主题。有生物学家认为,21世纪对人类最具有挑战性的生物学主题就是“基因的全基因组调控”和”细胞的全蛋白质的生理功能”这两大难题。 然而,转录因子是可与基因调控序列结合并调控基因转录的一类核蛋白,研究转录因子就是研究转录调控的分子机制,研究一种或一类特定的蛋白质分子与DNA的结合特性,研究与DNA结合的蛋白质分子是怎样调控基因转录等问题。转录因子的研究实际上已构成上述两大生物学难题的一个交叉点,因此,对转录因子的深入研究已是一件极其迫切而且重要的课题。 DNA转录及转录因子 定义 转录:是指以DNA为模板,在RNA聚合酶的作用下合成mRNA,将遗传信息从DNA分子上转移到mRNA分子上,这一过程成为转录。真核生物DNA的转录在细胞核中进行,原核生物的转录在细胞质的核质区
内进行。 转录单元 转录单元是一段以启动子开始至终止子结束的DNA序列。 转录起始(transcription initiation):转录因子通过识别基因启动子上的特异顺式元件并募集多种蛋白质因子,形成具有RNA聚合酶活性的转录起始复合体,从转录起始位点启动转录的过程。 转录终止子(transcription terminator):基因编码区下游使RNA聚合酶终止mRNA合成的密码子,是一种位于poly(A)位点下游,长度在几百碱基以内的结构。 终止子可分为两类。一类不依赖于蛋白质辅因子就能实现终止作用。另一类则依赖蛋白辅因子才能实现终止作用。这种蛋白质辅因子称为释放因子,通常又称ρ因子 转录因子:能够结合在某基因上游特异核苷酸序列上的蛋白质,活化后从胞质转位至胞核,通过识别和结合基因启动子区的顺式作用元件,启动和调控基因表达。 转录因子是转录起始过程中RNA聚合酶所需的辅助因子。真核生物基因在无转录因子时处于不表达状态,RNA聚合酶自身无法启动基因转录,只有当转录因子(蛋白质)结合在其识别的DNA序列上后,基因才开始表达。转录因子是结合在某基因上游特异核苷酸序列上的蛋白质,这些蛋白质能调控该基因的转录。转录因子可以调控核糖核酸聚合酶(RNA聚合酶)与DNA模板的结合。转录因子不单与DNA序列上的启动子结合,也可以和其它转录因子形成-转录因子聚合体,来影
酵母转录因子结合位点保守性的生物信息学分析 【摘要】目的:本研究拟发掘出酵母基因组中转录因子结合位点的保守性位点和规律。方法:本研究采用生物信息学中保守性模体参数Mi分析基因上游不同区域与真核生物转录因子结合位点保守性之间的关系。结果:转录因子Sok2、Swi4结合位点的保守性在基因转录起始位点上游各个区间的差异主要由其本身的序列特性决定。此外,本研究分别发掘出转录因子Sok2、Swi4结合位点的保守性位点。结论:本研究结果有助于提供新的参数用以改进现有预测转录因子结合位点的方法,在此基础上为深入研究真核生物的转录水平调控模式奠定理论基础。 【关键词】酵母; 转录因子; 结合位点;保守性;生物信息学 真核基因的表达调控可在多个层次上进行,但主要表现在对基因转录活性的调控上[1]。转录因子与对应DNA序列结合调控其目标靶基因的表达是基因表达调控的核心问题,因此转录水平的调控是真核基因表达最基本的调控方式[2]。转录因子不但可以结合在DNA序列上调控基因转录的起始,同时也可以招募组蛋白修饰酶,对转录因子结合位点附近的组蛋白进行修饰,而组蛋白修饰又可以促进DNA与转录因子的结合,还可能产生新的转录因子结合位点。正是由于不同发育阶段特异、细胞特异的反式作用因子与相应DNA调节元件的结合,导致了基因的差异表达[3]。 本研究以真核模式生物酵母的转录因子为研究材料,从酵母基因组的数据库SGD里提取转录因子结合位点的数据。研究结果将为为更加准确的预测真核生物转录因子结合位点提供数据支持,并且为更深入的解析真核生物转录调控网络奠定理论基础。 1材料与方法 1.1通过SGD数据库获得结合位点数据 酵母基因组数据库SGD 是已经完成基因组全序列测定的啤酒酵母基因组数据库, 包括啤酒酵母的分子生物学及遗传学等大量信息。从文献所报道的117个转录因子及其所调节的基因中,选取转录因子调控基因数目最多的两个转录因子Sok2、Swi4,研究其结合位点保守性。 1.2一致性序列选取 转录因子的一致性序列分别确定为:Sok2 TGCAGNNA(SGD);Gcn4 TGACTCA(TRANSFAC);对于转录因子Swi4有特殊处理,因为其常见结合一致性序列为CAAGAAAA和CGCSAAA(SGD),并且SGD数据里所给转录因子Swi4在TSS上游的结合位点为九位。
用ECRBrowser预测转录因子结合位点的方法 CST中国公司学术经理 李振亚 博? 经常会有一些朋友因为转录相关研究而需要进行转录因子结合位点的预测,以通过染色质免疫沉淀(ChIP)方法或其他研究转录调控的方法进行验证。我在这里给大家分享一个我经常使用的在线工具— ECRBrowser(https://https://www.doczj.com/doc/b517463985.html,),并介绍一下如何用这个工具进行转录因子结合位点的预测。 首先,打开网址,会跳转到一个界面,如下图所示: 然后,在搜索引擎选择你所要研究的物种,等待3-5秒钟,让页面更新: 然后,在第二个信息框内填写你想要预测转录因子结合位点的基因名称(如IL6,可根据NCBI标准基因 名称填入)
然后,点击Submit,等待页面跳转如下:
在其中选择正确的对应基因名称,由于我一般喜欢用Refseq数据库,所以我点击了套红的那个链接,即chr7:22766766-22771621。 等待页面跳转如下: 此时,这个页面显示的是整个IL6基因的转录区域,即mRNA全长(含内含子)所对应的基因组区域。注意,这个区域并不包括该基因的启动子区域。 那可能大家就要问了,我要预测转录因子结合位点,得需要基因启动子区域啊,那怎么做呢? 按照很多主流转录组学研究的核心启动子区纳入范围,都是在mRNA的起始位点的上游2kb以内。我也是按照这个标准去获取核心启动子区域。对于这个基因IL6,如果要在IL6假定的启动子区域搜索转录因子结合
位点,那么需要手动更改基因组的起止位点。请大家注意,现在页面的显示区域是chr7:22766766-22771621,起始位点是22766766,终止位点是22771621。由于这个基因位于正链(即从左向右转录),我按照上述核心 启动子区域的选定标准,把基因组区域显示范围更改为:chr7:22764766-22766766。大家发现什么规律没有?对啦,就是原来基因组显示的起始位置对应的一串数字变成基因组显示区域终止位置的数字,然后将这串数字减去2000,即得到需要的假定启动子区域起始位置对应的数字了! 那可能有人会问了,如果某一个基因位于负链呢?这个时候你首先会发现这个基因对应的mRNA会显示为 从右向左转录。然后再获取这样的基因假定的启动子区域时,就需要把原来基因组显示的终止位置对应的一串数字变成基因组显示区域起始位置的数字,然后将这串数字加上2000,即得到需要的需要的假定启动子区域 终止位置对应的数字了! 当然,大家可以选择更大的范围(如包含启动子和部分mRNA区域或者全部mRNA区域)进行相关比对和预测,本文只选择了启动子区域进行后续分析。 确定了某一个基因假定启动子区域的起始和终止位置后,点击submit,页面跳转如下: 然后点击页面上部的Synteny/Alignments按钮,如下图所示高亮区域对应按钮:
转录因子是一种具有特殊结构、行使调控基因表达功能的蛋白质分子,也称为反式作用因子。植物中的转录因子分为二种,一种是非特异性转录因子,它们非选择性地调控基因的转录表达,如大麦(Hordeum vulgare) 中的HvCBF2 (C-repeat/DRE binding factor 2) (Xue et al., 2003)。还有一种称为特异型转录因子,它们能够选择性调控某种或某些基因的转录表达。典型的转录因子含有DNA 结合区(DNA-binding domain)、转录调控区(activation domain)、寡聚化位点(oligomerization site) 以及核定位信号(nuclear localization signal) 等功能区域。这些功能区域决定转录因子的功能和特性(Liu et al., 1999)。DNA结合区带共性的结构主要有:1)HTH 和HLH 结构:由两段α-螺旋夹一段β-折叠构成,α-螺旋与β-折叠之间通过β-转角或成环连接,即螺旋-转角-螺旋结构和螺旋-环-螺旋结构。2)锌指结构:多见于TFIII A 和类固醇激素受体中,由一段富含半胱氨酸的多肽链构成。每四个半光氨酸残基或组氨酸残基螯合一分子Zn2+ ,其余约12-13 个残基则呈指样突出,刚好能嵌入DNA 双螺旋的大沟中而与之相结合。3)亮氨酸拉链结构:多见于真核生物DNA 结合蛋白的 C 端,与癌基因表达调控有关。由两段α - 螺旋平行排列构成,其α - 螺旋中存在每隔7 个残基规律性排列的亮氨酸残基,亮氨酸侧链交替排列而呈拉链状,两条肽链呈钳状与DNA 相结合。 同一家族的转录因子之间的区别主要在转录调控区。转录调控区包括转录激活区(transcription activation domain) 和转录抑制区(transcription repression domain) 二种。近年来,转录的激活区被深入研究。它们一般包含DNA结合区之外的30-100个氨基酸残基,有时一个转录因子包含不止一个转录激活区。如控制植物储藏蛋白基因表达的VP1和PvALF转录因子,它们的N-末端酸性氨基酸保守序列都具有转录激活能力,与酵母转录因子GCN4和病毒转录因子的VP16的酸性氨基酸转录激活区有较高同源性(Bobb et al., 1996)。典型的植物转录因子激活区一般富含酸性氨基酸、脯氨酸或谷氨酰胺等,如GBF (G-box binding factor) 含有的GCB盒(GBF conserved box) 激活结构域(lunwen114 and Bevan, 1998)。 转录抑制区也是转录因子调控表达的重要位点,但是对其作用机理研究尚不深入。可能的作用方式有三种:1)与启动子的调控位点结合,阻止其它转录因子的结合;2)作用于其它转录因子,抑制其它因子的作用;3)通过改变DNA的高级结构阻止转录的发生。 转录因子必须在核内作用,才能起到调控表达的目的。因此,转录因子上的核定位序列是其重要的组成部分。一般一个或多个核定位序列在转录因子中不规则分布,同时也存在不含核定位序列的转录因子,它们通过结合到其它转录因子上进入细胞核。核定位序列一般是转录因子中富含精氨酸和赖氨酸残基的区段。目前,水稻中的GT-2、西红柿中的HSFA1-2、玉米的O2和碗豆的PS-IAA4和6等转录因子中的核定位序列都已被鉴定(Boulikas, 1994; Dehesh et al., 1995; Lyck et al., 1997; Varagona et al., 1992; Abel and Theologis, 1995)。 绝大多数转录因子结合DNA前需通过蛋白质-蛋白质相互作用形成二聚体或多聚体。所谓二聚体化就是指两分子单体通过一定的结构域结合成二聚体,它是转录因子结合DNA时最常见的形式。由同种分子形成的二聚体称同二聚体,异种分子间形成的二聚体称异二聚体。这种多聚体的形成是转录因子上的寡聚化位点