
折叠在传统园林里的现代性——以北京2013年园博会设计师广场获奖作品“步移景异”为例
(2012-10-07 09:44:25)
转载▼
分类:发表文章
标签:
中国园林
景观设计
现代性
1引言
长久以来,由于我们深陷“中国传统”与“西方现代”的泥潭,在处理传统与现代性的关系上总是经历曲折与反复,似乎传统与现代,这一对立的关系难以处理,因而极大地影响了中国园林可持续发展相关理论与实践的研究与探索,以至于“创新”需要被当作一种政治口号由班府来加以提倡。为此,本文以北京2013年园博会设计师广场获奖作品“步移景异”为例,试图摆脱这种“中国传统”与“西方现代””的困惑,探讨“中国园林的现代性”这一新的命题,走向“现代中国”,并由此作为在中国语境下的园林现代性或者在全球化语境下的园林中国性,进行一点我们的解读与思考。
2设计构思——折叠与叠加
中国古典园林的传承应该是审美精神和造园手法的传承,学习古典园林艺术的文化内涵,而不是追随其表现形式,简单搬用原型(这方面我们已经有了上世
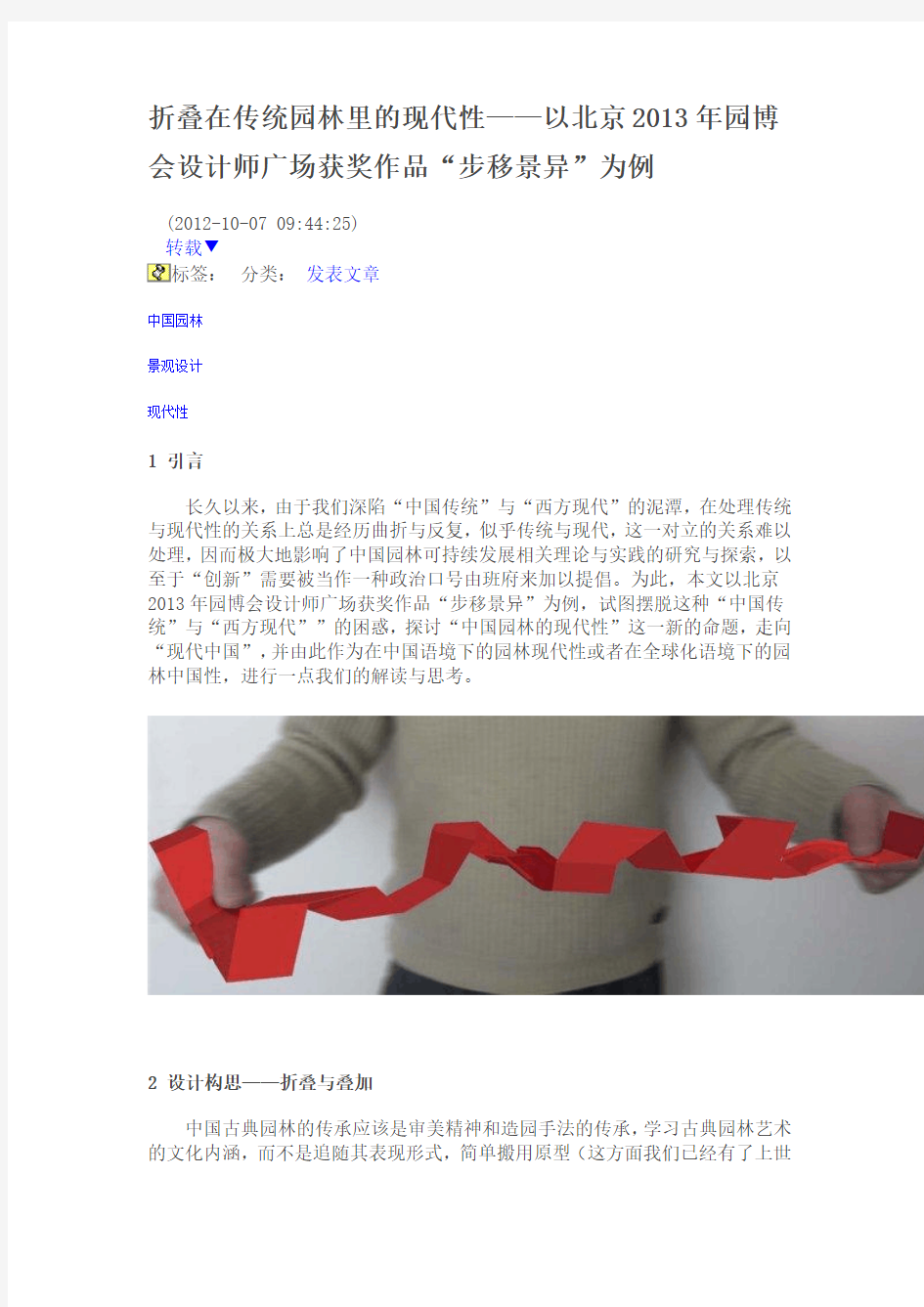
纪80年代园林上大街失败的教训)。要将传统精粹的东西,特别是站在此次北京2013年国际园博会的背景下,对古典园林造诣上的精粹进行推敲、消化、接力与传承,再融合于当代环境背景下,生成高度城市化特质的园林景观。用另一种视角来看待传统瑰宝的遗续,并用另一种整合手法——“园”素的折叠与叠加,来传承开拓(图1)。
(图1)。
古典园林中的游廊——一种狭而长的经典过渡空间形式,它通常孕育了必然的暗示与导向,通过利用人们怀有期待的情绪,巧妙地借用这一形式自然地将人们引导至目的地。本次园博会设计师广场5号地块地处两大景点(锦绣谷和航天主题馆)之间,设计充分借鉴了这一空间营造手法,将传统亭廊布局和组合转换成一种简单的折叠关系,借助于空间的对比:引导与暗示、疏与密、虚与实的组合以及如对景、借景、障景、衬景、框景等一系列传统造景艺术手法的运用,解决主与次、内与外、围与透的关系,从而完成“物理折叠”到“虚拟折叠”的过程,创造富有层次却又相互渗透的流动空间。其次,我们将古典园林的造园要素(铺地、亭廊、小径及水面等)从传统布局和组合关系中拆分解构出来,再按现代景观的空间与视觉手法以及审美及功能要求重新整合重组:新组成的“园林地
毯”成为整个广场景观元素的载体。通过这种折叠与叠加的手段达到的一种整体性大于局部之和的关系,形成新的共享空间,从而满足现代人的多功能与审美需求,所谓用现代的手法讲传统的故事(图2)。
(图2)。
3折叠的进程——古
典园林感知体验模式
的现代诠释
3.1 巧于因借的视域
拓展
中国古典园林,
尤其江南园林,尽管
建造在狭小的空间
中,但并不局限于园
址边界的限定,而是以场地的视觉边界为设计范围,通过巧妙的借景手法来形成完整统一的园林景观。其中,“以有限面积,营无限空间”,强调“小中见大” 是中国古典园林常用的设计手法。本次设计的5号地块面积仅为1000 m2,而且不规则,这与传统园林的用地极其相似。借助借景、衬景、框景等手法,高度抽象后的亭、廊组合,经过折叠不仅起到组织空间的作用,而且加强了空间之间的联系,使各个空间之间相互渗透,形成整一性,同时还起到了扩大空间效果的作用。通过借景形成园内景观与周围环境的融合同时把周围的景观引入园中,这与中国古典园林追求的无限外延的空间视觉效果有异曲同工之妙(图3)。
3.2 欲扬先抑的视觉对比
园林造景绝不会让人一走进门口就看到最好的景色,而最好的景色往往藏在后面。采取抑景的手法透过漏隙,见园内的美景,这叫做“先藏后露”、“欲扬先抑”、“山重水复疑无路,柳暗花明又一村”。围绕这一原则,设计中“Z”字型的亭、廊组合紧密结合对景展开,很好地起到了空间组织与向导作用,它曲折变化的线性与广场上直线小径的穿插形成鲜明的对比,为空间体验增添了丰富情趣,也为游人提供了由开放至私密空间过渡的心理需求。在经由开敞的入口进入景观亭,人群的视线先抑后扬地由局限转而开阔的反复,让有限空间曲折多变,这与市井中造园的根本追求如出一辙(图4)。
(图4)
3.3 步移景异的时空过程
为增强景的深度感和层次感,园林中的主角大多被置于陪衬之后,被藏的主角并非为了不被人所观见,而是在于被观的过程,藏匿是为了更好的显露,在这一辨证关系面前,引导形式的存在便呼之欲出,而借助于空间的组织与向导性俨然能作用在引导与暗示上,正如著名的后现代建筑理论家查尔斯·詹克斯(Charles Jencks)在解释后现代主义的空间形式与本质时,曾举中国园林的例子来说明这种性质:“后现代就像中国园林的空间,把清晰的最终结果悬在半空,以求一种曲径通幽的,永远达不到某种确定目标的‘路线’。”本次设计通过折叠,以一系列庭园或园中园的方式来强调过渡关系,使空间错落有序,层次变化异常丰富,游人在不步移景异的观赏过程中知不觉中被引导至本次园博会的主景点——锦绣谷(图5)。
(图5)
4空间的重组——游廊、小径与园林地毯的叠加
4.1 园林地毯
传统园林中的铺地、水面、植被之间有一定的寓意, 从而存在一种固有关系,在本次设计中,我们试图运用解构的手法将这些元素在布局和空间上加以拆分,再以象征性的符号形式把它们浓缩,以柔和环保的新材料,按照现代人的审美和功能需求重新排列组合。事实上是以排比夸张的手法传递了园林意向,形成独特的中国式拼图。这种以园林铺地中经典元素重新拼接而成的地毯给人以视觉联想,延伸了历史文化内涵,从而唤醒人们对古典园林重新思考的力量。
4.2 亭的张力折叠在游廊的流动之下
本次设计师广场景观设计,如同周旋于偶然的联系与必然的巧合之间。平面上的景观亭收敛为一系列的景点与节点,其位置处地总是精密地位于游人的视觉
场中,俨然为布局中有着强烈吸引力的半隐蔽空间。对门楣的洗涤简练与现代语言化的漏窗剪影则是一种对园林精髓的铺陈总结,从而使传统的意境表达方式更加符合现代人的审美标准。通过将明显差异的两个空间(亭、廊)毗邻地安排在一起,借两者的对比作用从而突出各自的特点。亭、廊线性的曲折变化则紧密结合对景展开,同时曲折变化的韵律使空间的平面层次更易于表现,全方位拓展了视线,使意境得以扩充,从而获得了一种特殊的观赏路径(图6)。
(图6)
4.3亭廊的流动叠加在园林地毯的包容之上
承载亭、廊的园林地毯同时也是直线交叉网路体系,它引导的景观步道游刃于丰富而变化的空间之中,以贯穿整个场地的开敞手笔联系了周边其他设计师广场及主要景点。这些直线小径看似偶然,却已经必然使参观的人流穿行于此,寓偶然于必然之中(图18)。它们是后人对于江南园林的直率表达,以流畅而简明扼要的方式为人们提供了包括水面、水生植物、树木和花卉到景观亭、廊道在
内,相互穿插且不停息的绚丽三维空间的体验,使观赏者在动态观赏中的印象更为深刻(图7)。
(图7)
4.4 园林设计融入现代生活的环境之中
当代中国园林最大的困惑就是传统如何面对现代的挑战。因为毕竟,时代总是为当代人的生存环境服务的,中国古典园林与现代园林的概念已经有了很大的不同:功能和服务的对象与现代人的生活方式相距甚远,同时古典园林所表现的人生哲理和审美情趣也与今天有着很大的距离。为此,本设计力求打破传统园林单一功能的限定性,赋予园林全面新的内涵,使设计师广场成为自由、共享、多功能的载体,在强调观赏者同景观相融相合的同时,满足不同人群的需求的同时,给游客提供了一个自由发挥的舞台。在开放性、整体性、参与性较强的现代城市共享空间中,对景观进行欣赏、品味、聆听,触摸,暇想, 甚至去表演,使观者在停留与观赏之中体会传统与现代的交流(图8, 9)。
(图8、9)
结语
本次设计试图运用抽象概括的设计方法,从空间和元素的特点出发,运用现代园林的设计语汇,对中国古典园林的感知体验模式进行“再”表现:将亭的张力折叠在游廊的流动之下,将亭、廊的流动叠加在园林地毯的包容之上,从而营造一个简约而又不失丰富的场所。那些环保的用材、简明的室外家具、重组构图的力量与节奏,都富有韵律地诠释了对传统园林精粹的传承并彰显其现代性的追求。区别于文人墨客时代的赏析意境,其本质是为了述说对于空间淋漓尽致的表达和在“虽由人作,宛自天开”中窥见的美学更替。虽然这种研究方法难免带有很强的主观性,甚至有失偏颇,但较于常见的“只可意会不可言传”,仅仅停留在文学层面上的探索方法,多了一层形象的表述。这无疑更有助于深入认识中国古典园林的典型特征,并对现代园林设计更有借鉴意义,毕竟形象的东西更能使我们印象深刻。
革命和反传统不应当成为中国园林现代性的标准,而抛弃古典园林的历史局限,把握传统观念的启示意义,融入现代生活的环境需求,在保持民族文化延续基础上,探索中国园林创作的历史机遇才是时代赋予中国园林现代性的必由之路。我们希望的是中国园林的道路并非越走越窄,以致遭误解、被挥斫零落。经典的造园心得要经过融会贯通才能在当今社会中发挥它的内涵价值,而我们这次所做的设计师广场景观的探索,本身也正是对中国园林的理性注解和思考。
古典传统
染色体步移技术(Genome Walking)是一种重要的分子生物学研究技术,使用这种技术可以有效获取与已知序列相邻的未知序列。染色体步移技术主要有以下几方面的应用: 1. 根据已知的基因或分子标记连续步移,获取人、动物和植物的重要调控基因,可以用于研究结构基因的表达调控。如分离克隆启动子并对其功能进行研究; 2. 步查获取新物种中基因的非保守区域,从而获得完整的基因序列; 3. 鉴定T-DNA 或转座子的插入位点,鉴定基因枪转基因法等转基因技术所导致的外源基因的插入位点等; 4. 用于染色体测序工作中的空隙填补,获得完整的基因组序列; 5. 用于人工染色体PAC、YAC 和BAC 的片段搭接。 其主要原理是根据 已知DNA 序列,分别设计三条同向且退火温度较高的特异性引物(SP Primer),与退火温度较低的兼并引物,进行热不对称PCR 反应。 现在有很多Genome Walking Kit,相应的说明书都介绍的很详尽,下面给你发一个原理图:
大致的原理是这样的,依据TaKaRa的试剂盒,具体的操作步骤如下: 1. 基因组DNA 的获取。 基因组DNA 的质量是侧翼序列获取成功与否的关键因素之一。建议不要使用只经过简单处理的基因 组DNA(例如:只进行细胞热处理或蛋白酶处理)作为模板,而要使用经过充分纯化的完整的基因组 DNA。此外,由于本方法灵敏度极高,模板DNA 一定不要污染,所需的DNA 量不要少于3 μg。 2. 已知序列的验证。 在进行PCR 实验之前必须对已知序列进行验证,以确认已知序列的正确性。具体方法为:根据已知序 列设计特异性引物(扩增长度最好不少于500 bp),对模板进行PCR 扩增,然后对PCR 产物进行测 序,再与参考序列比较确认已知序列的正确性。 3. 特异性引物的设计。 根据验证的已知序列,按照前述的特异性引物设计原则设计三条特异性引物,即:SP1、SP2、SP3。 4. 1st PCR 反应。 基因组DNA 经OD 测定准确定量后,取适量作为模板(不同物种的最佳反应DNA 量并不相同,实际 用量参考下面的注*1),以AP Primer(四种中的任意一种,以下以AP1 Primer 为例)作为上游引 物,SP1 Primer 为下游引物,进行1st PCR 反应。
遗 传HEREDIT AS (Beijing )28(5):587~595,2006 专论与综述 收稿日期:20050907;修回日期:20051109 作者简介:刘 博(1979— ),男,黑龙江人,硕士生,专业方向:植物基因工程。Tel :0411287509077;E 2mail :ppic9k @https://www.doczj.com/doc/984250508.html, 通讯作者:安利佳(1955— ),男,教授,博士生导师,研究方向:植物基因工程及基因工程药物。Tel :0411284706365;E 2mail :bioeng @https://www.doczj.com/doc/984250508.html, 应用于染色体步移的PCR 扩增技术的研究进展 刘 博1,2,苏 乔1,汤敏谦2,袁晓东2,安利佳1 (1.大连理工大学环境与生命科学院,大连116023;2.宝生物工程(大连)有限公司,大连116600) 摘 要:各种建立在PCR 基础上染色体步移的方法能够根据已知的基因序列得到侧翼的基因序列。染色体步移技术主要应用于克隆启动子、步查获得新物种中基因的非保守区域、鉴定T 2DNA 或转座子的插入位点、染色体测序工作中的空隙填补,从而获得完整的基因组序列等方面。其方法主要有3种:反向PCR 的方法,连接法介导的PCR 的方法以及特异引物PCR 的方法。文章就各种方法进行举例说明并加以分析比较。关键词:染色体步移;反向PCR ;连接法PCR ;特异引物PCR 中图分类号:Q75 文献标识码:A 文章编号:0253-9772(2006)05-0587-09 Progres s of the PCR Amplification T echniques f or Chromos ome Walking LI U Bo 1,2,SU Qiao 1,T ANG Min 2Qian 2,Y UAN X iao 2Dong 2,AN Li 2Jia 1 (1.School of Environmental and Biological Science and Technology ,Dalian University of Technology ,Dalian 116023,China ;2.Ta KaRa Biotechnology (Dalian )Co.,Ltd.Dalian 116600,China ) Abs t ra ct :Various PCR 2based methods are available for chromosome walking from a known sequence to an unknown region.It is promising in genome 2related re search for the following experiments :promoter cloning ,obtaining non 2con 2servative parts of genes in new species ,identification of T 2DNA or transposon insertion site s and filling in gap s or un 2known chromosome regions in genome sequencing.The se methods consisted of three type s :inverse PCR ,ligation 2me 2diated PCR and specific primer PCR.In this review ,we illustrated and compared the current techniques.Ke y w or ds :chromosome walking ;inverse PCR ;ligation 2mediated PCR ;specific primer PCR 获得与已知序列相邻的未知序列的染色体步移技术是一项重要的分子生物学研究技术。染色体步移技术主要有以下几方面的应用:(1)根据已知的基因或分子标记连续步移获取人、动物和植物的重要调控基因,用于研究结构基因的表达调控,如:分离克隆启动子并对其功能的研究;(2)步查获得新物种中基因的非保守区域,从而获得完整的基因序列;(3)鉴定T 2DNA 或转座子的插入位点,以及鉴定基因枪转基因法等转基因技术所导致的外源基因的插入位点;(4)用于染色体测序工作中的空隙填补,从而获得完整的基因组序列;(5)用于人工染色体PAC 、Y AC 和BAC 的片段搭接。对于基因组测序已 经完成的少数物种(如人、小鼠、线虫、水稻、拟南芥 等)来说,可以轻松地从数据库中找到已知序列的侧翼序列。但是这毕竟只是研究少数模式生物时的情况,对于自然界中种类繁多的其他生物而言,在不知道它们的基因组DNA 序列以前,想要知道一个已知区域两侧的DNA 序列,只能采用染色体步移技术。因而,染色体步移技术在现代分子生物学研究中占有举足轻重的地位,是结构基因组研究以及功能基因组研究的基础。 自从PCR 技术发明以来,人们就在PCR 技术的基础上设计了多种扩增未知序列的染色体步移的方法,这些方法依其原理可分为3类:反向PCR 的
1依赖酶切连接的PCR 这类方法首先对基因组DNA酶切之后,然后让酶切产物自我成环或在酶切产物末端加上相应的接头。以已知序列上的特异引物或接头上的锚定引物,扩增已知序列的上下游侧翼序列。 1.1 反向PCR 反向PCR(inversPCR,IPCR)是Triglia等提出的扩增已知序列上游和下游未知序列的方法。该方法的基本原理是对已知序列进行分析,选择已知序列中没有的限制性内切酶位点,酶切基因组DNA后,酶切片段环化自连,然后利用已知序列设计的反向引物,扩增已知序列两侧的未知序列,原理如图1。因此,这种方法包含以下3个步骤:(1)基因组DNA 酶切;(2)线性酶切产物环化;(3)已知序列处设计的反向引物扩增目标片段。在PCR 扩增之前,也可以根据已知序列用合适的内切酶将环化的DNA切割成线性DNA,这样更利于PCR反应的进行。 图1:反向PCR原理图 最初,IPCR用于扩增基因组DNA的侧翼未知序列,后来利用T4聚合酶补平双链cDNA 末端后再环化,可以有效扩增已知cDNA的侧翼未知序列。然而,IPCR存在技术缺点:(1)环化过程难以控制,基因组DNA酶切后,不同的酶切片段连接成线性串联体,致使非特异性扩增;(2)基因组DNA酶切位点随机分布,可能产生过长的酶切片段导致PCR扩增效率下降,成功率降低。Benkel等在此基础上对IPCR的反应体系改进,可以扩增长达40Kb的片段。Kohda等在对基因组DNA酶切之后,环化连接过程中,酶切位点之间加上一段桥连片段,有效扩增侧翼片段,这种方法称为桥连反向PCR(BI-PCR)。BI-PCR扩增的特异性更高,操作更加简便。最近,Tsaftaris等利用改进的滚环复制反向PCR从植物基因组DNA中成功分离出3Kb的启动子序列。以IPCR为代表的连接成环PCR策略是首次利用PCR技术进行的染色体步移技术。以此为基础,对基因组DNA酶切,在酶切末端加上接头或通过相应的方法在酶切末端加上已知序列,获得目标序列的PCR扩增的染色体步移技术大量涌现。 1.2 载体PCR 载体PCR(single specific primePCR,SSP-PCR)是通过把基因组DNA酶切之后,连接到质 粒载体(如SpBluescript KS)上,并用依据已知序列设计的特异引物和载体上的通用引物特异扩增目标片段的一种PCR染色体步移技术。
来自dxy 22003luocong 植物基因全长克隆几种方法的比较 基因是遗传物质基本的功能单位,分离和克隆目的基因是研究基因结构、揭示基因功能及表达的基础,因此,克隆某个功能基因是生物工程及分子生物学研究的一个重点。经典克隆未知基因的方法比如通过筛选文库等有个共同的弊病, 即实验操作繁琐, 周期较长、工作量繁重,且不易得到全长序列。又由于在不同植物中目的基因mRNA丰度不同,所以获得目的基因的难易程度又不一样,特别是对于丰度比较低的目的基因即使使用不用的方法也不一定能获得成功。近年来随着PCR技术的快速发展和成熟.已经有多种方法可以获得基因的全长序列, 比如经典的RACE技术,染色体步移法和同源克隆法等,本文主要综述几种重要的克隆方法的原理和运用,并且比较分析这几种方法的优缺点,为你的实验节约时间和成本。 1 RACE技术 1985年由美国PE-Cetus公司的科学家Mulis等[1]发明的PCR技术使生命科学得到了飞跃性的发展。1988年Frohman等[2] 在PCR技术的基础上发明了一项新技术, 即cDNA末端快速扩增技术( rapid amplification of cDNA ends, RACE), 其实质是长距PCR( long distance, PCR)。通过PCR由已知的部分cDNA 序列, 获得5′端和3′端完整的cDNA, 该方法也被称为锚定PCR ( anchored PCR) [3] 和单边PCR( one-sidePCR) [4]。RACE技术又分为3?RACE和5?端RACE。3′RACE 的原理是利用mRNA 的3′端天然的poly(A) 尾巴作为一个引物结合位点进行PCR, 以Oligo( dT) 和一个接头组成的接头引物( adaptor primer, AP)反转录mRNA得到加接头的第一链cDNA。然后用一个正向的基因特异性引物( gene-specific primer, GSP) 和一个含有接头序列的引物分别与已知序列区和poly(A) 尾区退火, 经PCR扩增位于已知序列区域和poly( A) 尾区之间的未知序列,若为了防止非特异性条带的产生, 可采用巢式引物( nested primer) 进行第二轮扩增, 即巢式PCR( nested PCR) [5,6]。5?RACE 跟3?RACE原理基本一样,但是相对于3?RACE来说难度较大。 5'-RACE受到诸多因素的影响而常常不能获取全长,因此研究者都着手改进它。这些措施主要是通过逆转录酶、5'接头引物等的改变来实现的,因此出现了包括基于“模板跳转反转录”的SMART RACE技术( switching mechanism at 5′ end of RNA transcript) [7] , 基于5′脱帽和RNA酶连接技术的RLM-RACE技术(RNA ligase mediated RACE)[8], 利用RNA连接酶为cDNA第一链接上寡聚核苷酸接头的SLC RACE技术(single strand ligation to single-stranded cDNA)[9] , 以及以内部环化的cDNA第一链为模板进行扩增的自连接或环化RACE技术(self-ligation RACE or circular RACE)[10],和通过末端脱氧核苷酸转移酶( TdT)加尾后引入锚定引物的锚定RACE技术( anchored RACE)[11]。 笔者主要介绍两种比较新的RACE技术,基于…模板跳转?的SMART RACE 技术和末端脱氧核苷酸转移酶( TdT)加尾技术。 1.1基于‘模板跳转’的SMART RACE技术[7,12]
染色体步移技术(Genome Walking) 染色体步行是一种常用的克隆已知基因旁侧序列的技术。染色体步行是指由生物基因组或基因组文库中的已知序列出发逐步探知其旁邻的未知序列或与已知序列呈线性关系的目的序列的核苷酸。 现有的染色体步行PCR技术包括反向PCR、锅柄PCR、连接介导PCR、热不对称PCR、SON PCR等染色体步移技术主要有以下几方面的应用: 1. 根据已知的基因或分子标记连续步移,获取人、动物和植物的重要调控基因,可以用于研究结构基因的表达调控。如分离克隆启动子并对其功能进行研究; 2. 步查获取新物种中基因的非保守区域,从而获得完整的基因序列; 3. 鉴定T-DNA或转座子的插入位点,鉴定基因枪转基因法等转基因技术所导致的外源基因的插入位点等; 4. 用于染色体测序工作中的空隙填补,获得完整的基因组序列; 5. 用于人工染色体PAC、Y AC 和BAC 的片段搭接。 其主要原理是根据已知DNA 序列,分别设计三条同向且退火温度较高的特异性引物(SP Primer),与退火温度较低的兼并引物,进行热不对称PCR 反应。 现在有很多Genome Walking Kit,相应的说明书都介绍得很详尽,下面给你发一个原理图: 依据TaKaRa的试剂盒,具体的操作步骤如下: 1. 基因组DNA的获取。 基因组DNA的质量是侧翼序列获取成功与否的关键因素之一。建议不要使用只经过简单
组DNA(例如:只进行细胞热处理或蛋白酶处理)作为模板,而要使用经过充分纯化的完整的基因组 DNA。此外,由于本方法灵敏度极高,模板DNA一定不要污染,所需的DNA量不要少于 3 μg。 2. 已知序列的验证。 在进行PCR 实验之前必须对已知序列进行验证,以确认已知序列的正确性。具体方法为:根据已知序 列设计特异性引物(扩增长度最好不少于500 bp),对模板进行PCR 扩增,然后对PCR 产物进行测 序,再与参考序列比较确认已知序列的正确性。 3. 特异性引物的设计。 根据验证的已知序列,按照前述的特异性引物设计原则设计三条特异性引物,即:SP1、SP2、SP3。 4. 1st PCR 反应。 基因组DNA经OD 测定准确定量后,取适量作为模板(不同物种的最佳反应DNA量并不相同,实际 用量参考下面的注*1),以AP Primer(四种中的任意一种,以下以AP1 Primer 为例)作为上游引 物,SP1 Primer 为下游引物,进行1st PCR 反应。 ①按下列组份配制1st PCR反应液。 Template(基因组DNA)x ng *1 dNTP Mixture(2.5 mΜ each )8 μl 10×LA PCR Buffer II(Mg2+ plus) 5 μl TaKaRa LA Taq ?(5 U/μl)0.5 μl AP1 Primer(100 pmol/μl) 1 μl SP1 Primer(10 pmol/μl) 1 μl dH2O up to 50 μl ②1st PCR 反应条件如下: 94℃ 1 min 98℃ 1 min 94℃30 sec 60~68℃*2 1 min 5 Cycles 72℃2~4 min*3 94℃30 sec;25℃ 3 min;72℃2~4 min*3 94℃30 sec;60~68℃*2 1 min;72℃2~4 min*3 94℃30 sec;60~68℃*2 1 min;72℃2~4 min*3 15 Cycles 94℃30 sec;44℃ 1 min;72℃2~4 min*3 72℃10 min 5. 2nd PCR 反应。 将1st PCR反应液稀释1~1000 倍后,取1 μl作为2nd PCR反应的模板,以AP1 Primer 为上游引 物,SP2 Primer为下游引物,进行2nd
螺旋讲堂2010年第4期总第23期 染色体步移 内容概览: 一、染色体步移技术概述 二、常见的染色体步移技术介绍 1、结合基因组文库的染色体步移技术 1) 物理剪切法构建亚克隆文库2) 限制性内切酶法构建亚克隆文库2、基于PCR 技术的染色体步移技术 1) 连接成环PCR 2)外源接头介导PCR (1)连接载体的PCR (2)连接单链接头的PCR (3)连接双链接头PCR 3)半随机引物PCR 策略 (1)新Alu-PCR (2)DW-ACP TM PCR (3)热不对称交错PCR (TAIL-PCR ) 三、染色体步移技术之TAIL-PCR 1、TAIL-PCR 的原理 2、TAIL-PCR 的优点以及技术难点 3、TAIL-PCR 的操作流程 4、TAIL-PCR 中的注意事项 5、常见问题分析 生物人的网上家园 plum 螺旋网HelixNet 染色体步移技术是一种常用的克隆已知片段旁侧序列的技术。本文中主要总结了近年来染色体步移技术的发展情况,介绍了结合基因组文库的染色体步移技术和 基于PCR 的染色体步移技术,并且比较了他们之间的优缺点,最后着重与大家一起 讨论一下TAIL-PCR 的相关内容,希望能对大家有所帮助。
一、染色体步移技术概述 染色体步移(Chromosome walking)又称为基因组步移(Genome walking)是指由生物基因组或基因组文库中的已知序列出发,逐步探知其旁邻的未知序列或与已知序列呈线性关系的目标序列的方法。对于已经完成基因组测序的模式生物种(如人、小鼠、线虫、水稻、拟南芥等),可直接从数据库中轻松的找到已知序列的侧翼序列。但是目前为止,自然界中大多数生物基因组DNA序列仍未知,要想知道一个已知区域两侧的DNA序列,染色体步移无疑是一种非常有效的方法。因此,染色体步移技术在现代分子生物学研究中较为重要,本文主要就染色体步移的相关方法,不同方法之间的比较,着重以TAIL-PCR的原理,实验操作及注意事项等进行讨论。 染色体步移技术的主要应用可归结为以下5个方面: (1)鉴定T—DNA或转座子的插入位点,鉴定转基因技术所导致的外源基因的插入位点; (2)根据基因的已知片段、EST或插入的转座子序列克隆目的基因,分离基因的启动子及调控元件; (3)用于人工染色体PAC、YAC和BAC的片段搭接; (4)构建图位克隆中的重叠群; (5)转化标记辅助育种中的STS或SCAR标记等。 二、常见的染色体步移技术介绍 目前,分离侧翼序列的染色体步移方法主要有两种,一是结合基因组文库为主要手段的染色体步移技术,构建基因组文库进行染色体步移尽管步骤比较繁琐,但是适于长距离步移,可以获得代表某一特定染色体的较长连续区段的重叠基因组克隆群。随着亚克隆文库条件构建条件的优化及测序技术的进步,这种方法也将更加快捷,准确。另一个是基于PCR扩增为主要手段的染色体步移技术。基于PCR扩增为主要手段的染色体步移技术步移距离相对较短,但是操作比较简单,尤其适合于已知一段核苷酸序列的情况下进行的染色体步移。在此基础上人们相继发明了十几种侧翼序列克隆的方法,依据其技术原理,可以将这些方法分为3类:连接成环PCR、外源接头介导PCR和半随机引物PCR。 1、结合基因组文库的染色体步移技术 基因组文库是指生物体全部DNA,经过合适的核酸内切酶消化或机械切割以后,克隆到适当的载体分子中,构成重组体分子群体,然后转化给诸如大肠杆菌这样的寄主菌株进行复制繁殖,如此构建的理论上含有生物体整个基因组全部遗传信息的克隆集合体,称作基因组文库,它是进行基因组学研究的重要技术平台,在基因组物理图谱的构建以及基因的图位克隆中发挥了重要作用。基因组文库的种类主要有入噬菌体文库、酵母人工染色体文库(yeast artificial chromosome,YAC)、P1噬菌体文库和细菌人工染色体文库(bacterial artificial chromosome,BAC)等。其中,BAC文库因其插入片段大、嵌合率低、遗传稳定性好、 易于操作等优点而备受青睐,近年来,小麦、棉花、水稻等越来越多的物种构建了BAC文库。 在基因克隆中,该方法用于鉴定一系列彼此重叠的DNA限制片段,分离序列及表达产物未知的基因,特别 是发育相关的基因。利用基因组文库克隆目的基因,首先要有一个根据目的基因建立起来的遗传分离群体, 找到与目的基因紧密连锁的分子标记,然后用遗传作图将目的基因定位在染色体的特定位置,找到与目的 基因紧密连锁的分子标记。构建含有插人大片段DNA的基因组文库(BAC或YAC),以与目的基因连锁的 分子标记为探针筛选基因组文库,鉴定出分子标记所在的大片段克隆,再以该克隆为染色体步移的起点, 用外侧克隆末端作为探针筛选基因组文库,分离新的重叠克隆用获得的阳性克隆,多次重复上述步骤,逐 渐逼近目的基因,构建目的基因区域的重叠群。随后通过亚克隆文库获得含有目的基因的小片段克隆,最 后通过遗传转化和功能互补验证最终确定目的基因的碱基序列。 目前构建亚克隆文库比较常用的DNA片段化方法主要有两种:物理剪切法和限制性内切酶法。
1、Each allele has a different phenotype ,why(illustrate by example). 位于染色体同一位置的分别控制两种不同性状的基因是等位基因,等位基因之间具有多种关系。比如,果蝇中white位点的存在对红眼的形成是必要的。这个位点是根据无义突变命名的,该突变使果蝇在突变杂合子中具有白色的眼睛。表述野生型和突变型基因时,通常在野生型基因后面加上+。 当等位基因存在时,一个动物可能是携带两种突变基因的杂合子。这种杂合子的表型依赖于每一个突变所遗留下的活性。从本质上讲,两个突变基因间的关系与野生型和突变型间没有区别,一个突变可能都是显性的,也可能部分是显性的。在任何一个遗传位点上并非一定要有一个野生型基因。人类学行系统的比较就提供了一个例子:缺失功能有空白型表示,即O型;但是功能性的A型和B 型是共显性的,并且对O型表现出显性。 2、Conservation of exons and its application. 外显子的保守性可以做为鉴定编码区的基础,即通过确认那些在许多生物体中都存在的序列片段。对于含有这样基因的区域,即在许多生物中这些基因的功能长期被保留下来,这个序列所代表的蛋白质应当有两个特性:它必须有一个开放的阅读框(ORF);在其它生物中很可能存在与它相关的序列。 物种杂交可作为鉴定基因的第一条标准,将来自一定区域的短片段作为探针,通过Southern杂交检测来自不同物种的相关DNA,如果我们发现几个物种中的杂交片段与某一探针相关(探针通常来源于人的DNA),则这个探针就可成为一条基因外显子的候补片段。将这些候补片段进行测序,如果它们有阅读框,则它们就可被用来分离周围的基因组区域;如果它们是一个外显子的一部分,则可以用它来鉴定整条基因,分离相应的cDNA和mRNA,从而最终分离出蛋白质。3、Chromosome walking and its application. 从第一个重组克隆插入片段的一端分离出一个片段作为探针从文库中筛选第二个重组克隆,该克隆插入片段含有与探针重叠的顺序和染色体的其它顺序。从第二个重组克隆的插入片段再分离出末端小片段筛选第三个重组克隆,如此重复,得到一个相邻片段,等于在染色体上移了一步,称为染色体步移。 染色体步移技术是一种重要的分子生物学研究技术,使用这种技术可以有效
1、如何理解结构决定功能? 胸腺嘧啶(T,thymine),5-甲基尿嘧啶; 尿嘌呤(U,Urncil),2,4二氧嘧啶; 尿嘌呤(U,Urncil),2,4二氧嘧啶; 胞嘧啶(C,cytosine),2-氧-4氨基嘧啶; T和U就因为5位上一个甲基的区别,虽都是与A发生互补配对,但是一个是RNA的碱基,一个是DNA的碱基。T在形式上也许就是U的甲基化,经过进化选择,就留下来了,因为甲基化后确实会稳定一些,甲基化也成了一种碱基保护的方式。由于DNA负责遗传信息的传递,所以甲基化的形式就被留下了,而RNA的阶段很短,可能就不那么必要了,还可以省点C源。 C和U因为4位上一个是氨基一个是羰基然后一个就和G配对,一个和A 配对。 1、Each allele has a different phenotype ,why(illustrate by example). ①位于染色体同一位置的分别控制两种不同性状的基因是等位基因,等位基因之间具有多种关系。同一条基因的变异体称为复等位基因,复等位基因的存在使突变体之间能够形成杂合子,这些复等位基因之间的关系有各种形式。除了最简单的情况外,通常一系列等位基因往往具有不同的表型。例如,黑腹果蝇的
w基因座上有一系列的等位基因所表现的从红眼一直到白眼的多种眼色,其颜色从深到浅均有。在黑腹果蝇控制眼色的w基因座的杂合子中,红色(野生型)相对于其他基因来说是显性的,它们有很多不同的突变等位基因,尽管一些突变基因并未产生眼睛颜色,但是一些基因产生了颜色。这种杂合子的表型依赖于每一个突变所遗留下的活性:每一种突变等位基因代表了该基因的不同突变,这些突变不会完全破坏基因的功能,而会保留一定的活性,由此会产生特征性的表型。 ②当等位基因存在时,可能是携带两种突变基因的杂合子,一个突变可能都是显性的,也可能部分是显性的,在任何一个遗传位点上并非一定要有一个野生型基因。例如人类血型系统:缺失功能用空白型即O型;功能性的A型和B型是共显性的,并对O型表现出显性。 2、Conservation of exons and its application. ①外显子就是在DNA序列中被转录成mRNA片段的一段序列; ②外显子序列通常处于编码氨基酸序列的选择压力下,因而它们很少发生变化,且很多改变不影响密码子意义,同时其不利突变可因不利选择而被有效地去除,因而它在进化中具有保守性。 ③许多生物中含有这样功能保守的基因区域,其基因序列所代表的蛋白质应当有两个特性:有一个开放的阅读框;在其它生物中很可能存在与它相关的序列。 ④根据外显子的保守性的特征可以用来分离和鉴定基因,确认那些在许多生物体中都存在的序列片段。物种杂交可作为鉴定基因的第一条标准,将杂交基因的候补片段进行测序,如果它们有阅读框,则它们就可被用来分离周围的基因组区域;如果它们是一个外显子的一部分,则可以用它来鉴定整条基因并最终分离出蛋白质。 3、Chromosome walking and its application. 从第一个重组克隆插入片段的一端分离出一个片段作为探针从文库中筛选出第二个重组克隆,该克隆插入片段含有与探针重叠的顺序和染色体的其它顺序。从第二个重组克隆的插入片段再分离出末端小片段筛选第三个重组克隆,如此重复,得到一个相邻片段,等于在染色体上移了一步,称为染色体步移。 染色体步移技术是一种重要的分子生物学研究技术,使用这种技术可以有效地获取与已知序列相邻的未知序列,即侧翼序列。主要应用有:
基因的染色体定位(一) 关键词:基因定位荧光原位杂交放射杂交体在人类基因组计划(HGP)中有二大部分内容,一是在2005年之前完成对人类基因组DNA约3×109个核苷酸序列的测定,同时完成对基因的染色体定位工作;二是开展基因功能的研究。基因定位与基因序列两者相辅相成,基因染色体的定位既有助于基因序列的测定,又有利于对基因结构和功能的研究,有利于进一步提示生物的遗传信息。基因测序技术,尤其是大规模测序技术的建立,极大地提高了基因测序的速度。到1999年1月25日为止,全世界已测出全部人类基因序列的7.3%,而部分生物,如酵母、大肠杆菌等的序列已全部测定完毕,这样cDNA的染色体定位工作就显得尤为迫切。然而由于目前的基因大规模测序是建立在基因定位的基础之上,而基因组的染色体定位(基因作图)工作跟不上基因测序技术的发展,因而成了限制基因序列测定的主要因素。基因的染色体定位方法多种多样,它可分为基因的遗传作图和物理作图,而物理作图中最主要是荧光原位杂交(FISH)和放射杂交体(RH)二种。本文就目前主要的几种基因作图方法作一介绍。 1荧光原位杂交(FISH) 原位杂交(ISH)原早被用于染色体组型和核酸分布的分析,后来,随着技术的发展,它被用于基因染色体定位的研究,特别是非同位素标记技术的发展,使得ISH的应用日前广泛。目前它已被用于肿瘤的细胞遗传改变、人类的早期发育、核与基因组的分子结构、不同种属间的
基因图谱比较、动物的细胞发生和无数的基因定位研究。用于基因染色体定位的FISH已被称为CO-FISH或COD-FISH (chromosomeorientationanddirectionFISH)1]。 CO-FISH技术是以生物素或地高辛等半抗原为标记物,采用随机引物法或缺口平移法标记DNA探针,将探针变性后即可用于细胞分裂中期或间期细胞核染色体的原位杂交并产生DNA-DNA杂交体,这种杂交体可以被与半抗原有高度亲和力的荧光标记分子所检测到,而这种杂交信号又可以被与荧光分子偶联的单抗所放大。此外,DNA探针也可以直接标以荧光分子,这些荧光分子有:FITC、德克萨斯红(texasred)及最近开发的花青染料(cyaninedyes)等,如果这样,就不需要进行信号检测和放大就可以直接观察DNA-DNA杂交体2]。用于FISH的DNA 底物已经由原先固定的染色体和间期细胞核发展到伸展的单链DNA分子、细胞核及染色体的自然形态等3]。由于FISH技术已经可以在伸展的DNA分子上进行,这样它的分辨率就达到1~2kb,并能在此区域内作图。而传统的在细胞中期和间期进行的FISH只能分别分辨1~5Mb 和50kb左右的区域,分辨率的提高使得更详细的基因结构和基因内重排研究成为可能;FISH对自然状态下细胞和染色体的研究可以阐明核成分的分布并且不会出现象计算机三维重建分析时的干扰现象;同时,还能对二条同源染色体转录活性的核功能进行研究,并可直接观察基因表达过程中核成分的位置。FISH不仅能对固定的标本进行研究,还能对新鲜的标本进行研究,更有报道认为它能分辨单一碱基的替换4]。
染色体步移步骤
染色体步移技术(Genome Walking)是一种重要的分子生物学研究技术,使用这种技术可以有效获取与已知序列相邻的未知序列。染色体步移技术主要有以下几方面的应用: 1. 根据已知的基因或分子标记连续步移,获取人、动物和植物的重要调控基因,可以用于研究结构基因的表达调控。如分离克隆启动子并对其功能进行研究; 2. 步查获取新物种中基因的非保守区域,从而获得完整的基因序列; 3. 鉴定 T-DNA 或转座子的插入位点,鉴定基因枪转基因法等转基因技术所导致的外源基因的插入位点等; 4. 用于染色体测序工作中的空隙填补,获得完整的基因组序列; 5. 用于人工染色体 PAC、YAC 和 BAC 的片段搭接。 其主要原理是根据 已知 DNA 序列,分别设计三条同向且退火温度较高的特异性引物 (SP Primer),与退火温度较低的兼并引物,进行热不对称 PCR 反应。 现在有很多Genome Walking Kit,相应的说明书都介绍的很详尽,下面给你发一个原理图:
大致的原理是这样的,依据TaKaRa的试剂盒,具体的操作步骤如下:1. 基因组 DNA 的获取。
基因组 DNA 的质量是侧翼序列获取成功与否的关键因素之一。建议不要使用只经过简单处理的基因 组 DNA(例如:只进行细胞热处理或蛋白酶处理)作为模板,而要使用经过充分纯化的完整的基因组 DNA。此外,由于本方法灵敏度极高,模板 DNA 一定不要污染,所需的 DNA 量不要少于3 μg。 2. 已知序列的验证。 在进行 PCR 实验之前必须对已知序列进行验证,以确认已知序列的正确性。具体方法为:根据已知序 列设计特异性引物(扩增长度最好不少于500 bp),对模板进行 PCR 扩增,然后对 PCR 产物进行测 序,再与参考序列比较确认已知序列的正确性。 3. 特异性引物的设计。 根据验证的已知序列,按照前述的特异性引物设计原则设计三条特异性引物,即:SP1、SP2、SP3。 4. 1st PCR 反应。 基因组 DNA 经 OD 测定准确定量后,取适量作为模板(不同物种的最佳反应DNA 量并不相同,实际 用量参考下面的注*1),以 AP Primer(四种中的任意一种,以下以 AP1 Primer 为例)作为上游引 物,SP1 Primer 为下游引物,进行 1st PCR 反应。 ①按下列组份配制 1st PCR反应液。
现代微生物学实验技术 马玉超 北京林业大学生物科学与技术学院办公地点:生物楼117;电话:62336016; Email:myc0307@https://www.doczj.com/doc/984250508.html,
实验内容 ?染色体步移法克隆已知序列的侧翼序列?转座子随机突变及目的表型的筛选 ?- 大肠杆菌β半乳糖苷酶的诱导 ?利用SDS-PAGE分析融合蛋白的可溶性?绿色糖单胞菌的发酵及孢外酶的提取
染色体步移法克隆已知序列的侧翼序列 TAIL PCR —TAIL-PCR 牛伯庆
实验目的 1.掌握TAIL-PCR的原理,增强对PCR重要性的认识;1TAIL PCR的原理增强对重要性的认识 2.掌握TAIL-PCR的实验流程,为将来课题研究奠定 2的实验流程为将来课题研究奠定基础。
特异引物序列: Sp1:CGAGCAGATGGTTCTTGG Sp2:TGGCGAGTGAGACGACGAGT Sp3:TGGAGAACTT GTAACCCTTG GTG a hot arbitrary degenerate primers: AD1:5′-TG(A/T)GNAG(A/T)ANC(G/C)AGA-3′; AD2:5AG(A/T)GNAG(A/T)ANCA(A/T)AGG3; 5′-AG(A/T)GNAG(A/T)ANCA(A/T)AGG-3′ AD3:5′-CA(A/T)CGICNGAIA(G/C)GAA-3′; AD4:5TC(G/C)TICGNACIT(A/T)GGA3。 5′-TC(G/C)TICGNACIT(A/T)GGA-3′
TAIL‐PCR:Thermal asymmetric interlaced PCR Liu et al., 1994 al
DNA Walking步移法加速试剂盒(扩增未知侧翼序列的最佳方法) | [<<][>>] 品! 科研人员已经发展了几种无需建立cD NA库或筛选克隆的PCR技术方法,用 于获取与已知序列相连的未知序列。 这些PCR方法包括反向PCR (IPCR), 连接介导PCR (LM-PCR)和随机引物P CR (RP-PCR)。虽然这些方法有一定的 效果并被不断改进,但是仍然受制于 繁琐的酶切,适配体连接 (adaptor ligation)和纯化步骤。而且,RP-PC R的随机引物经常会因为非特异性退 火而产生多余产物。DNA步移法加速 试剂盒通过应用专利的ACP(复性控 制引物)技术,提高了小片断寡聚核 苷酸的特异性,可以克服现有基因步 移法的缺点。 DNA步移法加速试剂盒由PCR酶混合 物与用于捕获未知目标序列的高特异 DNA Walking ACP (DW-ACP)引物组 成。据此优化的PCR反应条件(以下 称为DW ACP-PCR技术)可以获取长达 3kb的未知侧翼序列。 一种全新的扩增未知目标序列的快速 PCR方法。
试剂盒由PCR Master Mix和专为高 特异度捕捉未知靶点的独特DNA步移退火控制引物(DW-ACPs)组成。此最优化的PCR条件,称为 DW ACP-PCR TM 技术,可以获取长达3kb的未知侧区。此试剂盒是直接扩增未知序列的革命性方法。 "Nature 'Product focus-Transgeni cs' September 7, 2006"! 特点: *最简单 此方法无须建库,无须酶切,无须固定,也无须复杂PCR *最快捷 你的目标产物可在一天内获得,此方法超越其他现有方法 *最可靠 只获取目标产物 应用范围: ●基因组步移 - 启动子区域的克隆或者测序 - 基因结构(外显子/内含子结合区) - 已知序列上下游侧翼的扩增 - cDNA步移 - 裂隙填补(Gap Filling)
引物步移法测序 ? 复制网址 ? 新浪微博 ? 豆瓣社区 ? 腾讯微博 ? 开心网 ? 人人网 只看楼主 听说长片段(>500)测序一般采用所谓“引物步移”的方法,根据测序结果设计引物继续walking ,直至测通全长。那么根据测序结果设计一个引物,另一端用什么引物?是用随机引物吗?如果用随机引物的话,这组随机引物怎么设计? 各位高手有没有这方面的资料可以看看,先谢谢了 0票 票数 good good study day day up flameofstar edited on 2006-08-20 13:57 举报 ? ? 【专题讨论】将支气管镜操作进行到底。热烈欢迎上海长海医院呼吸内科黄海东主治医师做客 丁香园。 xrsgjt 2006-11-13 14:14 消息 引用 收藏 分享 测序引物只要一条就可以了 啊。为什么需要两条呢?呵呵。只要
Re:请问什么是步移法 步移法一般认为是由于反向PCR 可用于研究与已知DNA 片段相连的未知染色体序列,因此又被称为染色体步移法。此法所选择的引物虽与已知DNA 序列互补,但两引物3’ 端是反向的。染色体步移技术主要应用于克隆启动子、步查获得新物种中基因的非保守区域、鉴定T-DNA 或转座子的插入位点、染色体测序工作中的空隙填补,从而获得完整的基因组序列 等方面。 screen.width-333)this.width=screen.width-333" width=640 height=404 title="Click to view full 染色体步移法1.JPG (700 X 442)" border=0 align=absmiddle> 克隆步移法 作者:agui674 提交日期:2007-1-22 16:22:00 在基因组测序中,存在着两种不同的战略,即全基因组随机测序战略和以物理图为基础的以大片断克隆为单位的定向测序战略。两者的最大区别在于是否依赖基因组作图。 全基因组随机测序战略主要采用鸟枪法(shotgun )。鸟枪法,也俗称“霰弹法”,是随机先将整个基因组打碎成小片段进行测序,最终利用计算机根据序列之间的重叠关系进行排序和组装,并确定它们在基因组中的正确位置。“鸟枪法”优点是速度快,简单易行,成本较低,可以在较短的时间内通过集中机器和人力的方法获得大量的基因片断。但是用它来测序,最终排序结果的拼接组装比较困难,尤其在部分重复序列较高的地方难度较大。此外有许多序列片段难以定位在确切的染色体上,成为游离片断;同时又会有许多地方由于没有足够的
北京庄盟国际生物基因科技有限公司 ◆KX Genome Walking Kit ZT601--1 ◆目录号 目录号 ZT601 ◆包装 10 次 ◆版本号 2013 06--30 2013--06 仅用于体外 ◆实验室使用,仅用于体外
KX Genome Walking Kit 染色体步移技术简介与应用 染色体步移技术(Genome Walking )是一种重要的分子生物学研究技术,使用这种技术可以有效获取与已知序列相邻的未知序列。 1. 根据已知的基因或分子标记连续步移,获取人、动物和植物的重要调控基因,可以用于研究结构基因的表达调控。如分离克隆启动子并对其功能进行研究; 2. 步查获取新物种中基因的非保守区域,从而获得完整的基因序列; 3. 鉴定T-DNA 或转座子的插入位点,鉴定基因枪转基因法等转基因技术所导致的外源基因的插入位点等; 4. 用于染色体测序工作中的空隙填补,获得完整的基因组序列; 5. 用于人工染色体PAC 、YAC 和BAC 的片段搭接。 对于基因组测序已经完成的少数物种(如人、小鼠、线虫、水稻、拟南芥等)来说,可以轻松地从数据库中找到某物种已知序列的侧翼序列。但是,对于大多数生物而言,在不了解它们的基因组序列以前,想要知道一个已知区域两侧的 DNA 序列,只能采用染色体步移技术。 产品说明 以PCR 技术为基础的染色体步移的主要问题是在预先不了解未知区域序列信息的情况下,如何设计两个特异性引物来扩增未知区域。而传统的染色体步移方法,如:反向 PCR 法、连接接头法等,都有操作复杂、非特异性扩增、连接效率低等弊端。 相对于其它传统方法,本试剂盒具有高效、简便、特异性高、灵敏度高、一次性获得的未知序列较长等特点。 主要原理主要原理是客户根据已知是客户根据已知DNA 序列序列,,分别设计三条同向且退火温度较高的特异性引物(SP Primer ,),与试剂盒中提供的九种经过独特设计的退火温度较低的兼并引物与试剂盒中提供的九种经过独特设计的退火温度较低的兼并引物与试剂盒中提供的九种经过独特设计的退火温度较低的兼并引物((即 ZFP1、ZFP2、ZFP3、ZFP4、ZFP5、ZFP6、ZFP7、ZFP8、ZFP9)进行热不对称 PCR 反应反应。。通常情况下,其中随机选四种至少有一种兼并引物可以与特异性引物之间利用退火温度的差异进行热不对称 PCR 反应,通过三次巢式PCR 反应即可获取已知序列的侧翼序列。如果一次实验获取的长度不能满足实验要求时,还可以根据第一次步移获取的序列信息,继续进行侧翼序列获取。 此外,本试剂盒中还含有 Control DNA 及 Control Primer ,可以方便进行 Control 实验。 产品内容产品内容((10 次量次量)) 1. ZFP1 Primer (10 pmol/μl ) 100μl 2. ZFP2 Primer (10 pmol/μl ) 100μl 3. ZFP3 Primer (10 pmol/μl ) 100μl 4. ZFP4 Primer (10 pmol/μl ) 100μl 5. ZFP5 Primer (10 pmol/μl ) 100μl 6. ZFP6 Primer (10 pmol/μl ) 100μl 7. ZFP7 Primer (10 pmol/μl ) 100μl 8. ZFP8 Primer (10 pmol/μl ) 100μl 9. ZFP9 Primer (10 pmol/μl ) 100μl 10.ZSP1 Primer (10 pmol/μl ) 20μl