
Primer-BLAST是NCBI的引物设计和特异性检验工具。
Primer-Blast介绍
Primer-BLAST,在线设计用于聚合酶链反应(PCR)的特异性寡核苷酸引物。Primer-BLAST可以直接从Blast主页(https://www.doczj.com/doc/4614792957.html,/)找到,或是直接用下面的链接进入:
https://www.doczj.com/doc/4614792957.html,/tools/primer-blast/
这个工具整合了目前流行的Primer3软件,再加上NCBI的 Blast进行引物特异性的验证。Primer-BLAST免除了用另一个站点或工具设计引物的步骤,设计好的引物程序直接用Blast进行引物特异性验证。并且,Primer-BLAST能设计出只扩增某一特定剪接变异体基因的引物–an important feature for PCR protocols measuring tissue specific expression(注:没办法准确的翻译,只好作罢,汗!)。Primer-BLAST有许多改进的功能,这样在选择引物方面比单个的用 Primer3和NCBI BLAST更加准确。
Primer-BLAST的输入
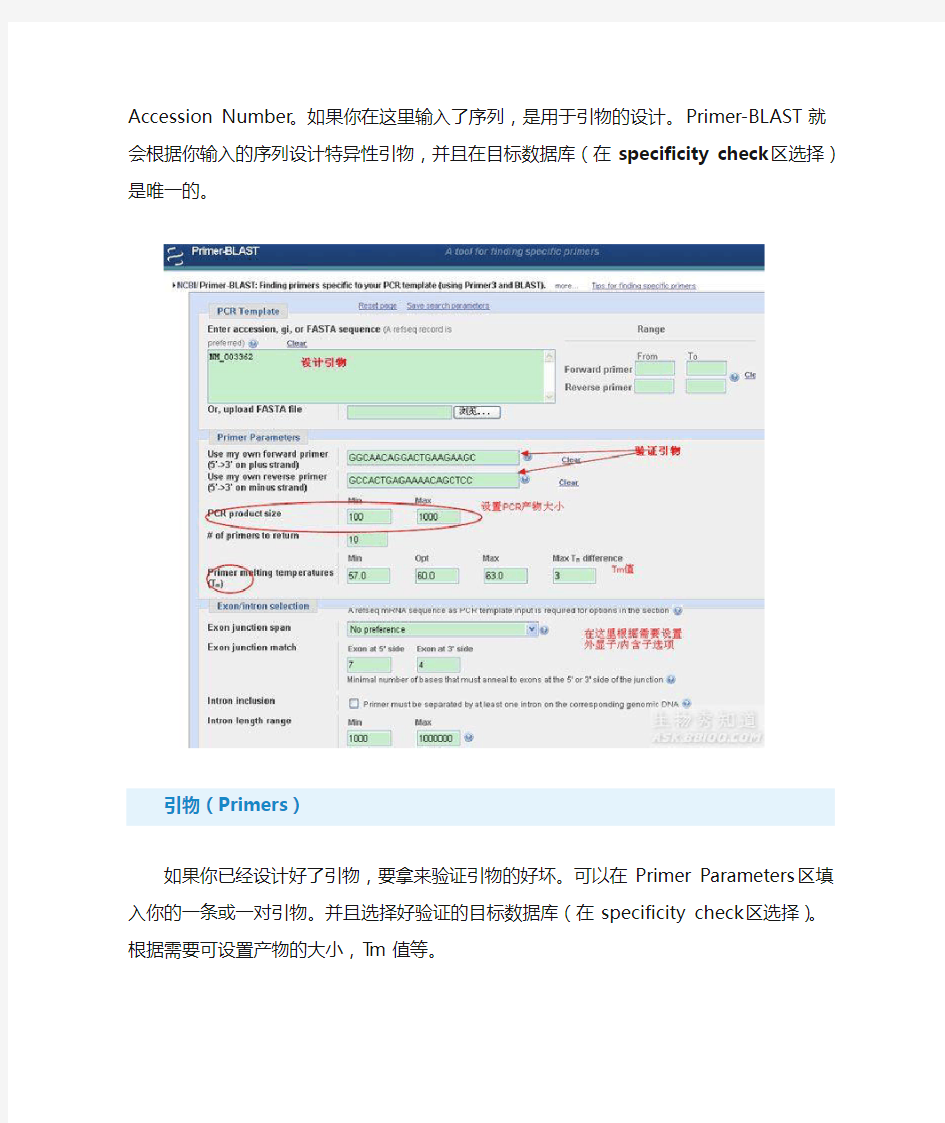
Primer-BLAST界面包括了Primer3和BLAST的功能。提交的界面主要包括三个部分:target template(模板区), the primers(引物区), 和specificity check(特异性验证区)。跟其它的BLAST一样,点击底部的“Advanced parameters”有更多的参数设置。
在“PCR Template”下面的文本框,输入目标模板的序列,FASTA格式或直接用Accession Number。如果你在这里输入了序列,是用于引物的设计。Primer-BLAST 就会根据你输入的序列设计特异性引物,并且在目标数据库(在specificity check区选择)是唯一的。
如果你已经设计好了引物,要拿来验证引物的好坏。可以在Primer Parameters 区填入你的一条或一对引物。并且选择好验证的目标数据库(在specificity check区选择)。根据需要可设置产物的大小,Tm值等。
在specificity check区,选择设计引物或验证引物时的目标数据库和物种。这一步是比较重要的。这里提供了4种数据库:RefSeq mRNA, Genome (selected reference assemblies), Genome (all chromosomes), and nr (the standard non-redundant database)。前两个数据库是经过专家注释的数据,这样可以给出更准确的结果。特别是,当你用NCBI的参考序列作为模板和参考序列数据库
作为标准来设计引物时,Primer-BLAST可以设计出只扩增某一特定剪接变异体基因的特异引物。selected reference assemblies 包括以下的物种: human, chimpanzee, mouse, rat, cow, dog, chicken, zebrafish, fruit fly, honeybee, Arabidopsis, 和 rice。Nr数据库覆盖NCBI所有的物种。
实例分析
用人尿嘧啶DNA糖基化酶(uracil-DNA glycosylase genes, UNG, GeneID: 7374)的两个转录本序列作为一个例子来分析。UNG1的序列长一点(NM_003362),UNG2的序列短一点(NM_080911,注:拿这两个基因的序列ClustalW一下就可以了)。这里用UNG2的序列设计引物,选择RefSeq mRNA database,物种是Human,其它默认。结果如下图A-B所示,设计的引物只能扩增出UNG2。看上面的图,把“Allow primer to amplify mRNA splice variants”这个选项给勾上,出现的结果如下图-C所示,新的引物也可以扩增出UNG1(注:我试了一下,不能得到预期的结果,可能参数没设对)。
Figure. Primer-BLAST results for UNG transcript variant 2. The NCBI Reference sequence NM_080911 was used as a template. Top panel: Primers specific to the single splice variant are reported by default with the mRNA RefSeq database limited to human sequences. Bottom panel: Primers that amplify both splice variants are found with the option to allow splice variants.(点击看大图)
一些Tips
1,在任何时候都要优先使用参考序列的Gi号或Accession 号(尽量不要Fasta 格式的序列)。另外,确保你的序列是最新版本的(在填Accession Number时后面不加版本号就会自动拿最新的序列)
2,就算你对整个序列的某部分感兴趣(如某条染色体上的某个区域),你也应该优化使用Gi号或Accession 号(Primer-BLAST有参数可以设置设计引物的范围,”Form-To”,如上面的第一幅图所示)。因为用Gi号或Accession 号,NCBI会自动读取该序列的一些注释数据,对引物的设计更加有利。
3,尽量使用没有冗除的数据库(如refseq_rna 或 genome database),nr数据库包括了太多的冗除的序列,会干扰引物的设计。
4,请指定一个或几个PCR扩增的目标物种。如果不指定在所有的物种搜索,将会使程序变得很慢,引物的结果也会受其它不相关的物种影响。
参考文献
1. Steve Rozen and Helen J. Skaletsky (2000) Primer3 on the WWW for general users and for biologist programmers. In: Krawetz S, Misener S (eds) Bioinformatics Methods and Protocols: Methods in Molecular Biology. Humana Press, Totowa, NJ, pp 365-386.
来源于 Primer-BLAST:NCBI的引物设计和特异性检验工具 | 柳城博客
N C B I在线B L A S T使用方法与结果详解 IMB standardization office【IMB 5AB- IMBK 08- IMB 2C】
N C B I在线B L A S T使用方法与结果详解 BLAST(BasicLocalAlignmentSearchTool)是一套在蛋白质数据库或DNA数据库中进行相似性比较的分析工具。BLAST程序能迅速与公开数据库进行相似性序列比较。BLAST结果中的得分是对一种对相似性的统计说明。 BLAST采用一种局部的算法获得两个序列中具有相似性的序列。 Blast中常用的程序介绍: 1、BLASTP是蛋白序列到蛋白库中的一种查询。库中存在的每条已知序列将逐一地同每条所查序列作一对一的序列比对。 2、BLASTX是核酸序列到蛋白库中的一种查询。先将核酸序列翻译成蛋白序列(一条核酸序列会被翻译成可能的六条蛋白),再对每一条作一对一的蛋白序列比对。 3、BLASTN是核酸序列到核酸库中的一种查询。库中存在的每条已知序列都将同所查序列作一对一地核酸序列比对。 4、TBLASTN是蛋白序列到核酸库中的一种查询。与BLASTX相反,它是将库中的核酸序列翻译成蛋白序列,再同所查序列作蛋白与蛋白的比对。 5、TBLASTX是核酸序列到核酸库中的一种查询。此种查询将库中的核酸序列和所查的核酸序列都翻译成蛋白(每条核酸序列会产生6条可能的蛋白序列),这样每次比对会产生36种比对阵列。 NCBI的在线BLAST: 下面是具体操作方法 1,进入在线BLAST界面,可以选择blast特定的物种(如人,小鼠,水稻等),也可以选择blast所有的核酸或蛋白序列。不同的blast程序上面已经有了介绍。这里以常用的核酸库作为例子。 2,粘贴fasta格式的序列。选择一个要比对的数据库。关于数据库的说明请看NCBI在线blast数据库的简要说明。一般的话参数默认。 3,blast参数的设置。注意显示的最大的结果数跟E值,E值是比较重要的。筛选的标准。最后会说明一下。 4,注意一下你输入的序列长度。注意一下比对的数据库的说明。 5,blast结果的图形显示。没啥好说的。 6,blast结果的描述区域。注意分值与E值。分值越大越靠前了,E值越小也是这样。7,blast结果的详细比对结果。注意比对到的序列长度。评价一个blast结果的标准主要有三项,E值(Expect),一致性(Identities),缺失或插入(Gaps)。加上长度的话,就有四个标准了。如图中显示,比对到的序列长度为1405,看Identities这一值,才匹配到1344bp,而输入的序列长度也是为1344bp(看上面的图),就说明比对到的序列要长一
一、伺服驱动器外部接线及说明。 SRV-CTRL PLC 外部电源 (DC 24V) SRV-CTRL 说明: 1、SRV-CTRL指PLC的伺服控制模块; 2、以→指向者为输入,以←指向者为输出。 3、各信号含义如下: INH: 禁止输入脉冲指令(开路时禁止);OZ-: Z相输出;OZ+: Z相输出; S-RDY: 伺服驱动器已准备就绪;ALM: 伺服驱动器故障报警; COIN: 定位已完成;SRV-ON: 伺服驱动器“开”信号; COM-: 电源负极;COM+: 电源正极; PULS1: 指令脉冲输入端; PULS2: 指令脉冲输入端; SIGN1: 指令脉冲符号输入端; SIGN2: 指令脉冲符号输入端; 二、参数说明: 1、参数设置方法。
操作面板上共有5个按键,意义如下: MODE:模式转换键,按此键可在4个模式间切换,这4个模式是: DP-××××: 选择监视项目(共有7个, 在按 MODE键显示DP-××××后先按SET,再按↑或↓选择) ⑴、DP-EPS: 位置偏差;⑵、DP-SPD: 转速; ⑶、DP-TRQ: 转矩;⑷、DP-CNT: 控制方式; ⑸、DP-IO: 输入输出信号状态;⑹、DP-ERR: 错误信息; ⑺、DP_NO: PR-××××: 设定参数。 EE-××××: 写入参数。 AT-××××: 自动增益调整。 SET:为设定及确认键。 ↑:数值增加或移动到下一个选项; ↓:数值减少或移动到上一个选项; ←:数位间移动; 具体设置步骤详见有关Drive r的补充信息
说明: 1、参数号码后加“#“者为需要更改的,如02号参数实际应用中应设为 0; 2、控制方式含义 T:转矩控制 S:速度控制 P:位置控制 3、出厂设定为”*”者为出厂时未设置,需根据实际自行设置; 4、其余参数可使用出厂设置; 5、最后一栏为新Driver与之对应的参数(有些没有); 6、更详细的说明参见Driver手册(旧)或参考Driver手册(新)中对 应的参数; 7、以下表格为Driver实际参数设置表。
感谢您使用本产品,本使用操作手册提供LCDA系列伺服驱动器的相关信息。内容包括: ●伺服驱动器和伺服电机的安装与检查 ●伺服驱动器的组成说明 ●试运行操作的步骤 ●伺服驱动器的控制功能介绍与调整方法 ●所有参数说明 ●通讯协议说明 ●检测与保养 ●异常排除 ●应用例解说 本使用操作手册适合下列使用者参考: ●伺服系统设计者 ●安装或配线人员 ●试运行调机人员 ●维护或检查人员 在使用前,请您仔细详读本手册以确保使用上的正确。此外,请将它妥善保存在安全的地点以便随时查阅。下列在您尚未读完本手册时,务必遵守事项: ●安装的环境必须没有水气,腐蚀性气体或可燃性气体。 ●接线时,禁止将三相电源接至马达U、V、W的连接器,因为一旦接错 时将损坏伺服驱动器。 ●接地工程必须确实实施。 ●在通电时,请勿拆解驱动器、马达或更改配线。 ●在通电动作前,请确定紧急停机装置是否随时开启。 ●在通电动作时,请勿接触散热片,以免烫伤。 如果您在使用上仍有问题,请洽询经销商或者本公司客服中心。
安全注意事项 LCDA 系列为一开放型(Open Type )伺服驱动器,操作时须安装于遮蔽式的控制箱内。本驱动器利用精密的回授控制与结合高速运算能力的数字信号处理器(Digital Signal Processor,DSP ),控制IGBT 产生精确的电流输出,用来驱动三相永磁式同步交流伺服马达(PMSM )达到精准定位。 LCDA 系列可使用于工业应用场合上,且建议安装于使用手册中的配线(电)箱环境(驱动器、线材与电机都必须安装于符合环境等级的安装环境最低要求规格)。 在按收检验、安装、配线、操作、维护与检查时,应随时注意以下安全注意事项。 标志[危险]、[警告]与[禁止]代表的含义: ? 意指可能潜藏危险,若未遵守要求可能会对人员造成严 重伤或致命 ? 意指可能潜藏危险,若未遵守可能会对人员造成中度的 伤害,或导致产品严重损坏,甚至故障 ? 意指绝对禁止的行动,若未遵守可能会导致产品损坏, 或甚至故障而无法使用
NCBI(美国国立生物技术信息中心)资源介绍及使用手册 作者:未知来源:中科院上海生命科学研究院生物信息中心时间:2006-12-27 NCBI 资源介绍 本文目录: NCBI(美国国立生物技术信息中心) 简介 NCBI 站点地图 NCBI癌症基因组研究 NCBI-Coffee Break NCBI-基因和疾病 NCBI-UniGene Cluster of Orthologous Groups of proteins (COG)介绍 Gene Expression Omnibus (GEO)介绍 LocusLink介绍 关于RefSeq:NCBI参考序列 NCBI(美国国立生物技术信息中心)简介 介绍 理解自然无声但精妙的关于生命细胞的语言是现代分子生物学的要求。通过只有四个字母来代表DNA化学亚基的字母表,出现了生命过程的语法,其最复杂形式就是人类。阐明和使用这些字母来组成新的“单词和短语”是分子生物学领域的中心焦点。数目巨大的分子数据和这些数据的隐秘而精细的模式使得计算机化的数据库和分析方法成为绝对的必须。挑战在于发现新的手段去处理这些数据的容量和复杂性,并且为研究人员提供更好的便利来获得分析和计算的工具,以便推动对我们遗传之物和其在健康和疾病中角色的理解。 国立中心的建立 后来的参议员Claude Pepper意识到信息计算机化过程方法对指导生物医学研究的重要性,发起了在1988年11月4日建立国立生物技术信息中心(NCBI)的立
法。NCBI是在NIH的国立医学图书馆(NLM)的一个分支。NLM是因为它在创立和维护生物信息学数据库方面的经验被选择的,而且这可以建立一个内部的关于计算分子生物学的研究计划。NCBI的任务是发展新的信息学技术来帮助对那些控制健康和疾病的基本分子和遗传过程的理解。它的使命包括四项任务: 建立关于分子生物学,生物化学,和遗传学知识的存储和分 析的自动系统 实行关于用于分析生物学重要分子和复合物的结构和功能 的基于计算机的信息处理的,先进方法的研究 加速生物技术研究者和医药治疗人员对数据库和软件的使用。 全世界范围内的生物技术信息收集的合作努力。 NCBI通过下面的计划来实现它的四项目的: 基本研究 NCBI有一个多学科的研究小组包括计算机科学家,分子生物学家,数学家,生物化学家,实验物理学家,和结构生物学家,集中于计算分子生物学的基本的和应用的研究。这些研究者不仅仅在基础科学上做出重要贡献,而且往往成为应用研究活动产生新方法的源泉。他们一起用数学和计算的方法研究在分子水平上的基本的生物医学问题。这些问题包括基因的组织,序列的分析,和结构的预测。目前研究计划的一些代表是:检测和分析基因组织,重复序列形式,蛋白domain 和结构单元,建立人类基因组的基因图谱,HIV感染的动力学数学模型,数据库搜索中的序列错误影响的分析,开发新的数据库搜索和多重序列对齐算法,建立非冗余序列数据库,序列相似性的统计显著性评估的数学模型,和文本检索的矢量模型。另外,NCBI研究者还坚持推动与NIH内部其他研究所及许多科学院和政府的研究实验室的合作。 数据库和软件
伺服驱动器参数设置方法 在自动化设备中,经常用到伺服电机,特别是位置控制,大部分品牌的伺服电机都有位置控制功能,通过控制器发出脉冲来控制伺服电机运行,脉冲数对应转的角度,脉冲频率对应速度(与电子齿轮设定有关),当一个新的系统,参数不能工作时,首先设定位置增益,确保电机无噪音情况下,尽量设大些,转动惯量比也非常重要,可通过自学习设定的数来参考,然后设定速度增益和速度积分时间,确保在低速运行时连续,位置精度受控即可。 1.位置比例增益:设定位置环调节器的比例增益。设置值越大,增益越高,刚度越大,相同频率指令脉冲条件下,位置滞后量越小。但数值太大可能会引起振荡或超调。参数数值由具体的伺服系统型号和负载情况确定。 2.位置前馈增益:设定位置环的前馈增益。设定值越大时,表示在任何频率的指令脉冲下,位置滞后量越小位置环的前馈增益大,控制系统的高速响应特性提高,但会使系统的位置不稳定,容易产生振荡。不需要很高的响应特性时,本参数通常设为0表示范围:0~100% 3.速度比例增益:设定速度调节器的比例增益。设置值越大,增益越高,刚度越大。参数数值根据具体的伺服驱动系统型号和负载值情况确定。一般情况下,负载惯量越大,设定值越大。在系统不产生振荡的条件下,尽量设定较大的值。 4.速度积分时间常数:设定速度调节器的积分时间常数。设置值越小,积分速度越快。参数数值根据具体的伺服驱动系统型号和负载情况确定。一般情况下,负载惯量越大,设定值越大。在系统不产生振荡的条件下,尽量设定较小的值。 5.速度反馈滤波因子:设定速度反馈低通滤波器特性。数值越大,截止频率越低,电机产生的噪音越小。如果负载惯量很大,可以适当减小设定值。数值太大,造成响应变慢,可能会引起振荡。数值越小,截止频率越高,速度反馈响应越快。如果需要较高的速度响应,可以适当减小设定值。 6.最大输出转矩设置:设置伺服驱动器的内部转矩限制值。设置值是额定转矩的百分比,任何时候,这个限制都有效定位完成范围设定位置控制方式下定位完成脉冲范围。本参数提供了位置控制方式下驱动器判断是否完成定位的依据,当位置偏差计数器内的剩余脉冲数小于或等于本参数设定值时,驱动器认为定位已完成,到位开关信号为 ON,否则为OFF。 在位置控制方式时,输出位置定位完成信号,加减速时间常数设置值是表示电机从0~2000r/min的加速时间或从2000~0r/min的减速时间。加减速特性是线性的到达速度范围设置到达速度在非位置控制方式下,如果伺服电机速度超过本设定值,则速度到达开关信号为ON,否则为OFF。在位置控制方式下,不用此参数。与旋转方向无关。7.手动调整增益参数 调整速度比例增益KVP值。当伺服系统安装完后,必须调整参数,使系统稳定旋转。首先调整速度比例增益KVP值.调整之前必须把积分增益KVI及微分增益KVD调整至零,然后将KVP值渐渐加大;同时观察伺服电机停止时足否产生振荡,并且以手动方式调整KVP参数,观察旋转速度是否明显忽快忽慢.KVP值加大到产生以上现象时,必须将KVP 值往回调小,使振荡消除、旋转速度稳定。此时的KVP值即初步确定的参数值。如有必要,经KⅥ和KVD调整后,可再作反复修正以达到理想值。 调整积分增益KⅥ值。将积分增益KVI值渐渐加大,使积分效应渐渐产生。由前述对积分控制的介绍可看出,KVP值配合积分效应增加到临界值后将产生振荡而不稳定,如同KVP值一样,将KVI值往回调小,使振荡消除、旋转速度稳定。此时的KVI值即初步确定的参数值。
NCBI (National Center for Biotechnology Information), 美国国家生物技术信息中心 [url]https://www.doczj.com/doc/4614792957.html,/[/url] NCBI是NIH的国立医学图书馆(NLM)的一个分支。 NCBI提供检索的服务包括: 1.GenBank(NIH遗传序列数据库):一个可以公开获得所有的DNA序列的注释过的收集。GenBank是由NCBI受过分子生物学高级训练的工作人员通过来自各个实验室递交的序列和同国际核酸序列数据库(EMBL和DDBJ)交换数据建立起数据库的。它同日本和欧洲分子生物学实验室的DNA数据库共同构成了国际核酸序列数据库合作。这三个组织每天交换数据。其中的数据以指数形式增长,最近的数据为它已经有来自47000个物种的30亿个碱基。 2.Molecular Databases(分子数据库): Nucleotide Sequence(核酸序列库):从NCBI其他如Genbank数据库中收集整理核酸序列,提供直接的检索。 Protein Sequence (蛋白质序列库):与核酸类似,也是从NCBI多个不同资源中编译整理的,方便研究者的直接查询。 Structure(结构)-——关于NCBI结构小组的一般信息和他们的研究计划,另外也可以访问三维蛋白质结构的分子模型数据库(MMDB)和用来搜索和显示结构的相关工具。MMDB:分子模型数据库—一个关于三维生物分子结构的数据库,结构来自于X-ray晶体衍射和NMR色谱分析。 Taxonomy(分类学)——NCBI的分类数据库,包括大于7万余个物种的名字和种系,这些物种都至少在遗传数据库中有一条核酸或蛋白序列。其目的是为序列数据库建立一个一致的种系发生分类学。 3.Literature Databases(文献数据库) (1)PubMed是NLM提供的一项服务,能够对MEDLINE上超过1200万条的上世纪六十年代中期至今的杂志引用和其他的生命科学期刊进行访问,并可以连接到参与的出版商网络站点的全文文章和其他相关资源。 (2)PMC/PubMed Center:也是NLM的生命科学期刊文献的数字化存储数据库,用户可以免费获取PMC的文章全文,除了部分期刊要求对近期的文章付费。 (3)OMIM(孟德尔人类遗传):有关人类基因和无序基因的目录数据库由Victor A.McKusick和他的同事共同创造和编辑的,由NCBI网站负责开发,其中也包括对MEDINE众多资源和Entrez系统的序列记录,以及NCBI中其他有关资源的链接。
一步一步教你使用NCBI 查找DNA、mRNA、cDNA、Protein、promoter、引物设计、BLAST序列比对等 作者:urbest 2007-8-1 苏州大学生命科学学院
最近看到很多战友在论坛上询问如何查询基因序列、如何进行引物设计、如何使用BLAST进行序列比对……,这些问题在NCBI上都可以方便的找到答案。现在我就结合我自己使用NCBI的一些经历(经验)跟大家交流一下BCBI的使用。希望大家都能发表自己的使用心得,让我们共同进步! 我分以下几个部分说一下NCBI的使用: Part one 如何查找基因序列、mRNA、Promoter Part two 如何查找连续的mRNA、cDNA、蛋白序列 Part three 运用STS查找已经公布的引物序列 Part four 如何运用BLAST进行序列比对、检验引物特异性 特别感谢本版版主,将这个帖子置顶! 从发帖到现在,很多战友对该帖给与了积极的关注,在此向给我投票的(以及想给我投票却暂时不能投票的)各位战友表示真诚的感谢,谢谢各位战友! 请大家对以下我发表的内容提出自己的意见。关于NCBI其他方面的使用也请水平较高的战友给予补充 First of all,还是让我们从查找基因序列开始。 第一部分 利用Map viewer查找基因序列、mRNA序列、 启动子(Promoter) 下面以人的IL6(白细胞介素6)为例讲述一下具体的操作步骤 1.打开Map viewer页面,网址为:https://www.doczj.com/doc/4614792957.html,/mapview/index.html 在search的下拉菜单里选择物种,for后面填写你的目的基因。操作完毕如图所示: 2.点击“GO”出现如下页面:
NCBI在线BLAST使用方法与结果详解 BLAST(Basic Local Alignment Search Tool)是一套在蛋白质数据库或DNA 数据库中进行相似性比较的分析工具。BLAST程序能迅速与公开数据库进行相似性序列比较。BLAST结果中的得分是对一种对相似性的统计说明。 BLAST 采用一种局部的算法获得两个序列中具有相似性的序列。 Blast中常用的程序介绍: 1、BLASTP是蛋白序列到蛋白库中的一种查询。库中存在的每条已知序列将逐一地同每条所查序列作一对一的序列比对。 2、BLASTX是核酸序列到蛋白库中的一种查询。先将核酸序列翻译成蛋白序列(一条核酸序列会被翻译成可能的六条蛋白),再对每一条作一对一的蛋白序列比对。 3、BLASTN是核酸序列到核酸库中的一种查询。库中存在的每条已知序列都将同所查序列作一对一地核酸序列比对。 4、TBLASTN是蛋白序列到核酸库中的一种查询。与BLASTX相反,它是将库中的核酸序列翻译成蛋白序列,再同所查序列作蛋白与蛋白的比对。 5、TBLASTX是核酸序列到核酸库中的一种查询。此种查询将库中的核酸序列和所查的核酸序列都翻译成蛋白(每条核酸序列会产生6条可能的蛋白序列),这样每次比对会产生36种比对阵列。 NCBI的在线BLAST:下面是具体操作方法 1,进入在线BLAST界面,可以选择blast特定的物种(如人,小鼠,水稻等),也可以选择blast所有的核酸或蛋白序列。不同的blast程序上面已经有了介绍。这里以常用的核酸库作为例子。
2,粘贴fasta格式的序列。选择一个要比对的数据库。关于数据库的说明请看NCBI在线blast数据库的简要说明。一般的话参数默认。 3,blast参数的设置。注意显示的最大的结果数跟E值,E值是比较重要的。筛选的标准。最后会说明一下。
NCBI资源介绍及使用手册 NCBI资源介绍 本文目录: NCBI(美国国立生物技术信息中心) 简介 NCBI站点地图 NCBI癌症基因组研究 NCBI-Coffee Break NCBI-基因和疾病 NCBI-UniGene Cluster of Orthologous Groups of proteins(COG)介绍 Gene Expression Omnibus (GEO)介绍 LocusLink介绍 关于RefSeq:NCBI参考序列 NCBI(美国国立生物技术信息中心)简介 介绍 理解自然无声但精妙的关于生命细胞的语言是现代分子生物学的要求。通过只有四个字母来代表DNA化学亚基的字母表,出现了生命过程的语法,其最复杂形式就是人类。阐明和使用这些字母来组成新的“单词和短语”是分子生物学领域的中心焦点。数目巨大的分子数据和这些数据的隐秘而精细的模式使得计算机化的数据库和分析方法成为绝对的必须。挑战在于发现新的手段去处理这些数据的容量和复杂性,并且为研究人员提供更好的便利来获得分析和计算的工具,以便推动对我们遗传之物和其在健康和疾病中角色的理解。 国立中心的建立 后来的参议员Claude Pepper意识到信息计算机化过程方法对指导生物医学研究的重要性,发起了
在1988年11月4日建立国立生物技术信息中心(NCBI)的立法。NCBI是在NIH的国立医学图书馆(NLM)的一个分支。NLM是因为它在创立和维护生物信息学数据库方面的经验被选择的,而且这可以建立一个内部的关于计算分子生物学的研究计划。NCBI的任务是发展新的信息学技术来帮助对那些控制健康和疾病的基本分子和遗传过程的理解。它的使命包括四项任务: 建立关于分子生物学,生物化学,和遗传学知识的存储和分析的自动系统 实行关于用于分析生物学重要分子和复合物的结构和功能的基于计算机的信息处理的,先进方法的研究 加速生物技术研究者和医药治疗人员对数据库和软件的使用。 全世界范围内的生物技术信息收集的合作努力。 NCBI通过下面的计划来实现它的四项目的: 基本研究 NCBI有一个多学科的研究小组包括计算机科学家,分子生物学家,数学家,生物化学家,实验物理学家,和结构生物学家,集中于计算分子生物学的基本的和应用的研究。这些研究者不仅仅在基础科学上做出重要贡献,而且往往成为应用研究活动产生新方法的源泉。他们一起用数学和计算的方法研究在分子水平上的基本的生物医学问题。这些问题包括基因的组织,序列的分析,和结构的预测。目前研究计划的一些代表是:检测和分析基因组织,重复序列形式,蛋白domain和结构单元,建立人类基因组的基因图谱,HIV感染的动力学数学模型,数据库搜索中的序列错误影响的分析,开发新的数据库搜索和多重序列对齐算法,建立非冗余序列数据库,序列相似性的统计显著性评估的数学模型,和文本检索的矢量模型。另外,NCBI研究者还坚持推动与NIH内部其他研究所及许多科学院和政府的研究实验室的合作。 数据库和软件 在1992年10月,NCBI承担起对GenBank DNA序列数据库的责任。NCBI受过分子生物学高级训练的工作人员通过来自各个实验室递交的序列和同国际核酸序列数据库(EMBL和DDBJ)交换数据建立起数据库。同美国专利和商标局的安排使得专利的序列信息也被整合。 GenBank是NIH遗传序列数据库,一个所有可以公开获得的DNA序列的注释过的收集。GenBank同日本和欧洲分子生物学实验室的DNA数据库共同构成了国际核酸序列数据库合作。这三个组织每天交换数据。 GenBank以指数形式增长,核酸碱基数目大概每14个月就翻一个倍。最近,GenBank拥有来自47,000个物种的30亿个碱基。 孟德尔人类遗传(OMIM),三维蛋白质结构的分子模型数据库(MMDB),唯一人类基因序列集合
怎么使用NCBI (National Center for Biotechnology Information), 美国国家生物技术信息中心 [url][/url] NCBI是NIH的国立医学图书馆(NLM)的一个分支。 NCBI提供检索的服务包括: 1.GenBank(NIH遗传序列数据库):一个可以公开获得所有的DNA序列的注释过的收集。GenBank是由NCBI受过分子生物学高级训练的工作人员通过来自各个实验室递交的序列和同国际核酸序列数据库(EMBL和DDBJ)交换数据建立起数据库的。它同日本和欧洲分子生物学实验室的DNA数据库共同构成了国际核酸序列数据库合作。这三个组织每天交换数据。其中的数据以指数形式增长,最近的数据为它已经有来自47000个物种的30亿个碱基。 2.Molecular Databases(分子数据库): Nucleotide Sequence(核酸序列库):从NCBI其他如Genbank数据库中收集整理核酸序列,提供直接的检索。 Protein Sequence (蛋白质序列库):与核酸类似,也是从NCBI多个不同资源中编译整理的,方便研究者的直接查询。 Structure(结构)-——关于NCBI结构小组的一般信息和他们的研究计划,另外也可以访问三维蛋白质结构的分子模型数据库(MMDB)和用来搜索和显示结构的相关工具。MMDB:分子模型数据库—一个关于三维生物分子结构的数据库,结构来自于X-ray晶体衍射和NMR色谱分析。 Taxonomy(分类学)——NCBI的分类数据库,包括大于7万余个物种的名字和种系,这些物种都至少在遗传数据库中有一条核酸或蛋白序列。其目的是为序列数据库建立一个一致的种系发生分类学。 3.Literature Databases(文献数据库) (1)PubMed是NLM提供的一项服务,能够对MEDLINE上超过1200万条的上世纪六十年代中期至今的杂志引用和其他的生命科学期刊进行访问,并可以连接到参与的出版商网络站点的全文文章和其他相关资源。 (2)PMC/PubMed Center:也是NLM的生命科学期刊文献的数字化存储数据库,用户可以免费获取PMC的文章全文,除了部分期刊要求对近期的文章付费。 (3)OMIM(孟德尔人类遗传):有关人类基因和无序基因的目录数据库由Victor A.McKusick 和他的同事共同创造和编辑的,由NCBI网站负责开发,其中也包括对MEDINE众多资源和Entrez系统的序列记录,以及NCBI中其他有关资源的链接。
NCBI使用方法 NCBI (National Center for Biotechnology Information), 美国国家生物技术信息 中心 [url]https://www.doczj.com/doc/4614792957.html,/[/url] NCBI是NIH的国立医学图书馆(NLM)的一个分支。 NCBI提供检索的服务包括: 1.GenBank(NIH遗传序列数据库):一个可以公开获得所有的DNA序列的注释过的收集。GenBank是由NCBI受过分子生物学高级训练的工作人员通过来自各个实验室递交的序列和同国际核酸序列数据库(EMBL和DDBJ)交换数据建立起数据库的。它同日本和欧洲分子生物学实验室的DNA数据库共同构成了国际核酸序列数据库合作。这三个组织每天交换数据。其中的数据以指数形式增长,最近的数据为它已经有来自47000个物种的30亿个碱基。 2.Molecular Databases(分子数据库): Nucleotide Sequence(核酸序列库):从NCBI其他如Genbank数据库中收集整理核 酸序列,提供直接的检索。 Protein Sequence (蛋白质序列库):与核酸类似,也是从NCBI多个不同资源中编 译整理的,方便研究者的直接查询。 Structure(结构)-——关于NCBI结构小组的一般信息和他们的研究计划,另外也可以访问三维蛋白质结构的分子模型数据库(MMDB)和用来搜索和显示结构的相关工具。MMDB:分子模型数据库—一个关于三维生物分子结构的数据库,结构来自于X-ray晶体衍 射和NMR色谱分析。 Taxonomy(分类学)——NCBI的分类数据库,包括大于7万余个物种的名字和种系,这些物种都至少在遗传数据库中有一条核酸或蛋白序列。其目的是为序列数据库建立一个一 致的种系发生分类学。 3.Literature Databases(文献数据库) (1)PubMed是NLM提供的一项服务,能够对MEDLINE上超过1200万条的上世纪六十年代中期至今的杂志引用和其他的生命科学期刊进行访问,并可以连接到参与的出版商网络 站点的全文文章和其他相关资源。 (2)PMC/PubMed Center:也是NLM的生命科学期刊文献的数字化存储数据库,用户可以免费获取PMC的文章全文,除了部分期刊要求对近期的文章付费。 (3)OMIM(孟德尔人类遗传):有关人类基因和无序基因的目录数据库由Victor A.McKusick和他的同事共同创造和编辑的,由NCBI网站负责开发,其中也包括对MEDINE 众多资源和Entrez系统的序列记录,以及NCBI中其他有关资源的链接。 (4)Books:NCBI的书库不断收集生物医学方面的书籍,提供这些书籍的出版信息、摘要、目录和全文的连接,用户可以直接在检索文本框内输入一个观念就可以查询。 4.NCBI提供的附加的软件工具有:
无刷驱动器DBLS-02 一概述: 本控制驱动器为闭环速度型控制器,采用最近型IGBT和MOS功率器,利用直流无刷电机的霍尔信号进行倍频后进行闭环速度控制,控制环节设有PID速度调节器,系统控制稳定可靠,尤其是在低速下总能达到最大转矩,速度控制范围150~10000rpm。 二产品特征: 1、PID速度、电流双环调节器 2、高性能低价格 3、20KHZ斩波频率 4、电气刹车功能,使电机反应迅速 5、过载倍数大于2,在低速下转矩总能达到最大 6、具有过压、欠压、过流、过温、霍尔信号非法等故障报警功能 三电气指标 标准输入电压:24VDC~48VDC,最大电压不超过60VDC。最大输入过载保护电流:15A、30A两款连续输出电流:15A 加速时间常数出厂值:秒其他可定制 四端子接口说明: 1、电源输入端: 引角序号引角名中文定义 1V+直流+24~48VDC输入 2GND GND输入 引角序号引角名中文定义 1MA电机A相 2MB电机B相
3MC电机C相 4GND地线 5HA霍尔信号A相输入端 6HB霍尔信号B相输入端 7HC霍尔信号C相输入端 8+5V霍尔信号的电源线 G ND:信号地F/R:正、反转控制,接GND反转,不接正转,正反转切换时,应先关断EN E N:使能控制:EN接地,电机转(联机状态),EN不接,电机不转(脱机状态)B K:刹车控制:当不接地正常工作,当接地时,电机电气刹车,当负载惯量较大时,应采用脉宽信号方式,通过调整脉宽幅值来控制刹车效果。S V ADJ:外部速度衰减:可以衰减从0~100%,当外部速度指令接时,通过该电位器可以调速试机P G:电机速度脉冲输出:当极对数为P时,每转输出6P个脉冲(OC门输入)A LM:报警输出:当电路处于报警状态时,输出低电平(OC门输出)+5V:调速电压输出,可用电位器在SV和GND形成连续可调内置电位器:调节电机速度增益,可以从0~100%范围内调速。 五驱动器与无刷电机接线图 六机械安装:
一步一步教你使用 NCBI 查找DNA、mRNA、cDNA、... 最近看到很多战友在论坛上询问如何查询基因序列、如何进行引物设计、如何使用BLAST 进行序列比对……,这些问题在 NCBI 上都可以方便的找到答案。现在我就结合我自 己使用 NCBI的一些经历(经验)跟大家交流一下 BCBI 的使用。希望大家都能发表自己的使 用心得,让我们共同进步! 我分以下几个部分说一下 NCBI 的使用: Part one 如何查找基因序列、mRNA、Promoter Part two 如何查找连续的 mRNA、cDNA、蛋白序列 Part three 运用 STS 查找已经公布的引物序列 Part four 如何运用 BLAST 进行序列比对、检验引物特异性 特别感谢本版版主,将这个帖子置顶! 从发帖到现在,很多战友对该帖给与了积极的关注,在此向给我投票的(以及想给我 投票却暂时不能投票的)各位战友表示真诚的感谢,谢谢各位战友! 请大家对以下我发表的内容提出自己的意见。关于NCBI 其他方面的使用也请水平较高 的战友给予补充 First of all,还是让我们从查找基因序列开始。 第一部分利用Map viewer 查找基因序列、mRNA 序列、 启动子(Promoter) 下面以人的 IL6(白细胞介素 6)为例讲述一下具体的操作步骤 1.打开Map viewer 页面,网址为: https://www.doczj.com/doc/4614792957.html,/mapview/index.html 在 search 的下拉菜单里选择物种,for 后面填写你的目的基因。操作完毕如图所示:
2.点击“GO”出现如下页面: 3.在步骤二图示的右下角有一个Quick Filter,下面是让你选择的几个复选框,在Gene 前面的小方框里打勾,然后点击Filter. 出现下图:
气压式血液循环驱动器概述 (一)原理 气压式血液循环驱动器用于在进行间歇式气动压迫的过程中,充气压力带通过压迫肢体从而增强静脉血液的流动。在完一次压迫过程之后,主机将对静脉血管再次充满血液的时间进行测量,从而在经过相应时间段的等待之后,重新启动下一次压迫过程。从而有助于防止出现深静脉血栓和肺部栓塞(DVT/PE)。 (二)适应证 1、高风险手术全髋关节置换术,全膝关节置换术,髋关节骨折。 2、存在发生DVT风险的无禁忌证的患者。 3、可用于对抗凝治疗有禁忌的患者(如神经外科手术、头部创伤的患者等)。 (三)禁忌证 1.任何有可能妨碍充气压力带作用的腿局部情况,例如:皮炎,静脉结扎(在手术后即刻),坏疽或者刚做完皮肤移植手术。 2.严重的动脉硬化症或其他缺血性血管病。 3.腿部大范围水肿或由充血性心力衰竭引发的肺部水肿。 4.腿部严重畸形。 5.疑似已出现深静脉血栓。 (四)尺寸的选择 腿长<55. 9cm,选择小号充气压力带。 腿长在55. 9~71. 7cm之间,选择中号充气压力带。 腿长>71. 1 cm,选择大号充气压力带。 (五)人员资格 1.具有执业资格证书的护士。 2.经过“气压式血液循环驱动器护理指南”培训合格的护士。 (六)养护要点 1.使用时动作轻柔,保证管路畅通。 2.及时收回,放置固定地点保存。 3.将充气压力带、连接管整理好,管路勿打折保存。 4.发现污垢及时清理干净。 5.如使用过程中发现异常及时送专业维修部门进行维修。 (七)评估要点 1.评估患者患肢伤口情况、精神状况及配合程度。 2.评估患肢是否有深静脉血栓。 3.评估患者皮肤是否有破损。 (八)宣教要点 1.告知患者使用气压式血液循环驱动器的作用和意义。 2.患者治疗过程中如有不适,及早通知护士。
BLDC无刷电机驱动器 (UB510) 使用手册w w w.u p u ru.c o m
感谢您使用本产品,本使用操作手册提供UB510驱动器的配置、调试、控制相关信息。内容包括。 l驱动器和电机的安装与检查 l试转操作步骤 l驱动器控制功能介绍及调整方法 l检测与保养 l异常排除 本使用操作手册适合下列使用者参考 l安装或配线人员 l试转调机人员 l维护或检查人员 在使用之前,请您仔细详读本手册以确保使用上的正确。此外,请将它妥善放置在安全的地点以便随时查阅。下列在您尚未读完本手册时,请务必遵守事项: l安装的环境必须没有水气,腐蚀性气体及可燃性气体 l接线时禁止将电源接至电机 U、V、W 的接头,一旦接错时将损坏驱动器 l在通电时,请勿拆解驱动器、电机或更改配线 l在通电运作前,请确定紧急停机装置是否随时启动 l在通电运作时,请勿接触散热片,以免烫伤 警告: 驱动器用于通用工业设备。要注意下列事项: (1).为了确保正确操作,在安装、接线和操作之前必须通读操作说明书。 (2).勿改造产品。 (3).当在下列情况下使用本产品时,应该采取有关操作、维护和管理的相关措施。在这种情况下,请与我们联系。 ①用于与生命相关的医疗器械。 ②用于可能造成人身安全的设备,例如:火车或升降机。 ③用于可能造成社会影响的计算机系统 ④用于有关对人身安全或对公共设施有影响的其他设备。 (4).对用于易受震动的环境,例如:交通工具上操作,请咨询我们。 (5).如未按上述要求操作,造成直接或间接损失,我司将不承担相关责任。
1概述 本公司研发生产的BLDC驱动器是一款高性能,多功能,低成本的带霍尔传感器直流无刷驱动器。全数字式设计使其拥有灵活多样的输入控制方式,极高的调速比,低噪声,完善的软硬件保护功能,驱动器可通过串口通信接口与计算机相连,实现PID参数调整,保护参数,电机参数,加减速时间等参数的设置,还可进行IO输入状态,模拟量输入,告警状态及母线电压的监视。 1.1驱动器参数列表 输入电压DC18V-50V 工作电流<=10A 电机霍尔类型60度,300度,120度,240度 工作模式霍尔速度闭环 调速方式0-5V模拟量输入, 0-100%PWM输入(PWM频率范围:1KHz-20KHz) 内部给定, 多段速1, 多段速2, 调速范围0—6000RPM 保护功能l短路:当异常电流大于50A时,产生短路保护 l过流:当电流超过工作电流设置值并持续一设定时间 后产生过流保护 l过压:当电压超过55V时产生过电压保护。 l欠压:当电压低于18V时产生欠电压保护。 l霍尔异常:包括相位异常及值异常. 工作环境场合:无腐蚀性,易燃,易爆,导电的气体,液体,粉尘 温度:-10-55 ℃(无冻霜) 湿度:小于90%RH(不结露) 海拔:小于1000m 振动:小于0.5G, 10hz—60hz(非连续运行) 防护等级:IP21 散热方式自然风冷 尺寸大小120*76*33 单位:mm 重量250g 1.2特点 l速度PID闭环控制,低速转矩大, l调速范转宽,0-6000RPM l运行加减速时间可由软件设定,实现平滑柔和运行。 l驱动器自身损耗小,效率高,温升低,因此体积小,易安装 l多种速度控制方式,由软件设定。 l使能,方向,刹车输入信号的极性可由软件设定 l多种完善的保护功能。 l内置刹车电阻及控制电路(可选),用于消耗再生能量,防止过电压。 2接口定义与连接图
交流伺服电机驱动器使用说明书 1.特点 ●16位CPU+32位DSP三环(位置、速度、电流)全数字化控制 ●脉冲序列、速度、转矩多种指令及其组合控制 ●转速、转矩实时动态显示 ●完善的自诊断保护功能,免维护型产品 ●交流同步全封闭伺服电机适应各种恶劣环境 ●体积小、重量轻 2.指标 ●输入电源三相200V -10%~+15% 50/60HZ ●控制方法IGBT PWM(正弦波) ●反馈增量式编码器(2500P/r) ●控制输入伺服-ON 报警清除CW、CCW驱动、静止 ●指令输入输入电压±10V ●控制电源DC12~24V 最大200mA ●保护功能OU LU OS OL OH REG OC ST CPU错误,DSP错误,系统错误 ●通讯RS232C ●频率特性200Hz或更高(Jm=Jc时) ●体积L250 ×W85 ×H205 ●重量 3.8Kg 3.原理 见米纳斯驱动器方框图(图1)和控制方框图(图2) 4.接线 4.1主回路 卸下盖板坚固螺丝;取下端子盖板。用足够线经和连接器尺寸作连接,导线应采用额定温度600C以上的铜体线,装上端子盖板,拧紧盖板螺丝。螺丝拧紧力矩大于1.2Nm M4或2.0 Nm M5时才可能损坏端子,接地线径为2.0mm2 具体见接线图3 4.2 CN SIG 连接器[ 具体见接线图4 ●驱动器和电机之间的电缆长度最大20M ●这些线至少要离开主电路接线30cm,不要让这些线与电源进线走一线槽; 或让它们捆扎在一起 ●线经0.18mm2或以上屏蔽双绞线,有足够的耐弯曲力 ●屏蔽驱动器侧的屏蔽应连接到CN.SIG 连接器的20脚,电机侧应连接到J 脚 ●若电缆长于10M,则编码器电源线+5V、0V应接双线 4.3 CN I/F 连接 ●控制器等周边设备与驱动器之间距离最大为3M ●这些线至少和主电路接线相隔30cm ,不要让这些线与电源进线走同一线槽 或和它们捆扎在一起 ●COM+和COM-之间的控制电源(V DC)由用户供给
NCBI 资源介绍 本文目录: NCBI(美国国立生物技术信息中心) 简介 NCBI 站点地图 NCBI癌症基因组研究 NCBI-Coffee Break NCBI-基因和疾病 NCBI-UniGene Cluster of Orthologous Groups of proteins( COG) 介绍 Gene Expression Omnibus ( GEO) 介绍 LocusLink介绍 关于RefSeq: NCBI参考序列 NCBI(美国国立生物技术信息中心)简介 介绍 理解自然无声但精妙的关于生命细胞的语言是现代分子生物学的要求。经过只有四个字母来代表DNA化学亚基的字母表, 出现了生命过程的语法, 其最复杂形式就是人类。阐明和使用这些字母来组成新的”单词和短语”是分子生物学领域的中心焦点。数目巨大的分子数据和这些数据的隐秘而精细的模式使得计算机化的数据库和分析方法成为绝正确必须。挑战在于发现新的手段去处理这些数据的容量和复杂性, 而且为研究人员提供更好的便利来获得分析
和计算的工具, 以便推动对我们遗传之物和其在健康和疾病中角色的理解。 国立中心的建立 后来的参议员Claude Pepper意识到信息计算机化过程方法对指导生物医学研究的重要性, 发起了在1988年11月4日建立国立生物技术信息中心( NCBI) 的立法。NCBI是在NIH的国立医学图书馆( NLM) 的一个分支。NLM是因为它在创立和维护生物信息学数据库方面的经验被选择的, 而且这能够建立一个内部的关于计算分子生物学的研究计划。NCBI的任务是发展新的信息学技术来帮助对那些控制健康和疾病的基本分子和遗传过程的理解。它的使命包括四项任务: 建立关于分子生物学, 生物化学, 和遗传学知识的存储和分析的自动系统 实行关于用于分析生物学重要分子和复合物的结构和功能的基于计算机的信息处理的, 先进方法的研究 加速生物技术研究者和医药治疗人员对数据库和软件的使用。 全世界范围内的生物技术信息收集的合作努力。 NCBI经过下面的计划来实现它的四项目的:
万能驱动助理【装机必备的万能驱动】 使用教程详解 前言 鉴于电脑的普及,以及电脑日常经常性的会遇到一些因为驱动问题而造成的故障现象,为解决这一技术难题,特发表此文章以供大家参考。 如果自己或者朋友的电脑遇到的驱动的故障,可以参考本文进行修复。 万能驱动助理是由IT天空出品的一个集易用性、广泛性、准确性、可靠性、高效性于一身的硬件驱动智能安装工具。本文部分摘自天空论坛,只是为了解决平时电脑遇到的一些常见的驱动问题,仅为了大家交流及推广。 如有侵权,作者敬请告知。 一、万能驱动助理 简介:万能驱动助理(简称WanDrv)是IT天空出品的一款智能识别电脑硬件并自动安装驱动的工具。它拥有简约友好的用户界面,使用起来十分方便。追求“万能”是一种态度,表达我们想要将产品做的尽善尽美的理念,也希望大家能和我们共同努力而使之不断完善,让大家切身感受到它在驱动安装方面的“无所不能”。 硬件支持:搭载了IT天空开发团队精心整理制作的驱动包。这是到目前为止,我们针对当前主流硬件设备收集和整理得最为全面的驱动集合,支持市面上绝大多数主流硬件,兼容以往多数旧硬件。驱动包经过合理的整合与压缩工作,以尽可能小的体积支持尽可能多的硬件设备。
二、万能驱动助理下载 万能驱动助理至今为止共五个版本:WinXP,Win7系统32位、64位,Win8系统32位、64位 WinXP万能驱动下载百度云网盘地址 https://www.doczj.com/doc/4614792957.html,/s/1hqn7PjA Win7系统32位万能驱动下载百度云网盘地址 https://www.doczj.com/doc/4614792957.html,/s/1mgjvXAo Win7系统64位万能驱动下载百度云网盘地址 https://www.doczj.com/doc/4614792957.html,/s/1hquZzwg Win8系统32位万能驱动下载百度云网盘地址 https://www.doczj.com/doc/4614792957.html,/s/1qWAYfyW Win8系统64位万能驱动下载百度云网盘地址 https://www.doczj.com/doc/4614792957.html,/s/1pJO7uzT 所有驱动包下载百度云网盘地址 https://www.doczj.com/doc/4614792957.html,/s/1i3mPODR 打开网址链接之后,打开相应的文件夹,选择下载文件即可 注,此链接只要百度云网盘还可使用,则链接永久有效,并且驱动包一直更新为最新驱动包