
实验三:多条序列比对——Clustalx
(一)ClustalX
Clustal是一种利用渐近法(progressive alignment)进行多条序列比对的软件。即从多条序列中最相似(距离最近)的两条序列开始比对,按照各个序列在进化树上的位置,由近及远的将其它序列依次加入到最终的比对结果。(Figure 3.1)
https://www.doczj.com/doc/0711934624.html,/
1.安装clustalx程序。
双击安装clustalx-2.0.12-win.msi.exe文件到自己的电脑上。
也可从https://www.doczj.com/doc/0711934624.html,/download/current/下载,列表中的倒数第二个文件。clustalx-2.0.12-win.msi
Figure 3.1 clustal 算法
2.准备要比对的序列
请查找至少存在于5个物种中的同源序列(核酸或蛋白质皆可),并保存为fasta格式,存为文本文件(所有的序列请粘贴到同一个文本文件中)。选择NM、XM或NP打头的序列,不要选择NC或NW打头的序列,那是全基因组序列。
做法可参照邮箱中的preparations for practice3.doc文件。
3.打开clustalX程序
开始菜单-程序-clustalX2- clustalX2
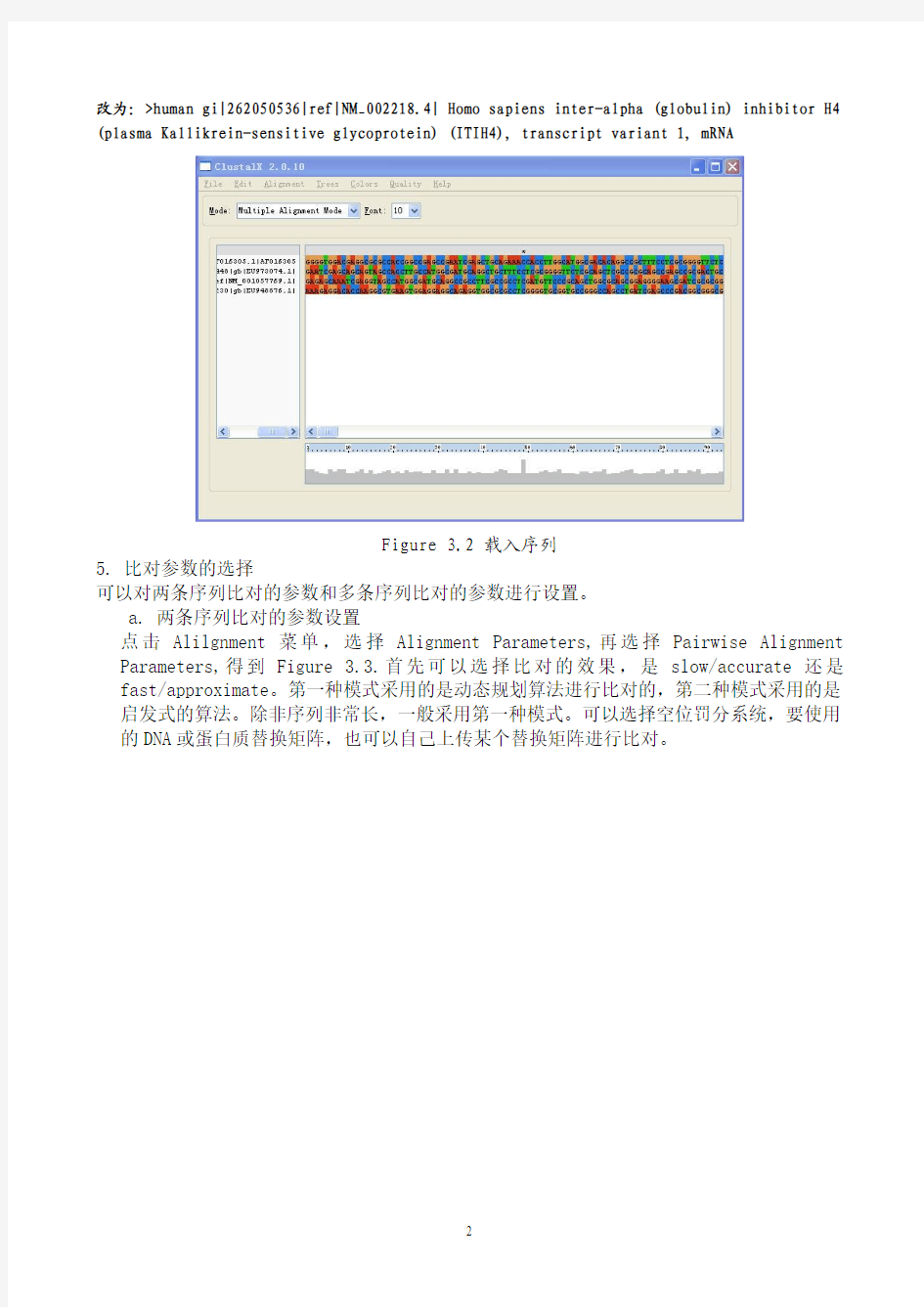
4.载入序列
点最上方的File菜单,选择Load Sequence-选择你刚保存的序列文件,点打开。
在左侧窗口里是fasta格式序列的标识号,取自序列第一行“>”后的字符。(Figure 3.2) 注意:ClustalX程序无法识别汉字,无法识别带空位的文件夹名,如 my document。各位同学保存的序列文件不要保存在桌面上或带汉字的文件夹中,推荐保存在D盘根目录下。
常见文件打开错误原因:
1.序列格式有问题,非正确的fasta格式。
2.文件中有序列重复粘贴。
TIPS: 想要方便识别序列所属物种,可在每条序列“>”后输入物种名,加空位即可。EXAMPLE:原格式:>gi|262050536|ref|NM_002218.4| Homo sapiens inter-alpha (globulin) inhibitor H4 (plasma Kallikrein-sensitive glycoprotein) (ITIH4), transcript variant 1, mRNA
改为:>human gi|262050536|ref|NM_002218.4| Homo sapiens inter-alpha (globulin) inhibitor H4 (plasma Kallikrein-sensitive glycoprotein) (ITIH4), transcript variant 1, mRNA
Figure 3.2 载入序列
5.比对参数的选择
可以对两条序列比对的参数和多条序列比对的参数进行设置。
a.两条序列比对的参数设置
点击Alilgnment菜单,选择Alignment Parameters,再选择Pairwise Alignment Parameters,得到Figure 3.3.首先可以选择比对的效果,是slow/accurate 还是fast/approximate。第一种模式采用的是动态规划算法进行比对的,第二种模式采用的是启发式的算法。除非序列非常长,一般采用第一种模式。可以选择空位罚分系统,要使用的DNA或蛋白质替换矩阵,也可以自己上传某个替换矩阵进行比对。
Figure 3.3 Pairwise Alignment Parameters
b.多条序列比对参数设置
点击Alilgnment菜单,选择Alignment Parameters,再选择Multiple Alignment Parameters,得到Figure 3.4.
Figure 3.4 Multiple Alignment Parameters
Delay divergent sequence是指当两条序列的差异大于某个值(百分比)的时候,这两条序列的比对将推迟进行,它们的比对结果会在最后加入到最终的多条序列比对结果。DNA
transition Weight等于0的时候,程序将转换(transition)当作错配(mismatch)看待,等于1的时候,将转换(transition)当作颠换(transversion)看待。当参与比对的序列差异较大时,DNA transition Weight应该选择的小些(接近0),如果参与比对的序列差异较小时,DNA transition Weight可选择的大些(接近1)。
6.更改输出格式
点击Alignment菜单,选择Output Format Options,页面如Figure 3.5 。
默认的是输出clustal format,如果需要其它格式,可在复选框里打勾。PHYLIP格式是利用PHYLIP软件进行建树时,需要输入的格式,我们将在实验6系统发育树构建中用到。
Figure 3.5 输出格式选项
7.进行比对
点击Aliglnment菜单,选择Do Complete Alignment.此时出现一个对话框,提示你比对结果保存的位置,你在上一步选择了多少种输出格式,这里就会给出多少个文件的路径。选择好了点OK即可。
要得到理想的比对结果,你可能需要选择不同的参数,进行多次比对,最后再对各种比对结果进行分析,选择哪个是最合理的结果(the result making biological sense)。
比对结束后生成的aln文件是多条序列比对的结果,可以用记事本打开浏览(Figure 3.6)。在某一列比对结果下方如果出现*,说明这列是完全匹配。dnd文件是比对过程中生成的进化树,可以用treeview(压缩包中的treev32.rar文件)打开浏览(Figure 3.7)。
Figure 3.6 记事本打开生成的aln文件
Figure 3.7 Treeview打开dnd文件
8.迭代比对
如果序列比对结果不理想,可以采用迭代选项,多次迭代寻找最佳比对结果。
点击Alignment菜单,选择iteration,选择iterate each alignment step或iterate final alignment.
然后再点击Aliglnment菜单,选择Do Complete Alignment进行比对。
9.概型(Profile)比对模式
以上介绍的都是Multiple alignment Mode,ClustalX还提供了一个概型比对模式,在菜单栏下方选择Profile Alignment Mode,可以对两个比对结果(alignment, termed profile here)进行再比对,或将一条序列与一个比对结果(profile)进行比对。(Figure 3.8)还
可以利用二级结构信息指导多条序列比对。
Figure 3.8 Profile Alignment Mode
(二)Treeview
Clustalx产生的guide tree(即后缀为dnd文件),可以通过treeview软件浏览。
解压缩并安装treev32.rar文件。双击后缀为dnd文件,选择treeview程序打开即可。
其它不详之处请参考clustalx_help.html或clustalx.pdf两个文件。
作业:
1.Clustalx是多条序列比对软件,为什么需要设置两条序列比对的参数?
2.利用entrez或srs搜索来自于不同物种的同源序列(othologs),利用clustalX进行比对,
给出所选序列简要信息(fasta格式第一行),比对所用的参数,比对过程中产生的guide
tree(dnd文件),并分析比对结果(序列之间相似度关系,保守位点所在位置等)。
实验三:多条序列比对——Clustalx (一)ClustalX Clustal是一种利用渐近法(progressive alignment)进行多条序列比对的软件。即从多条序列中最相似(距离最近)的两条序列开始比对,按照各个序列在进化树上的位置,由近及远的将其它序列依次加入到最终的比对结果。(Figure 3.1) https://www.doczj.com/doc/0711934624.html,/ 1.安装clustalx程序。 双击安装clustalx-2.0.12-win.msi.exe文件到自己的电脑上。 也可从https://www.doczj.com/doc/0711934624.html,/download/current/下载,列表中的倒数第二个文件。clustalx-2.0.12-win.msi Figure 3.1 clustal 算法 2.准备要比对的序列 请查找至少存在于5个物种中的同源序列(核酸或蛋白质皆可),并保存为fasta格式,存为文本文件(所有的序列请粘贴到同一个文本文件中)。选择NM、XM或NP打头的序列,不要选择NC或NW打头的序列,那是全基因组序列。 做法可参照邮箱中的preparations for practice3.doc文件。 3.打开clustalX程序 开始菜单-程序-clustalX2- clustalX2 4.载入序列 点最上方的File菜单,选择Load Sequence-选择你刚保存的序列文件,点打开。 在左侧窗口里是fasta格式序列的标识号,取自序列第一行“>”后的字符。(Figure 3.2) 注意:ClustalX程序无法识别汉字,无法识别带空位的文件夹名,如 my document。各位同学保存的序列文件不要保存在桌面上或带汉字的文件夹中,推荐保存在D盘根目录下。 常见文件打开错误原因: 1.序列格式有问题,非正确的fasta格式。 2.文件中有序列重复粘贴。 TIPS: 想要方便识别序列所属物种,可在每条序列“>”后输入物种名,加空位即可。EXAMPLE:原格式:>gi|262050536|ref|NM_002218.4| Homo sapiens inter-alpha (globulin) inhibitor H4 (plasma Kallikrein-sensitive glycoprotein) (ITIH4), transcript variant 1, mRNA
Clustalx 多重序列比对图解教程(By Raindy) 本帖首发于Raindy'blog,转载请保留作者信息,谢谢!欢迎有写生物学软件专长的战友,加入生信教程写作群:,接头暗号:你所擅长的生物学软件名称 软件简介: CLUSTALX-是CLUSTAL多重序列比对程序的Windows版本。Clustal X为进行多重序列和轮廓比对和分析结果提供一个整体的环境。 序列将显示屏幕的窗口中。采用多色彩的模式可以在比对中加亮保守区的特征。窗口上面的下拉菜单可让你选择传统多重比对和轮廓比对需要的所有选项。 主要功能: 你可以剪切、粘贴序列以更改比对的顺序; 你可以选择序列子集进行比对; 你可以选择比对的子排列(Sub-range)进行重新比对并可插入到原始比对中; 可执行比对质量分析,低分值片段或异常残基将以高亮显示。 当前版本:1.83 PS:如果你是新手或喜欢中文界面,推荐使用本人汉化的Clustalx 1.81版链接地址::ist&ID=7435(请完整复制) 应用:Clustalx比对结果是构建系统发育树的前提 实例:植物呼肠孤病毒属外层衣壳蛋白P8(AA序列)为例 流程:载入序列―>编辑序列―>设置参数―>完全比对―>比对结果 1.载入序列:运行ClustalX,主界面窗口如下所图(图1),依次在程序上方的菜单栏选择“File”-“Load Sequence”载入待比对的序列,如图2所示,如果当前已载入序列,此时会提示是否替换现有序列(Replace existing sequences),根据具体情形选择操作。
图1
图2 2.编辑序列:对标尺(Ruler)上方的序列进行编辑操作,主要有Cut sequences(剪切序列)、Paste sequences(粘贴)、Select All sequences(选定所有序列),Clear sequence Selection(清除序列选定)、Search for string(搜索字串)、Remove All gaps(移除序列空位)、Remove Gap-Only Columns(仅移除选定序列的空位)
利用clustalx 2.1对蛋白进行多序列比对目录 1. 方法介绍 1.1概念 1.2理论基础 1.3任务 1.4目的 2研究内容 3. 工具 3.1 clustalx简介 3.2 clustalx 后台运作流程 3.3 clustalx的下载 3.4 clustalx菜单设置 4.操作步骤 4.1获取目标序列 4.2执行比对 4.3 treeview软件制作进化树 5. 结果分析 正文
1. 方法介绍:多序列比对 1.1 概念:多序列比对即通过多个核苷酸或氨基酸的序列进行比较,确定 序列之间可能由于功能、结构或进化上的关联而形成的相似片 段。 1.2 理论基础:1)生物学一个最基本的假设是地球上所有物种都有共同的 祖先,从这个祖先开始以树状形式发展,通常称为生命 之树。 2)基于序列比对的同源即具有共同祖先。同源序列一般相 似;相似可以用百分比来描述。序列不一定是同源的, 相似序列在进化上具有趋同性。序列决定结构,结构决 定功能。 3)现有的基因、蛋白质等携带生物学信息、具有生物学功 能的分子都是由原有的分子演化而来;现有的基因及其 他核酸序列,都是由已经存在的基因或其他序列经过复 制、转移、合并、删减等方式形成的;不同物种的基因、 蛋白质在结构、序列上的相似性与其进化上亲缘关系密 切相关。 1.3 任务:发现序列之间的相似性,找出序列之间共同的区域,辨别序列 之间的差异。 1.4 目的:通过“相似序列→相似的结构→相似的功能“来判别序列之 间的同源性,进而推测序列之间的进化关系。 2. 研究内容:通过对人类、家鼠、大鼠和鸡体内BMP-2(bone morphogenetic protein 2)即骨形态发生蛋白2的多序列比对得到的dnd结果文 件来揭示在四种生物中的该蛋白的同源性。 3. 工具:clustalx 2.1 3.1 clustalx简介:Clustal是用来对核酸与蛋白序列进行多序列比对的软 件,可以用来发现特征序列,进行蛋白分类,证明序列 间的同源性,帮助预测新序列二级结构与三级结构,确 定PCR引物,以及在分子进化分析方面均有很大帮助。 Clustal包括Clustalw和Clustalx和Clustal omega。
序列比对之Clustalx与Clustalw使用指南 这几天实验需要做多序列比对,很久不做了,一时之间不知道如何使用clustal这个工具了。在网上搜集了一些资料,做个整理,总结了Clustalx和Clustalw的使用,省得以后久不使用又生疏了,又要去整理了,在此分享给大家,希望有所帮助。 1.先提供下载地址: 官方下载地址:https://www.doczj.com/doc/0711934624.html,/download/current/ Clustalx、Clustalw的各种最新版本都能下载到,包括linux、Win、Mac... 2.原理:序列同源性分析: 是将待研究序列加入到一组与之同源,但来自不同物种的序列中进行多序列同时比较,以确定该序列与其它序列间的同源性大小。这是理论分析方法中最关键的一步。完成这一工作必须使用多序列比较算法。常用的程序包有CLUSTAL等; Clustal是一个单机版的基于渐进比对的多序列比对工具,由Higgins D.G.等开发。有应用于多种操作系统平台的版本,包括linux版,DOS版的clustlw,clustalx等。 CLUSTAL是一种渐进的比对方法,先将多个序列两两比对构建距离矩阵,反应序列之间两两关系;然后根据距离矩阵计算产生系统进化指导树,对关系密切的序列进行加权;然后从最紧密的两条序列开始,逐步引入临近的序列并不断重新构建比对,直到所有序列都被加入为止。 3.操作 3.1 Clustalx的操作 第一步:输入序列文件。
第二步:设定比对的一些参数。 参数设定窗口。
第三步:开始序列比对。
第四步:比对完成,选择保存结果文件的格式 --------------------------------------------
多重序列比对及系统发生树的构建 作者:佚名来源:生物秀时间:2007-12-31 【实验目的】 1、熟悉构建分子系统发生树的基本过程,获得使用不同建树方法、建树材料和建树参数对建树结果影响的正确认识; 2、掌握使用Clustalx进行序列多重比对的操作方法; 3、掌握使用Phylip软件构建系统发生树的操作方法。 【实验原理】 在现代分子进化研究中,根据现有生物基因或物种多样性来重建生物的进化史是一个非常重要的问题。一个可靠的系统发生的推断,将揭示出有关生物进化过程的顺序,有助于我们了解生物进化的历史和进化机制。 对于一个完整的进化树分析需要以下几个步骤:⑴要对所分析的多序列目标进行比对(alignment)。⑵要构建一个进化树(phyligenetic tree)。构建进化树的算法主要分为两类:独立元素法(discrete character methods)和距离依靠法(distance methods)。所谓独立元素法是指进化树的拓扑形状是由序列上的每个碱基/氨基酸的状态决定的(例如:一个序列上可能包含很多的酶切位点,而每个酶切位点的存在与否是由几个碱基的状态决定的,也就是说一个序列碱基的状态决定着它的酶切位点状态,当多个序列进行进化树分析时,进化树的拓扑形状也就由这些碱基的状态决定了)。而距离依靠法是指进化树的拓扑形状由两两序列的进化距离决定的。进化树枝条的长度代表着进化距离。独立元素法包括最大简约性法(Maximum Parsimony methods)和最大可能性法(Maximum Likelihood methods);距离依靠法包括除权配对法(UPGMAM)和邻位相连法(Neighbor-joining)。⑶对进化树进行评估,主要采用Bootstraping法。进化树的构建是一个统计学问题,我们所构建出来的进化树只是对真实的进化关系的评估或者模拟。如果我们采用了一个适当的方法,那么所构建的进化树就会接近真实的“进化树”。模拟的进化树需要一种数学方法来对其进行评估。不同的算法有不同的适用目标。一般来说,最大简约性法适用于符合以下条件的多序列:i 所要比较的序列的碱基差别小,ii 对于序列上的每一个碱基有近似相等的变异率,iii 没有过多的颠换/转换的倾向,iv 所检验的序列的碱基数目较多(大于几千个碱基);
如何用MEGA5.0和Clustalx1.83构建进化树 MEGA是一个关于序列分析以及比较统计的工具包,从3.1版本到后来的4.0版本一直都广为大家熟悉,现在推出了Mega5.0版本。功能比以前多有改进。现主要介绍使用Mega 5.0构建系统进化树的方法。供大家参考。 用MEGA构建进化树有以下步骤: 1、测序: 将克隆扩增测序得到的16S rDNA序列进行测序。 2、NCBI上做Blast https://www.doczj.com/doc/0711934624.html,/blast/Blast.cgi 找到相似度最高的几个序列,确定一下你分离的细菌大约属于哪个科哪个属,如果相似度达到百分之百那基本可以确定你分离得到的就是Blast到的那个,然后寻找相似性最高的细菌,通常把该属的序列(Fasta格式文件)下载下来,或点击GenBank登录号,复制FSA TA 格式,整合在一个*.txt文档中(单独建立一个文件夹存放,后面的很多文件会自动装入该文件夹),如 >XXXX AGGCTTAACACA TGCAAGTCGAGCGGAGCGAGGGTGCTTGCACCTTAGCTTAGCGGCG GACGGGTGAGTAA TGCTTAGGAA TCTGCCTA TTAGTGGGGGACAACA TTCCGAAAGGA A TGCTAA TACCGCA TACGCCCTACGGGGGAAAGCAGGGGA TCTTCGGACCTTGCGCTAA TAGA TGAGCCTAAGTCGGA TTAGCTAGTTGGTGGG >gi|289469964|gb|GU388381.1| Acinetobacter tandoii strain DSM 14970 16S ribosomal RNA gene, partial sequence ACTTAGCGGCGGACGGGTGAGTAA TGCTTAGGAA TCTGCCTA TTAGTGGGGGACAACA TTCCGAAAGGGA TGCTAA TACCGCA TACGCCCTACGGGGGAAAGCAGGGGA TCTTCGG ACCTTGCGCTAA TAGA TGAGCCTAAGTCGGA TTAGCTAGTTGGTGGGGTAAAGGCCTAC CAAGGCGACGA TCTGTAGCGGGTCTGAGAGGA TGA…… ……………………. 参考序列选择注意事项: 1、不选非培养(unclutured)微生物为参比; 2、不选未定分类地位的微生物,最相近的仅作参考;c,在保证同属的前提下,优先选择16S rDNA全长测序或全基因组测序的种;d,每个种属选择一个参考序列,如果自己的序列中同一属的较多,可适当选择两个参考序列。 3、使用clustalx1.83进行序列比对 打开压缩文件clustalx1.83,运行其中的clustalx.exe文件,如图:
在寻找基因和致力于发现新蛋白的努力中,人们习惯于把新的序列同已知功能的蛋白序列作比对。由于这些比对通常都希望能够推测新蛋白的功能,不管它们是双重比对还是多序列比对,都可以回答大量的其它的生物学问题。举例来说,面对一堆搜集的比对序列,人们会研究隐含于蛋白之中的系统发生的关系,以便于更好地理解蛋白的进化。人们并不只是着眼于某一个蛋白,而是研究一个家族中的相关蛋白,看看进化压力和生物秩序如何结合起来创造出新的具有虽然不同但是功能相关的蛋白。研究完多序列比对中的高度保守区域,我们可以对蛋白质的整个结构进行预测,并且猜测这些保守区域对于维持三维结构的重要性。 显然,分析一群相关蛋白质时,很有必要了解比对的正确构成。发展用于多序列比对的程序是一个很有活力的研究领域,绝大多数方法都是基于渐进比对(progressive alignment)的概念。渐进比对的思想依赖于使用者用作比对的蛋白质序列之间确实存在的生物学上的或者更准确地说是系统发生学上的相互关联。不同算法从不同方面解决这一问题,但是当比对的序列大大地超过两个时(双重比对),对于计算的挑战就会很令人生畏。在实际操作中,算法会在计算速度和获得最佳比对之间寻求平衡,常常会接受足够相近的比对。不管最终使用的是什么方法,使用者都必须审视结果的比对,因为再次基础上作一些手工修改是十分必要的,尤其是对保守的区域。 由于本书偏重于方法而不是原理,这里只讨论一小部分现成的程序。我们从两个多序列比对的方法开始,接下去是一系列的利用蛋白质家族中已知的模体或是式样的方法,最后讨论两个具有赠送的方法,因为绝大多数公开的算法不能达到出版物的数量。在本章结尾部分将会列出更详细的多序列比对的算法。 渐进比对方法 CLUSTAL W CLUSTAL W算法是一个最广泛使用的多序列比对程序,在任何主要的计算机平台上都可以免费使用。这个程序基于渐进比对的思想,得到一系列序列的输入,对于每两个序列进行双重比对并且计算结果。基于这些比较,计算得到一个距离矩阵,反映了每对序列 Bioinformatics: A Practical Guide to the Analysis of genes and Proteins Edited by A.D. Baxevanis and B.E.E. Ouellette ISBN 0-471-191965. pages 172-188. Copyright ? 1998 Wiley – Liss. Inc.
C l u s t a l x多重序列比对图解教程(B y R a i n d y) 本帖首发于Raindy'blog 软件简介: CLUSTALX-是CLUSTAL多重序列比对程序的Windows版本。ClustalX为进行多重序列和轮廓比对和分析结果提供一个整体的环境。 序列将显示屏幕的窗口中。采用多色彩的模式可以在比对中加亮保守区的特征。窗口上面的下拉菜单可让你选择传统多重比对和轮廓比对需要的所有选项。 主要功能: 你可以剪切、粘贴序列以更改比对的顺序; 你可以选择序列子集进行比对; 你可以选择比对的子排列(Sub-range)进行重新比对并可插入到原始比对中; 可执行比对质量分析,低分值片段或异常残基将以高亮显示。 当前版本:1.83 PS:如果你是新手或喜欢中文界面,推荐使用本人汉化的Clustalx1.81版 链接地址:ist&ID=7435(请完整复制) 应用:Clustalx比对结果是构建系统发育树的前提 实例:植物呼肠孤病毒属外层衣壳蛋白P8(AA序列)为例 流程:载入序列―>编辑序列―>设置参数―>完全比对―>比对结果 1.载入序列:运行ClustalX,主界面窗口如下所图(图1),依次在程序上方的菜单栏选择“File”-“LoadSequence”载入待比对的序列,如图2所示,如果当前已载入序列,此时会提示是否替换现有序列(Replaceexistingsequences),根据具体情形选择操作。
图1
图2 2.编辑序列:对标尺(Ruler)上方的序列进行编辑操作,主要有Cutsequences(剪切序列)、Pastesequences(粘贴)、SelectAllsequences(选定所有序列),ClearsequenceSelection(清除序列选定)、Searchforstring(搜索字串)、RemoveAllgaps(移除序列空位)、 RemoveGap-OnlyColumns(仅移除选定序列的空位)
ClustalX Help 可以在下列地址得到 ClustalX 的最新版本: ftp://ftp-igbmc.u-strasbg.fr/pub/ClustalX/ General help for CLUSTAL X (1.8) Clustal X 是一个windows 界面多序列对比程序。可以用剪切和粘贴的方法改变对比的顺序;可以在比对中选择较小的区域重新比对,并将比对的结果插回到原来的比对结果中。能够进行比对质量评定,低分片断和多余的残基将突出显示。 序列输入 序列和轮廓(已经存在的比对)利用菜单文件输入,所有的序列必须放到一个文件中,7种序列可以被自动识别: NBRF/PIR, EMBL/SWISSPROT, Pearson (Fasta), Clustal (*.aln), GCG/MSF (Pileup), 除用于表示间隙的"-" 例外 ("." in MSF/RSF),所有的非字母字符将被忽略。 序列和轮廓比对 Clustal X 有两种比对格式: 多重比对格式和轮廓比对格式。 做一系列序列的多重比对时要保证选择多重比对模式,然后展示单一序列数据。比对菜单既可以产生比对的指导树又可根据指导树进行比对,还可以做全多重比对。 在轮廓比对模式下,出现两个序列数据区,允许对两个比对结果进行比对。轮廓允许添加新序列到旧的比对中,或者应用二级结构指导比对进程。旧比对中的间隙用 "-"表示。轮廓可以用以下任何一种格式输入,只有用 "-" (or "." for MSF/RSF) 代表每一个间隙位置。在轮廓比对状态下, "Lock Scroll"按钮 is displayed which allows you to scroll the two profiles together using a single scroll bar. When the Lock Scroll is turned off, the two profiles can be scrolled independently. 进化树 进化树可以从旧的比对或新比对中产生。 比对显示 比对展示在屏幕上,序列的名称在左侧。除了通过剪切和粘贴改变序列的顺序外,不能进行其他的编辑。 序列的下方有一标尺,从1开始表示第一个残基的位置。比对的上面一行用来标记保守残基的位置。分别使用三个字母'*', ':' and '.' 表示。 '*' 表示单一的完全保守残基。 ':' 表示以下'强' 群是完全保守的:- STA ; NEQK ; NHQK ;NDEQ ; QHRK ;MILV ; MILF ;HY ;FYW '.' 表示以下 '弱' 群是完全保守的:- CSA ; ATV ;SAG; STNK ; STPA ;SGND ; SNDEQK ;NDEQHK ;NEQHRK ; FVLIM ; HFY 这些都是Gonnet Pam250 matrix中的得分群,强群的得分 >0.5 ,弱群的得分=<0.5。 对于轮廓比对,在输入文件中的轮廓比对和空隙罚分展示在序列的上方。
Using ClustalX for multiple sequence alignment Jarno Tuimala December 2004
All rights reserved. The PDF version of this leaflet or parts of it can be used in Finnish universities as course material, provided that this copyright notice is included. However, this publication may not be sold or included as part of other publications without permission of the publisher.
Index Index (3) Quick Start (4) 1. Open ClustalX (4) 2. Read in the FastA-formatted sequences (4) 3. Modify the output format option, if necessary (5) 4. Create an alignment (5) Creating the input file for multiple sequence alignment (6) Multiple alignment theory (7) Getting the data into ClustalX (8) Setting up the alignment parameters (9) Pairwise alignment parameters (9) Multiple alignment parameters (11) Alignment output-format (12) Creating the alignment (13) Writing alignment as Postscript (14) Assessing the quality of the alignment (15) Advanced alignment strategies (16) Advanced options (17) Do alignment from the guide tree (17) Profile alignment (17) Using secondary structure information in the profile alignment (19)
生物学利用MEGA5.0和Clustalx1.83软件构建进化 树 MEGA是一个关于序列分析以及比较统计的工具包,从3.1版本到后来的4.0版本一直都广为大家熟悉,现在推出了Mega5.0版本。功能比以前多有改进。现主要介绍使用Mega 5.0构建系统进化树的方法。供大家参考。 用MEGA构建进化树有以下步骤: 1、测序: 将克隆扩增测序得到的16S rDNA序列进行测序。 2、NCBI上做Blast https://www.doczj.com/doc/0711934624.html,/blast/Blast.cgi 找到相似度最高的几个序列,确定一下你分离的细菌大约属于哪个科哪个属,如果相似度达到百分之百那基本可以确定你分离得到的就是Blast到的那个,然后寻找相似性最高的细菌,通常把该属的序列(Fasta格式文件)下载下来,或点击GenBank登录号,复制FSATA 格式,整合在一个*.txt文档中(单独建立一个文件夹存放,后面的很多文件会自动装入该文件夹),如 >XXXX AGGCTTAACACA TGCAAGTCGAGCGGAGCGAGGGTGCTTGCACCTTAGCTTAGCGGCG GACGGGTGAGTAA TGCTTAGGAA TCTGCCTA TTAGTGGGGGACAACATTCCGAAAGGA ATGCTAATACCGCATACGCCCTACGGGGGAAAGCAGGGGA TCTTCGGACCTTGCGCTAA TAGATGAGCCTAAGTCGGA TTAGCTAGTTGGTGGG >gi|289469964|gb|GU388381.1| Acinetobacter tandoii strain DSM 14970 16S ribosomal RNA gene, partial sequence ACTTAGCGGCGGACGGGTGAGTAATGCTTAGGAATCTGCCTATTAGTGGGGGACAACA TTCCGAAAGGGATGCTAATACCGCA TACGCCCTACGGGGGAAAGCAGGGGATCTTCGG ACCTTGCGCTAATAGATGAGCCTAAGTCGGATTAGCTAGTTGGTGGGGTAAAGGCCTAC CAAGGCGACGA TCTGTAGCGGGTCTGAGAGGATGA…… ……………………. 参考序列选择注意事项: 1、不选非培养(unclutured)微生物为参比; 2、不选未定分类地位的微生物,最相近的仅作参考;c,在保证同属的前提下,优先选择16S rDNA全长测序或全基因组测序的种;d,每个种属选择一个参考序列,如果自己的序列中同一属的较多,可适当选择两个参考序列。 3、使用clustalx1.83进行序列比对 打开压缩文件clustalx1.83,运行其中的clustalx.exe文件,如图:
Clustal的使用 1.Clustalx 2.Clustalw 序列同源性分析: 是将待研究序列加入到一组与之同源,但来自不同物种的序列中进行多序列同时比较,以确定该序列与其它序列间的同源性大小。这是理论分析方法中最关键的一步。完成这一工作必须使用多序列比较算法。常用的程序包有CLUSTAL等; Clustal是一个单机版的基于渐进比对的多序列比对工具,由Higgins D.G. 等开发。有应用于多种操作系统平台的版本,包括linux 版,DOS版的clustlw,clustalx等。 CLUSTAL是一种渐进的比对方法,先将多个序列两两比对构建距离矩阵,反应序列之间两两关系;然后根据距离矩阵计算产生系统进化指导树,对关系密切的序列进行加权;然后从最紧密的两条序列开始,逐步引入临近的序列并不断重新构建比对,直到所有序列都被加入为止。 Clustalx的工作界面(多序列比对模式) Clustalx的工作界面(剖面(profile)比对模式)
Clustal的工作原理 Clustal输入多个序列>>>快速的序列两两比对,计算序列间的距离,获得一个距离矩阵。>>>邻接法(NJ)构建一个树(引导树)>>>根据引导树,渐进比对多个序列。 Clustal的应用 1.输入输出格式。 输入序列的格式比较灵活,可以是前面介绍过的FASTA格式,还可以是PIR、SWISS-PROT、GDE、Clustal、GCG/MSF、RSF 等格式。 输出格式也可以选择,有ALN、GCG、PHYLIP和NEXUS等,用户可以根据自己的需要选择合适的输出格式。 2.两种工作模式。 a.多序列比对模式。 b.剖面(profile)比对模式。 3.一个实际的例子。 输入文件的格式(fasta):